Подготовка чтений программой trimmomatic
Чтобы триммировать риды из файла SRR4240380.fastq.gz нужно подготовить файл с адаптерами. Файл с адаптерами получен, путем соединения 3 предложенных файлов с помощью bash
cat /mnt/scratch/NGS/adapters/* > adapt.fasta
После этого первая операция триммирования
TrimmomaticSE SRR4240380.fastq.gz seqout ILLUMINACLIP:adapt.fasta:2:7:7
В выдаче программы также содержалось:
Input Reads: 5217318 Surviving: 5119144 (98.12%) Dropped: 98174 (1.88%)
Из чего можем сделать вывод, что 1,88% ридов были остатками адаптеров. Триммируем дальше, обрежем некачественные и уберем короткие риды
TrimmomaticSE seqout seqout2.fastq TRAILING:20 MINLEN:32
Input Reads: 5119144 Surviving: 4865359 (95.04%) Dropped: 253785 (4.96%)
Таким образом, удалено было еще 253785 (4.96%) ридов. Вес исходного файла был 0,503Gb, а у полученного 0.478Gb.
K-меры и сборка
Из ридов получили 31-меры
velveth kdir 31 -short -fastq seqout2.fastq
После чего получили из них контиги
velvetg kdir/
В выдаче содержалось сообщение, откуда узнаем, что N50 = 12042:
Final graph has 401 nodes and n50 of 12042, max 25915, total 660284, using 0/4865359 reads
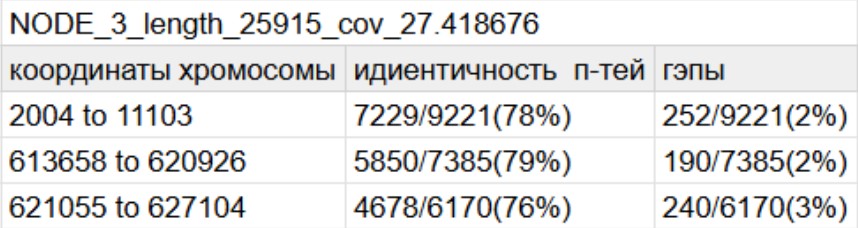
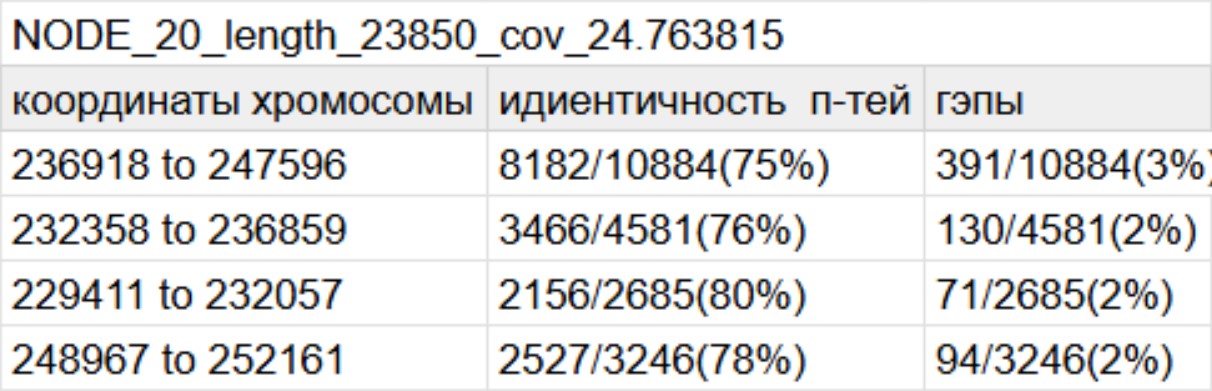
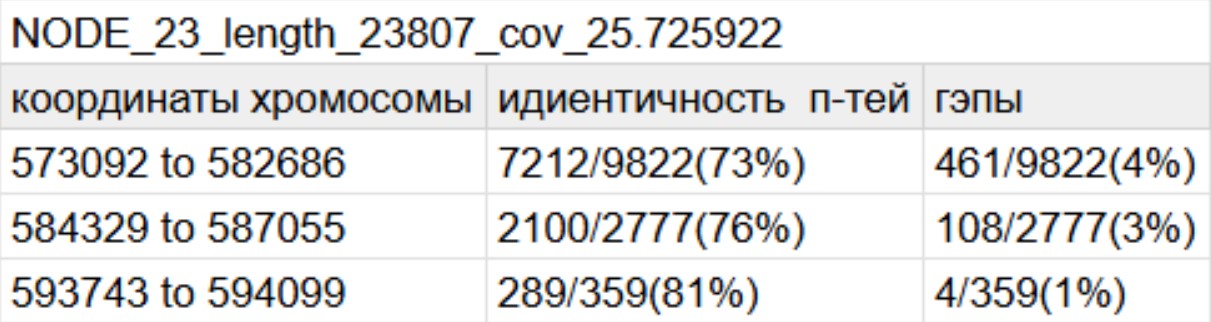
Получим длины самых больших контигов и их покрыте:
grep '>' kdir/contigs.fa | tr '_' '\t' | cut -f4,6 | sort -nr | head -n 3
25915 27.418676
23850 24.763815
23807 25.725922
Чтобы сравнивать с медианным покрытием надо найти медиану, тогда надо найти общее количество
grep '>' kdir/contigs.fa | wc -l
165
Тогда медианное значение - значение посередине отсортированного списка
kdir/contigs.fa | tr '_' '\t' | cut -f4 | sort -nr | head -n 83 | tail -n 1
22.568171
kdir/contigs.fa | tr '_' '\t' | cut -f4 | sort -nr | head -n 1
130.479660
kdir/contigs.fa | tr '_' '\t' | cut -f4 | sort -nr | tail -n 1
2.419355
Видим, что наибольший отличается в 5,8 раз, а наименьший в 9,3.
Наименьший можно привести полностью, хотя при этом он не самый короткий
>NODE_235_length_62_cov_2.419355
CTATGGTAGATTAATATAATCAATTAATACAAAATCCTAACCTAACCTAACCGTACTAAA
ATCAGATGAATAAACGCAAACGCATACAAAAC
С наибольшем же покрытием не самый длинный
>NODE_56_length_934_cov_130.479660