Практикум 2. Исследование форм ДНК (A, B, Z)
Задание 1
В ходе выполнения задания был использован пакет 3DNA, установленный на сервере kodomo. Использованная последовательность для A и B форм: AGTCAGTCAGTCAGTCAGTC, для Z-формы: GCGCGCGCGCGCGCGCGCGC.
Синтаксис: fiber -seq=(...) -(form) (filename).pdb
PDB со структурой дуплекса ДНК в A-форме.
PDB со структурой дуплекса ДНК в B-форме.
PDB со структурой дуплекса ДНК в Z-форме.
Задание 2
В заданиях 2 и 3 анализировались экспериментальные структуры ДНК из PDB: 1bna (B-форма) [1]; 1tne (Z-форма) [2]; 3v9d (A-форма) [3]. Визуально были найдены большая и малая бороздка двойной спирали ДНК в B-форме (1bna). Выбрано основание тимин на позиции 20:B, показано, в какую сторону (большой или малой бороздки) обращены его атомы. В сторону большой бороздки обращены атомы 20:B.C4 20:B.C5 20:B.C6 20:B.C7 20:B.O4. В сторону малой атомы 20:B.N1, 20:B.C2, 20:B.O2, 20:B.N3.
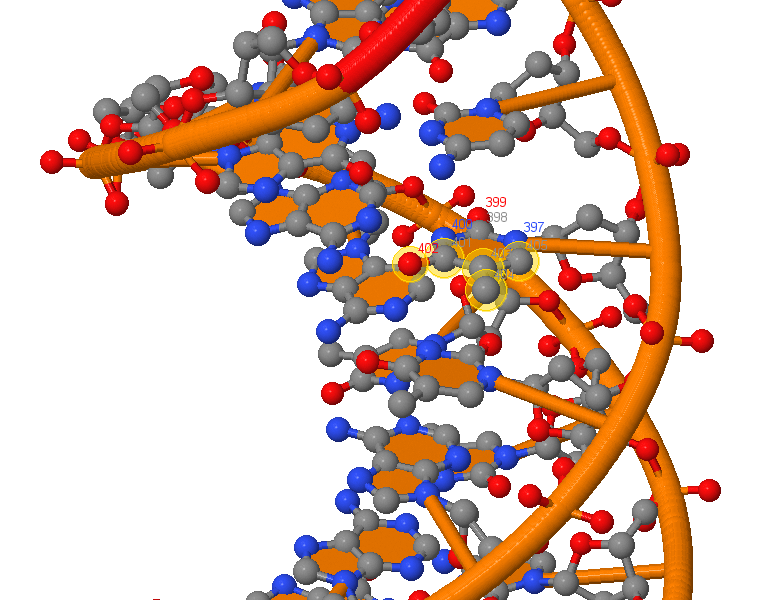
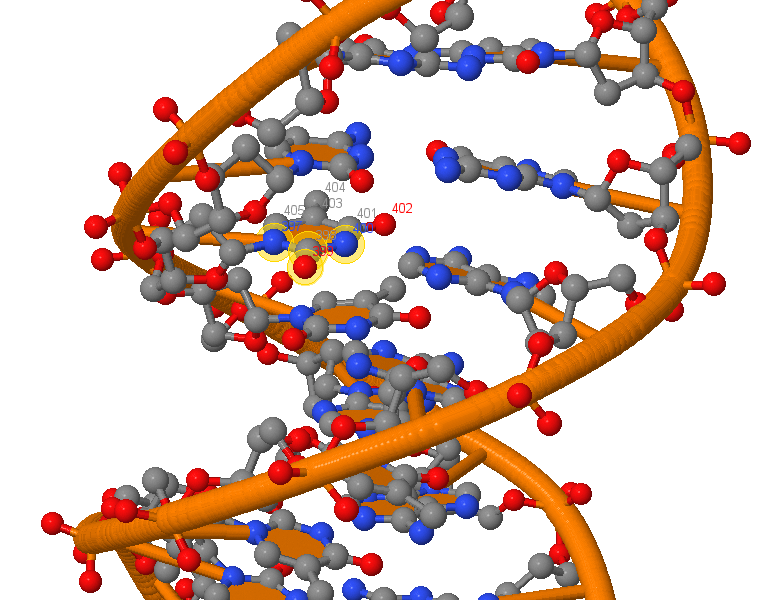
Затем были проанализированы для сравнения между собой все формы ДНК с использованием структур, сгенерированных 3DNA (так как они типовые и идеализированные, отображают типичное строение этой формы ДНК). Данные внесены в таблицу 1.
| Форма ДНК | A | B | Z |
| Тип спирали | Правая | Правая | Левая |
| Шаг спирали (ангстрем) | 28.03 | 33.75 | 43.50 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки | 16.81 (4:A-39:B) | 17.21 (4:A-34:B) | 16.08 (12:A-28:B) |
| Ширина малой бороздки | 7.98 (12:A-23:B) | 11.69 (12:A-33:B) | 7.2 (11:A-33:B) |
Задание 3.1
Торсионные углы нуклеотидов для тРНК и ДНК взяты медианные. Любопытно, что они отличаются от представленных в лекции.
| tRNA | A | B | Z | |
| alpha | -60.25 | -51.7 | -29.9 | -139.5 |
| beta | 165.2 | 174.8 | 136.3 | 21.15 |
| gamma | 55.2 | 41.7 | 31.1 | -61.5 |
| delta | 80.35 | 79.1 | 143.3 | 116.25 |
| epsilon | -146.7 | -147.8 | -140.8 | -103.6 |
| zeta | -73.9 | -75.1 | -160.5 | -64.8 |
| chi | -164.6 | -157.2 | -98 | -47.8 |
Задание 3.2
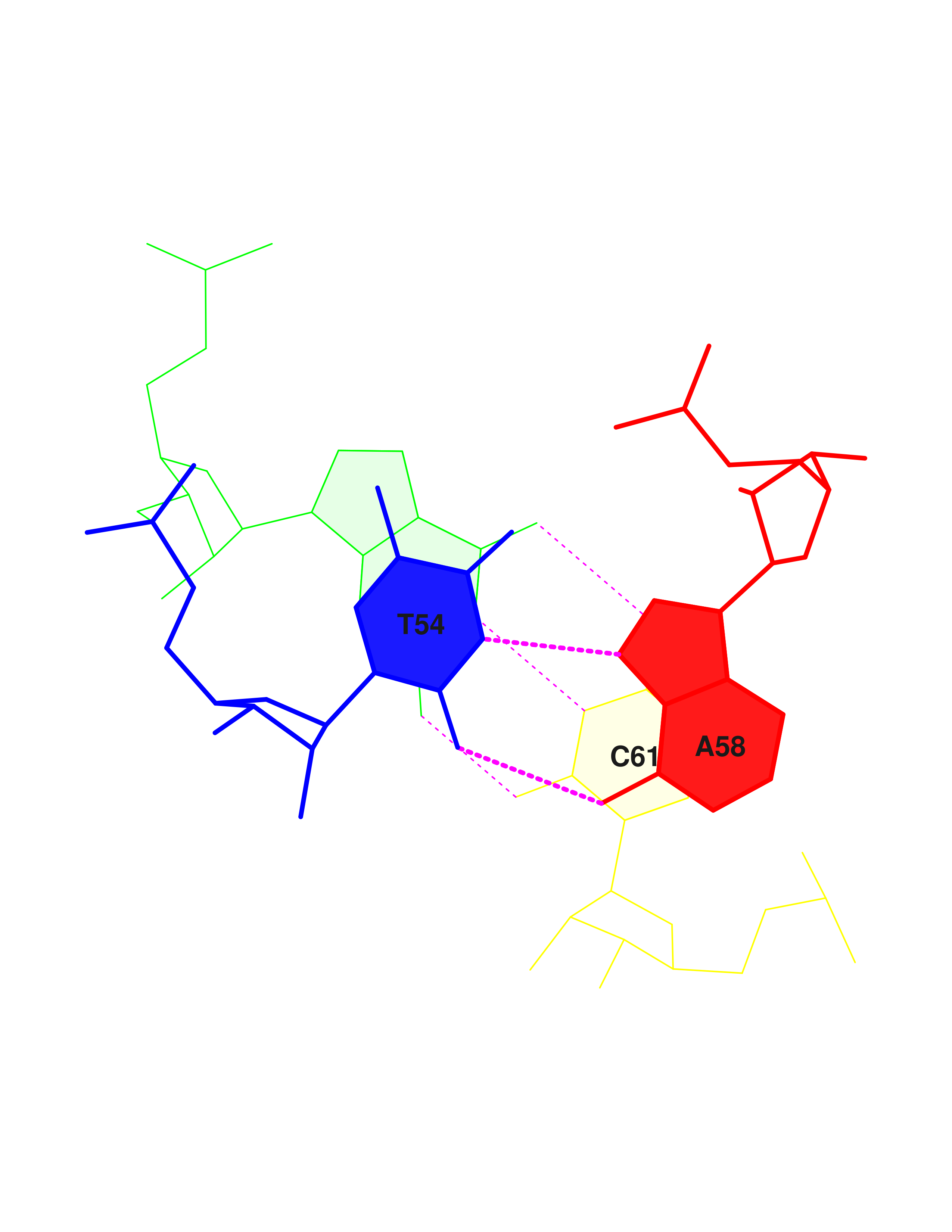
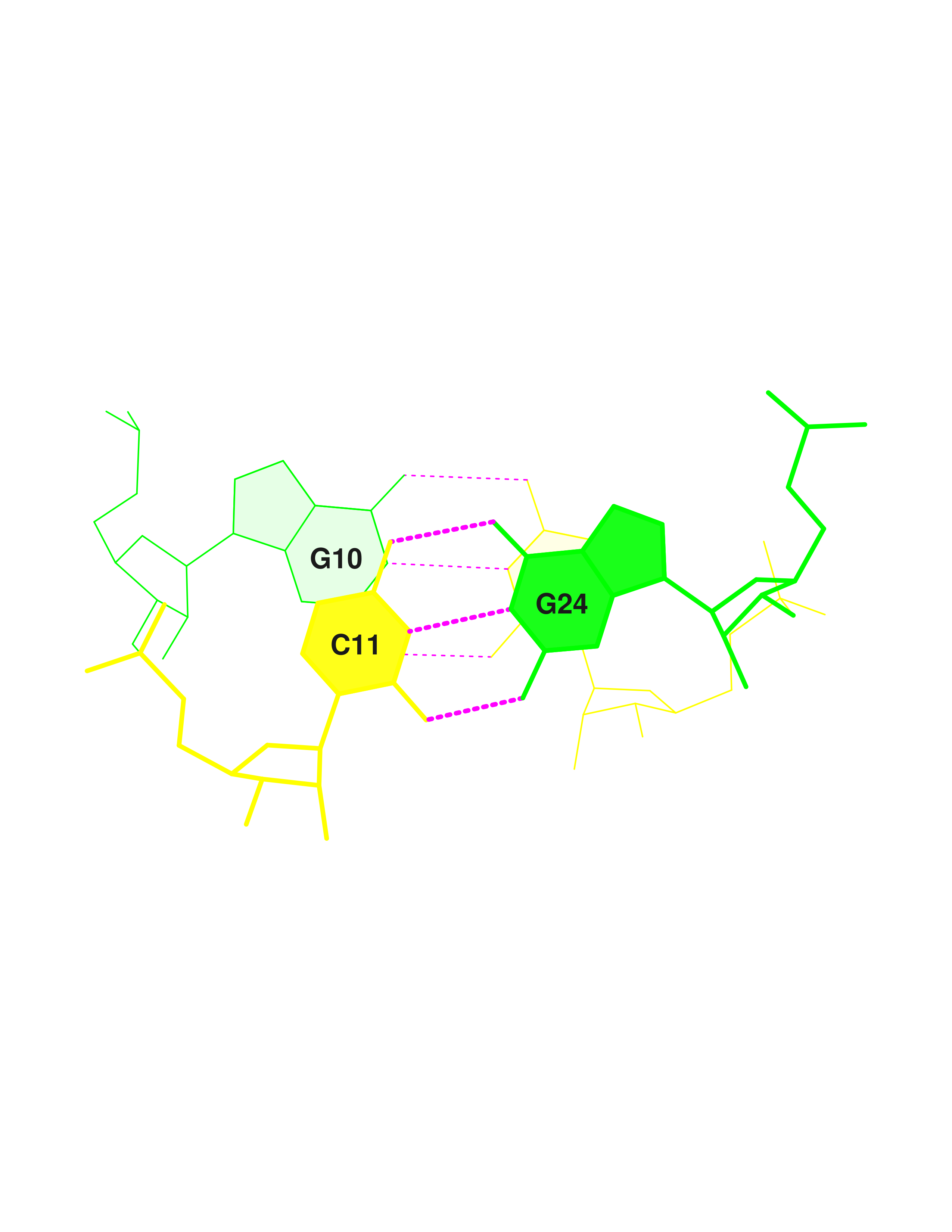
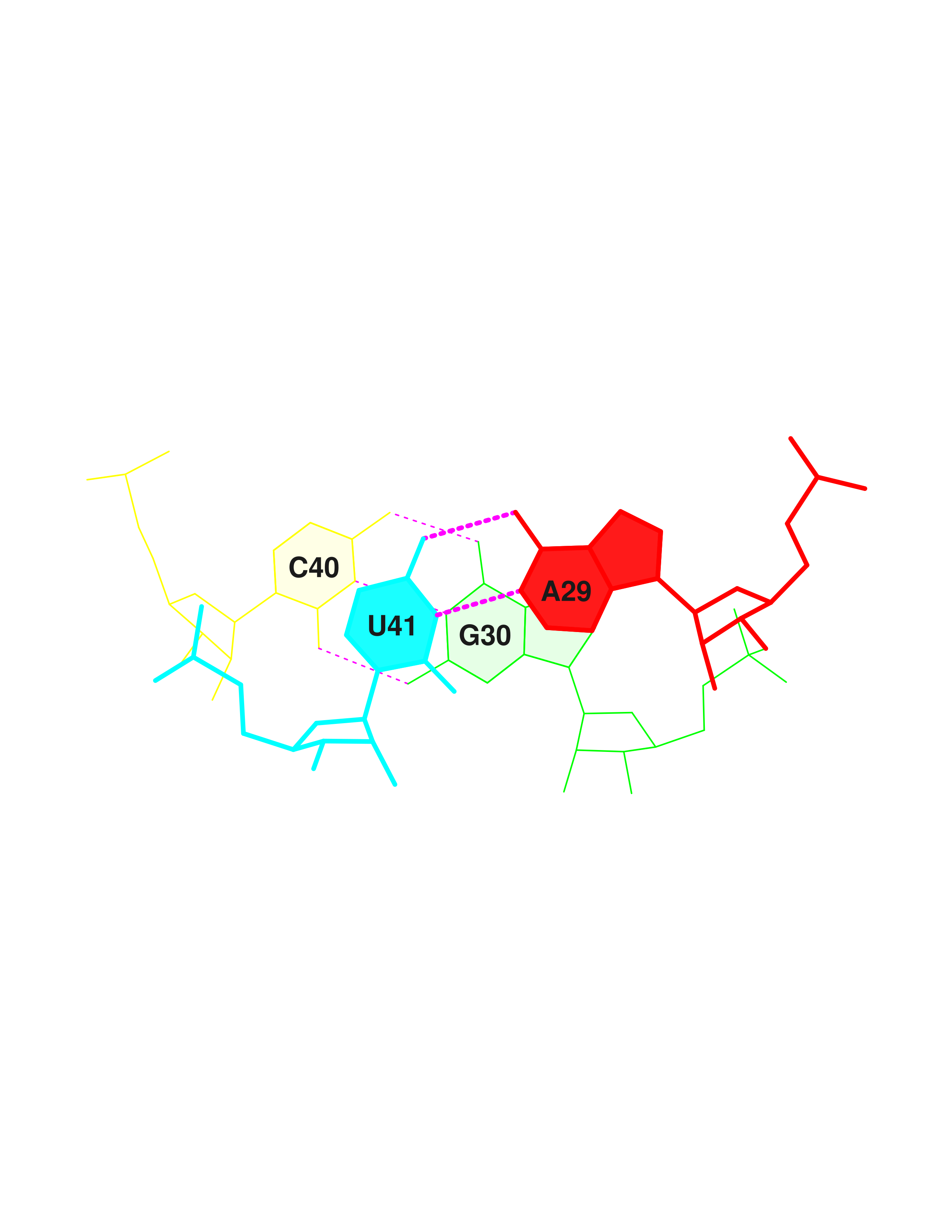
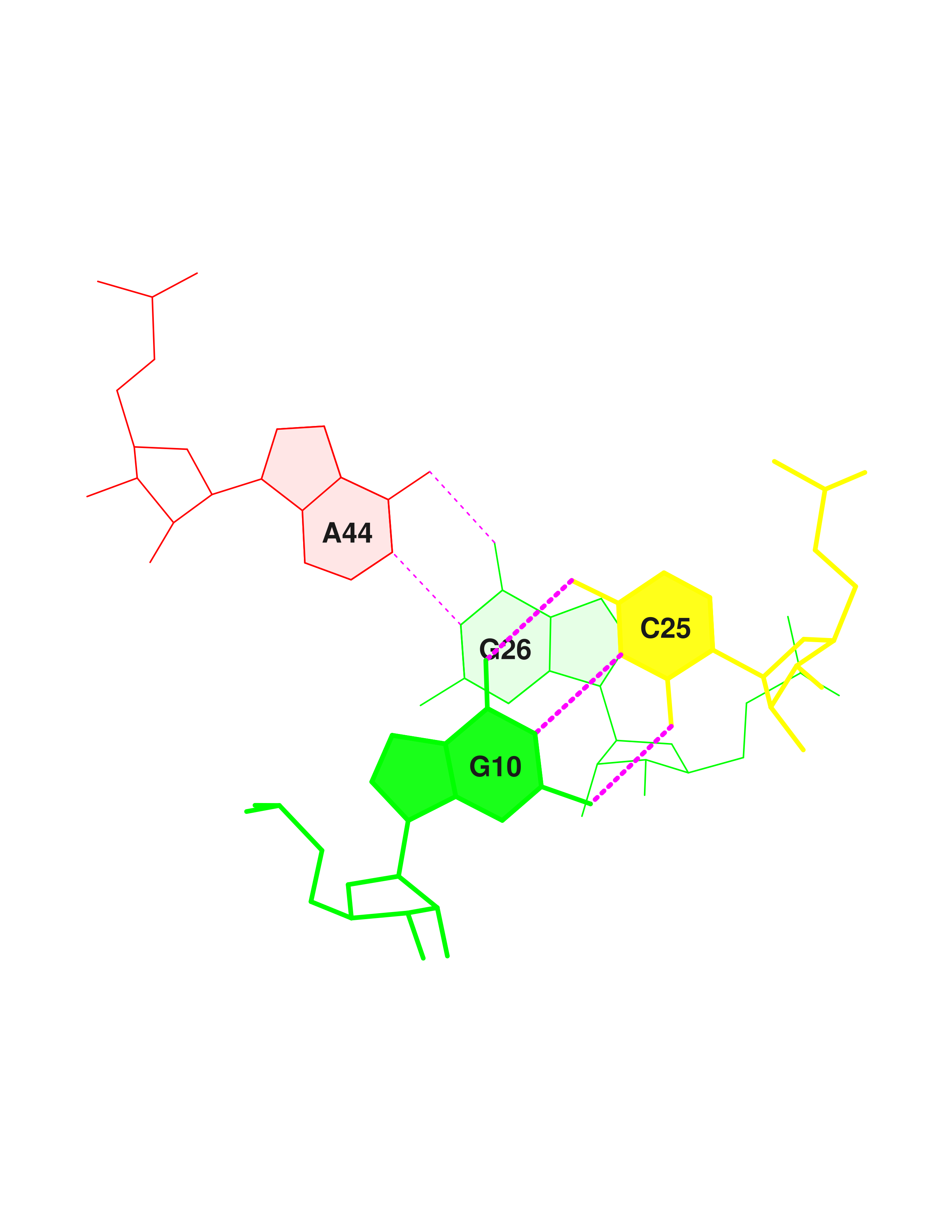
Далее с помощью конструкции find_pair | analyze были проанализированы возможные пары оснований и стебли во вторичной структуре тРНК (отмечены на рис. 4).

Можно видеть, что в тРНК присутствуют неканонические пары оснований (напротив них белые квадраты), а также взаимодействия между основаниями, не входящими в стебли, стабилизирующие третичную структуру (не выделенные прямоугольниками).
Задание 3.3
Определение стекинга в молекуле тРНК происходило также с использованием файла 1i9v.out, полученным вышеупомянутой конструкцией, а изображения были получены ex_str -8 stacking.pdb XXX.pdb и stack2img -cdolt XXX.pdb XXX.ps с дальнейшей конвертацией полученных файлов в формат png.