Практикум 8
Задание 1
Необходимо было поиском в BLAST найти организм и функцию последовательности, определённой в практикуме 6.
Скачать используемую последовательность.
В результате использования и megablast, и blastn со стандартными настройками было выявлено, что это частичная CDS субъединицы 1 цитохром с-оксидазы вида Ophiopholis aculeata семейства Ophiactidae отряда Ophiurida (Настоящие офиуры) класса Ophiuroidea (Офиуры) типа Echinodermata (Иглокожие), митохондриальный геном. Первые 20 совпадений BLAST имели покрытие и процент идентичности, близкие к 100%, что позволяет с уверенностью это утверждать. Пример выравнивания приведён ниже.
Ophiopholis aculeata voucher BIOUG:HUNT0050 cytochrome oxidase subunit 1 (COI) gene, partial cds; mitochondrial
Sequence ID: HM542291.1 Length: 841 Number of Matches: 1
Range 1: 1 to 660
Alignment statistics for match #1 Score Expect Identities Gaps Strand
1168 bits(1295) 0.0 655/660(99%) 0/660(0%) Plus/Plus
Query 42 AACACTATATTTTATATTTGGCGCCTGAGCAGGTACAGTAGGGACTGCCATGAGAAAAAT 101
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1 AACACTATATTTTATATTTGGCGCCTGAGCAGGTACAGTAGGGACTGCCATGAGAAAAAT 60
Query 102 TATACGAGTTGAACTTTCTCAGCCAGGCTCTTTAATACAAGATGATCAAGTATATAAAGT 161
||||||||||||||||||||||||||||||||||||||||||||||||||| ||||||||
Sbjct 61 TATACGAGTTGAACTTTCTCAGCCAGGCTCTTTAATACAAGATGATCAAGTGTATAAAGT 120
Query 162 TATGGTAACGGCCCACGCCTTCGTCATGATATTTTTTATGGTAATGCCCATAATGATAGG 221
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 121 TATGGTAACGGCCCACGCCTTCGTCATGATATTTTTTATGGTAATGCCCATAATGATAGG 180
Query 222 GGGGTTTGGCAAATGACTTGTCCCACTAATGTTAGGAGCGCCTGATATGGCTTTCCCCCG 281
||||||||||||||||||| ||||||||||||||||||||||||||||||||||||||||
Sbjct 181 GGGGTTTGGCAAATGACTTATCCCACTAATGTTAGGAGCGCCTGATATGGCTTTCCCCCG 240
Query 282 AATGaaaaaaaTGAGATTTTGGCTACTACCCCCAGCTTTTATACTTCTTCTAGCTTCAGC 341
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 241 AATGAAAAAAATGAGATTTTGGCTACTACCCCCAGCTTTTATACTTCTTCTAGCTTCAGC 300
Query 342 TGCAAACGAAGGAGGAGTAGGCACTGGATGAACTATTTATCCCCCTTTGTCAGGCCCTAC 401
|||||||||||||||||||||||||||||||||| |||||||||||||||||||||||||
Sbjct 301 TGCAAACGAAGGAGGAGTAGGCACTGGATGAACTGTTTATCCCCCTTTGTCAGGCCCTAC 360
Query 402 CGCACATGCAGGAGGCTGCGTAGACCTCGCAATTTTTTCTCTCCACCTAGCAGGTGCGTC 461
||||||||||||||||||||||||||||||||||||||||||||||||||||||||| ||
Sbjct 361 CGCACATGCAGGAGGCTGCGTAGACCTCGCAATTTTTTCTCTCCACCTAGCAGGTGCATC 420
Query 462 TTCAATTATGGCCTCAATAAAATTTATTACAACTATTATAAATATGCGTAGGCCCGGCAT 521
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 421 TTCAATTATGGCCTCAATAAAATTTATTACAACTATTATAAATATGCGTAGGCCCGGCAT 480
Query 522 GACCATGGATCGACTTCCACTTTTTGCTTGATCTATTTTCTTAACAACTATATTACTACT 581
|||||||||||||||||||||||||| |||||||||||||||||||||||||||||||||
Sbjct 481 GACCATGGATCGACTTCCACTTTTTGTTTGATCTATTTTCTTAACAACTATATTACTACT 540
Query 582 CCTTTCTCTGCCTGTTTTAGCAGGAGCTATTACAATGCTATTAACTGATCGTAACATAAA 641
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 541 CCTTTCTCTGCCTGTTTTAGCAGGAGCTATTACAATGCTATTAACTGATCGTAACATAAA 600
Query 642 AACAACGTTTTTTGATCCTACAGGAGGAGGAGACCCAATACTTTTCCAACATTTATTTTG 701
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 601 AACAACGTTTTTTGATCCTACAGGAGGAGGAGACCCAATACTTTTCCAACATTTATTTTG 660
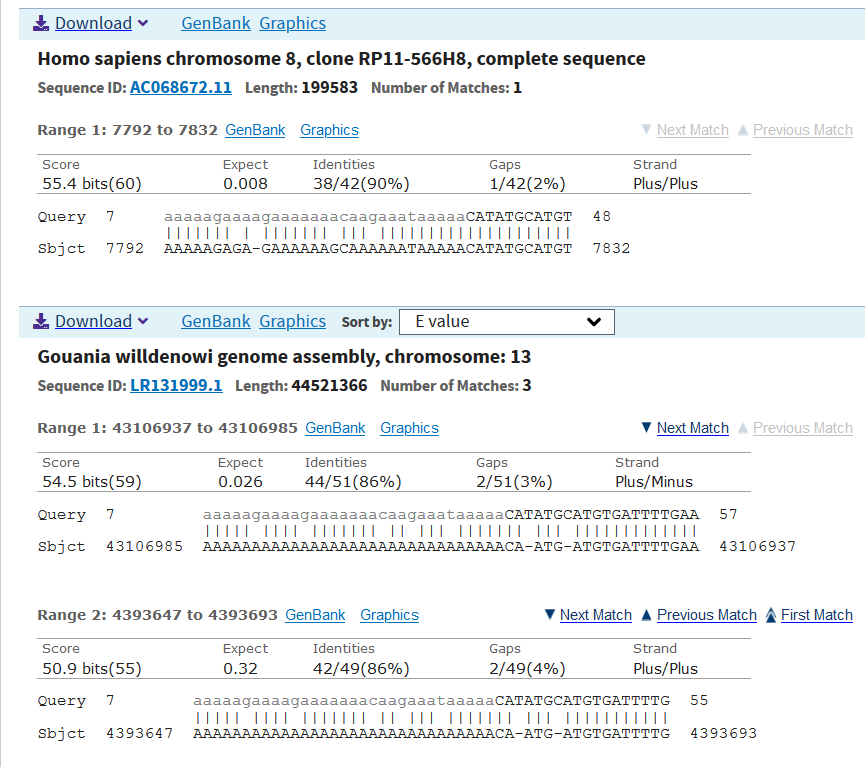
Стоит отметить, что среди первых 50 попаданий была также интересная находка последовательности с высоким сходством, но принадлежащей бабочке вида Agriades zullichi, а не иглокожим.
Agriades zullichi voucher BIOUG:09PROBE-02029 cytochrome oxidase subunit 1 (COI) gene, partial cds; mitochondrial
Sequence ID: GU670181.1 Length: 658 Number of Matches: 1
Range 1: 1 to 658
Alignment statistics for match #1 Score Expect Identities Gaps Strand
1138 bits(616) 0.0 644/658(98%) 0/658(0%) Plus/Plus
Query 42 AACACTATATTTTATATTTGGCGCCTGAGCAGGTACAGTAGGGACTGCCATGAGAAAAAT 101
||||||||| ||||||||||| |||||||||||||||||||| |||||||||||||||||
Sbjct 1 AACACTATACTTTATATTTGGTGCCTGAGCAGGTACAGTAGGAACTGCCATGAGAAAAAT 60
Query 102 TATACGAGTTGAACTTTCTCAGCCAGGCTCTTTAATACAAGATGATCAAGTATATAAAGT 161
||||||||||||||||||||||||||||||||||||||||||||||||||| ||||||||
Sbjct 61 TATACGAGTTGAACTTTCTCAGCCAGGCTCTTTAATACAAGATGATCAAGTGTATAAAGT 120
Query 162 TATGGTAACGGCCCACGCCTTCGTCATGATATTTTTTATGGTAATGCCCATAATGATAGG 221
|||||| |||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 121 TATGGTGACGGCCCACGCCTTCGTCATGATATTTTTTATGGTAATGCCCATAATGATAGG 180
Query 222 GGGGTTTGGCAAATGACTTGTCCCACTAATGTTAGGAGCGCCTGATATGGCTTTCCCCCG 281
||| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 181 GGGATTTGGCAAATGACTTGTCCCACTAATGTTAGGAGCGCCTGATATGGCTTTCCCCCG 240
Query 282 AATGaaaaaaaTGAGATTTTGGCTACTACCCCCAGCTTTTATACTTCTTCTAGCTTCAGC 341
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 241 AATGAAAAAAATGAGATTTTGGCTACTACCCCCAGCTTTTATACTTCTTCTAGCTTCAGC 300
Query 342 TGCAAACGAAGGAGGAGTAGGCACTGGATGAACTATTTATCCCCCTTTGTCAGGCCCTAC 401
|||||||||||||||||||||||||||||||||| |||||||||||||||||||||||||
Sbjct 301 TGCAAACGAAGGAGGAGTAGGCACTGGATGAACTGTTTATCCCCCTTTGTCAGGCCCTAC 360
Query 402 CGCACATGCAGGAGGCTGCGTAGACCTCGCAATTTTTTCTCTCCACCTAGCAGGTGCGTC 461
||||||||||||||||||||||||||||||||||||||||||||||||||||||||| ||
Sbjct 361 CGCACATGCAGGAGGCTGCGTAGACCTCGCAATTTTTTCTCTCCACCTAGCAGGTGCATC 420
Query 462 TTCAATTATGGCCTCAATAAAATTTATTACAACTATTATAAATATGCGTAGGCCCGGCAT 521
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 421 TTCAATTATGGCCTCAATAAAATTTATTACAACTATTATAAATATGCGTAGGCCCGGCAT 480
Query 522 GACCATGGATCGACTTCCACTTTTTGCTTGATCTATTTTCTTAACAACTATATTACTACT 581
|||||||||||||||||| ||||||| |||||||||||||||||||||||||||||| ||
Sbjct 481 GACCATGGATCGACTTCCGCTTTTTGTTTGATCTATTTTCTTAACAACTATATTACTCCT 540
Query 582 CCTTTCTCTGCCTGTTTTAGCAGGAGCTATTACAATGCTATTAACTGATCGTAACATAAA 641
||| |||||||||||||||||||||||||||||||||||| |||||||||||||||||||
Sbjct 541 CCTCTCTCTGCCTGTTTTAGCAGGAGCTATTACAATGCTACTAACTGATCGTAACATAAA 600
Query 642 AACAACGTTTTTTGATCCTACAGGAGGAGGAGACCCAATACTTTTCCAACATTTATTT 699
||||||||||||||||||||||||||| ||||||||||||||||||||||||||||||
Sbjct 601 AACAACGTTTTTTGATCCTACAGGAGGGGGAGACCCAATACTTTTCCAACATTTATTT 658
Задание 2.1 (белок из pr6)
Для сравнения выдачи megablast и blastn (стандартные и чувствительные настройки) я ограничила область поиска семейством Ophiactidae и исключила из выдачи род Ophiopholis, так как его представители в обоих случаях занимали большую часть выдачи. Тем не менее, первым же различием между алгоритмами стало количество найденных последовательностей - 146 для megablast, более 500 (далее не проверяла из-за долгого ожидания) для blastn с любыми настройками.
Настройки чувствительного blastn - длина слова 7 (default 10), match/mismatch score default.
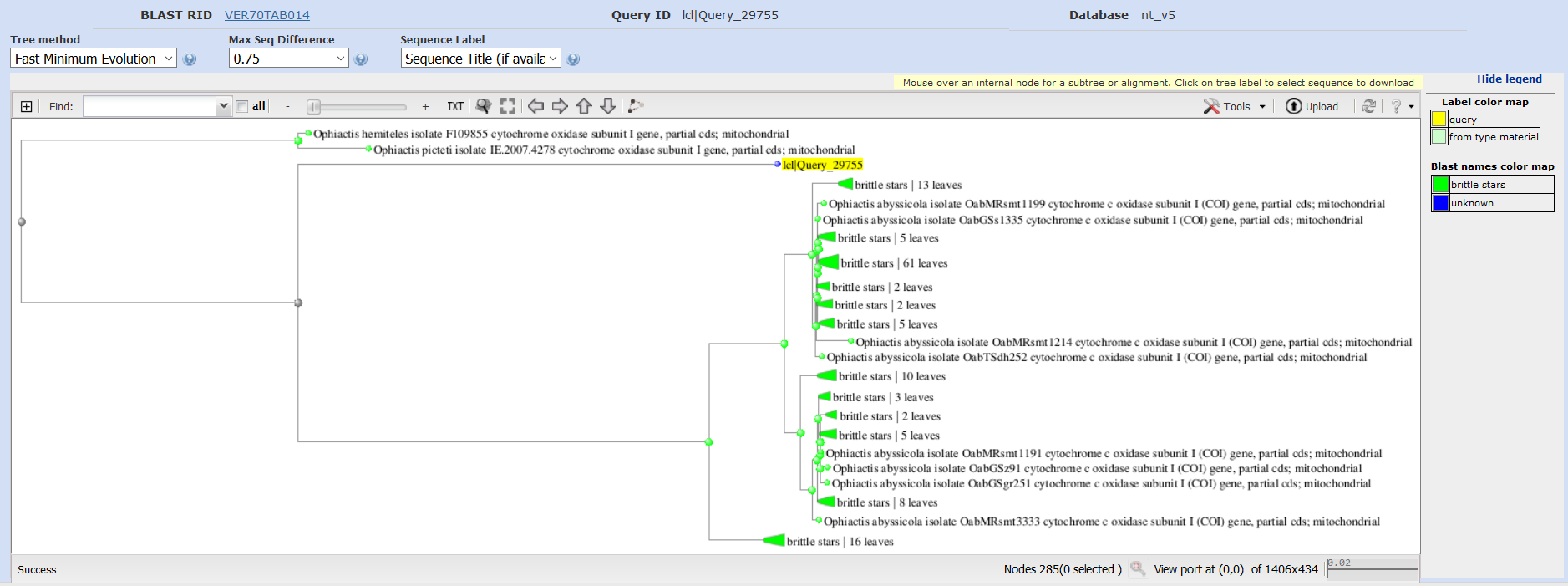
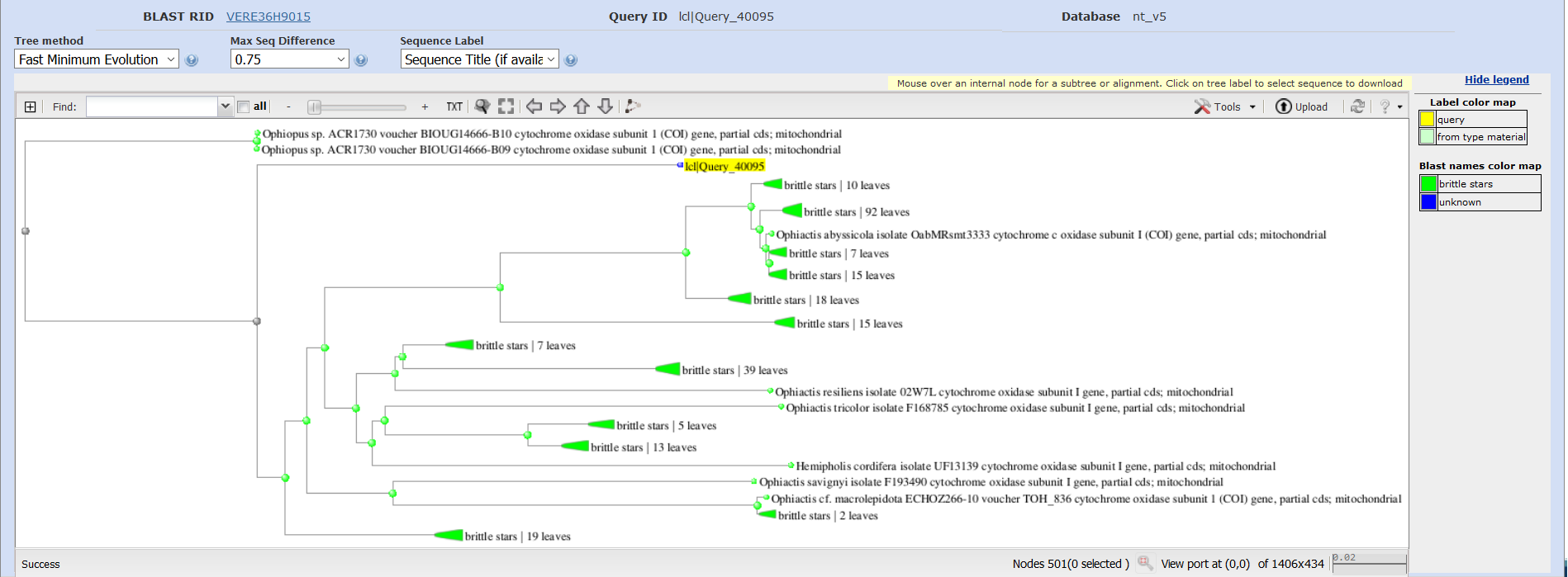
На скриншотах, показывающих топ выдачи с сортировкой по E-value (рис. 1-3), можно определить основные отличия. Во-первых, число находок у megablast значительно меньше, чем в остальных случаях. Во-вторых, никаких отличий между выдачей blastn с разными параметрами не обнаружилось. Во-третьих, megablast выдал в поиске только некоторые виды одного рода Ophiactis исследуемого семейства, в то время как blastn также выдавал, например, другие виды этого рода, а также представителей Ophiopus и Ophiodaphne. Возможно, такой характер выдачи связан с эволюционными взаимоотношениями этих организмов. Действительно, автоматически построенные на основе выдачи BLAST эволюционные деревья (рис. 4-5) подтверждают эту гипотезу. Таким образом, megablast менее чувствителен и более специфичен, чем blastn.
Далее можно увидеть, что одни и те же последовательности имеют различающиеся характеристики при использовании разных алгоритмов, например, Ophiactis hemiteles isolate F109855, но это абсолютно ничего не значит - у нас разные параметры алгоритма.
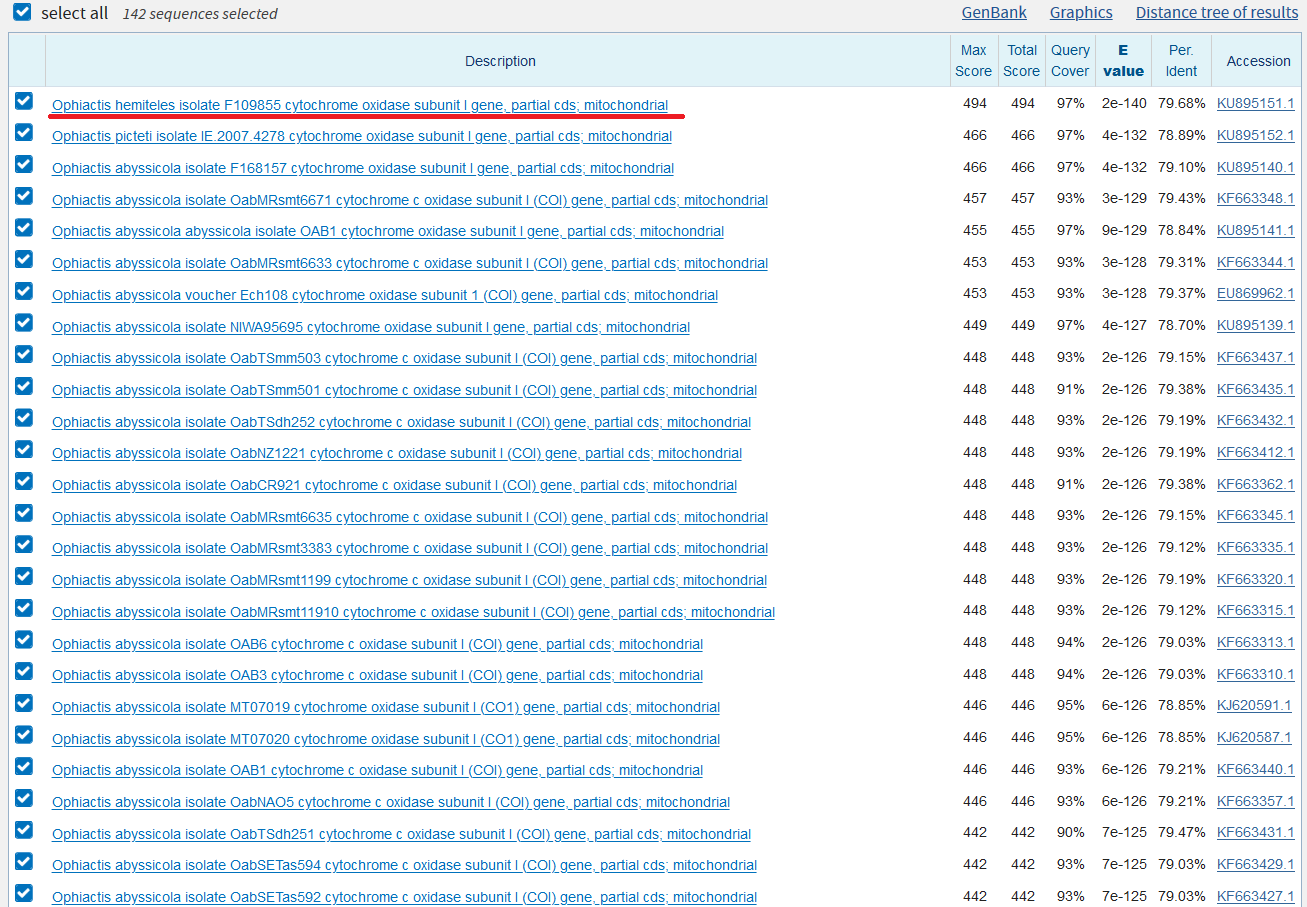
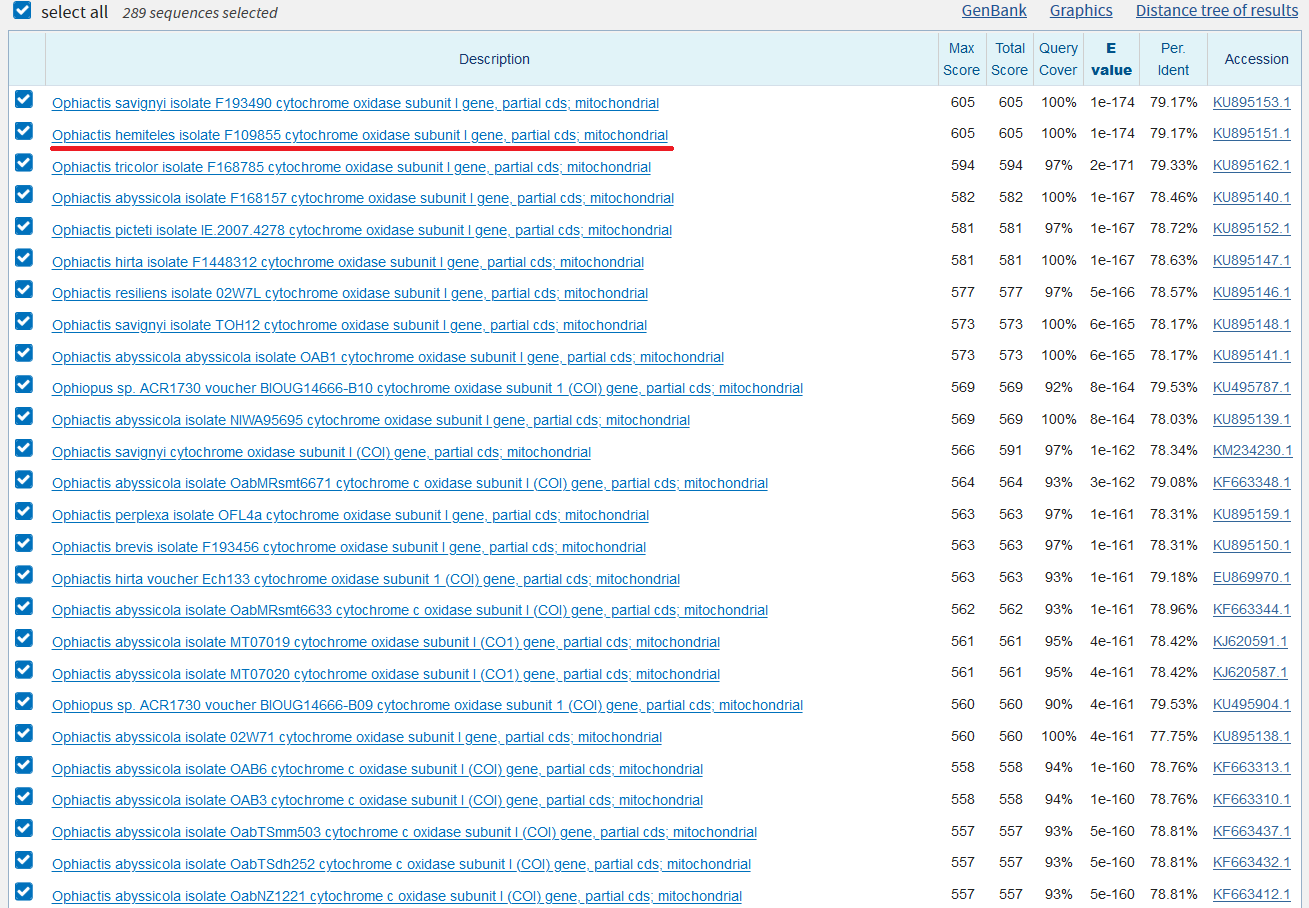
Задание 2.2 (CDS вируса из pr7)
Анализируемая CDS в формате fasta.
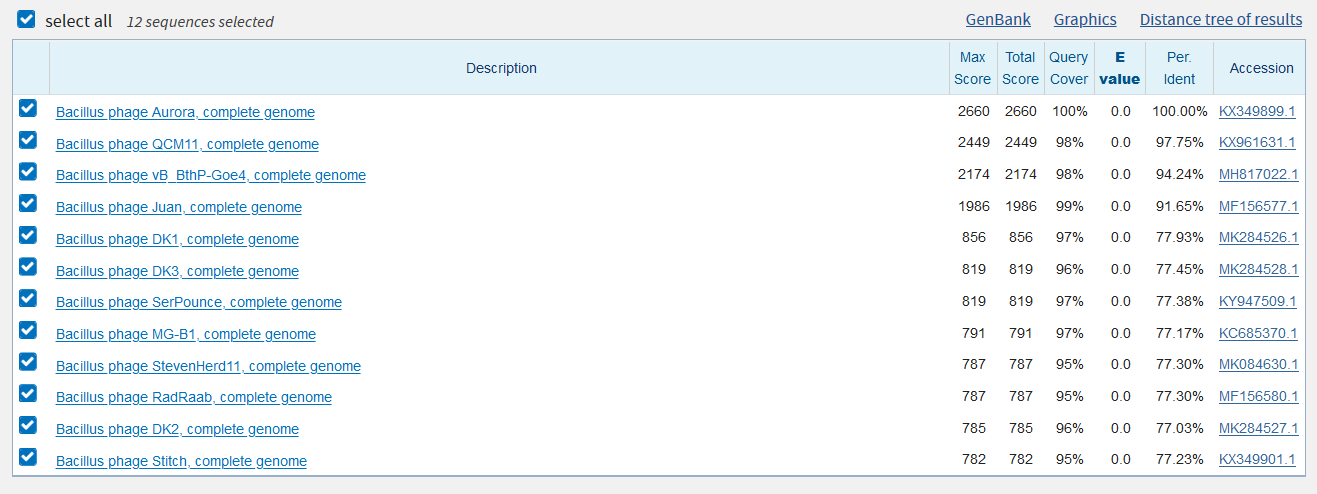
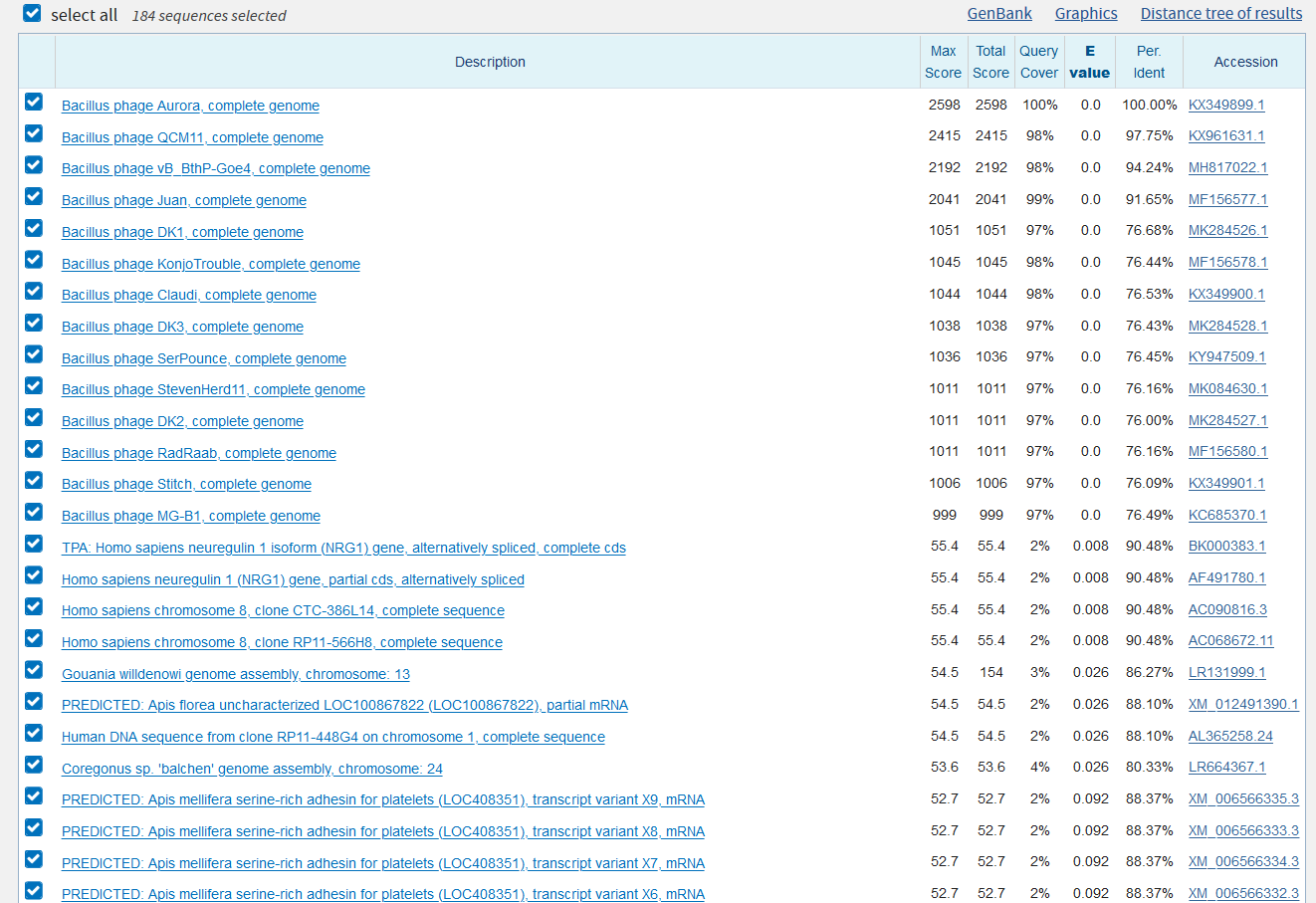
Во второй части, связанной с CDS вируса Aurora, ничего обнаружить не удалось. Хотя я взяла аннотированную как DNA polymerase последовательность, чтобы проверить результаты выдачи BLAST, на рисунках 6 и 7 можно видеть, что megablast не нашёл ничего, кроме вирусов того же семейства (Picoviridae), а blastn среагировал на поли-А последовательность (покрытие менее 10%) (рис. 8), встречаемую у многих видов. Настройка wordsize = 7 привела к сообщению об ошибке (рис. 9) - видимо, подобные поли-А последовательности встречаются слишком часто. Тогда я изменила match/mismatch score на 4/-5 (рис. 10). Во-первых, было найдено больше последовательностей, во-вторых, одна из них даже имела нужную функцию и достаточно высокие показатели. Выравнивание приведено. Тем не менее, это предсказанный белок австралийской зелёной мясной мухи, найденный, вероятно, в результате загрязнения препарата ДНК.
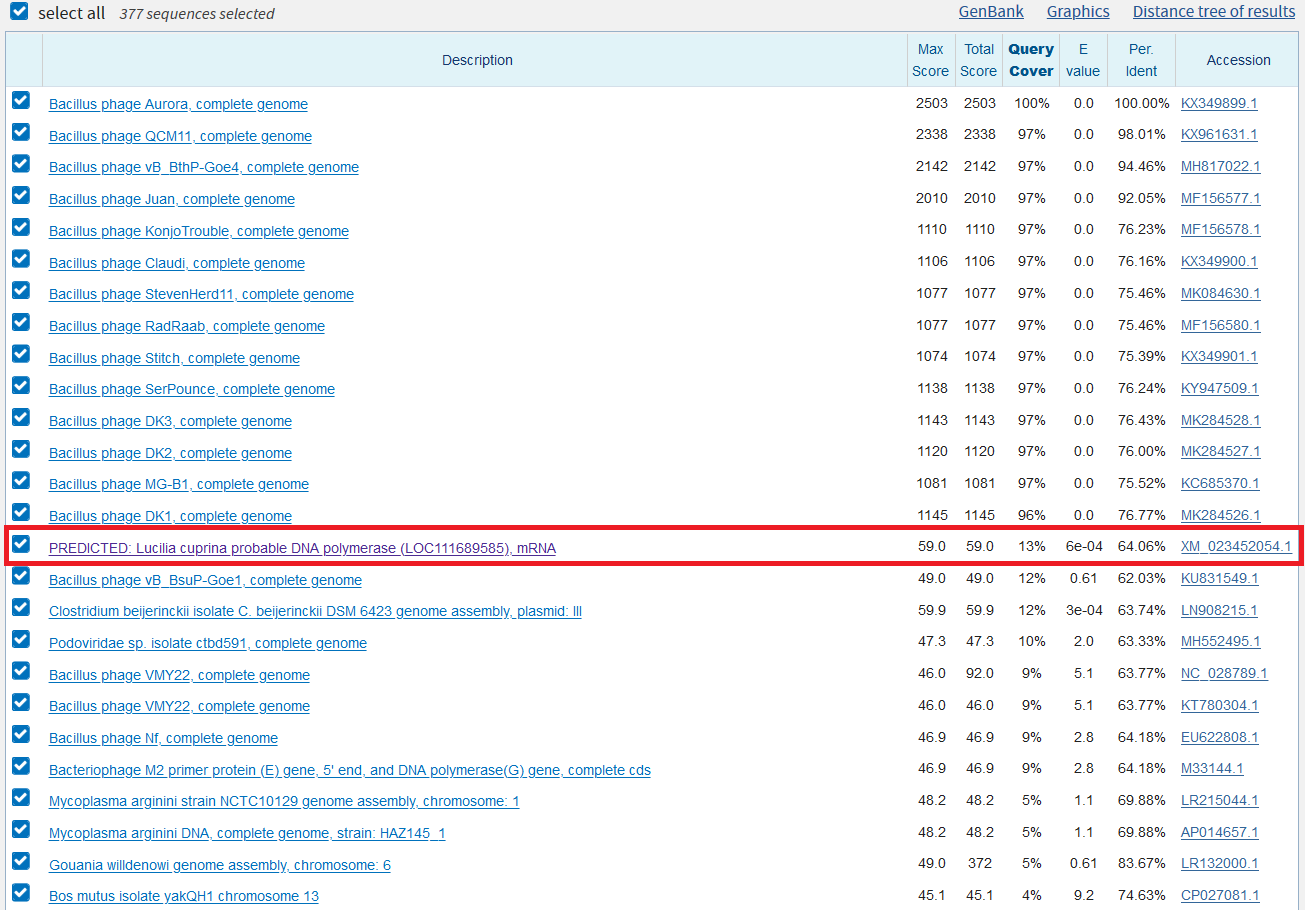
PREDICTED: Lucilia cuprina probable DNA polymerase (LOC111689585), mRNA
Sequence ID: XM_023452054.1Length: 1479Number of Matches: 1
Related Information
Gene-associated gene details
Genome Data Viewer-aligned genomic context
Range 1: 442 to 632GenBankGraphics
Next Match
Previous Match
Alignment statistics for match #1 Score Expect Identities Gaps Strand
59.0 bits(132) 6e-04 123/192(64%) 1/192(0%) Plus/Plus
Query 877 GAAGGTTGTGTGTTTGACATCAACTCAATGTATCCTTATGTTCAATATTATAAAGTTTTA 936
|| ||| ||| ||||| | || || ||||||| | ||| | || |||
Sbjct 442 GACGGTATTGTATTTGATGTTAATTCCCTGTATCCGTCACAAATGTATGACCGAGATTTG 501
Query 937 CCTTATGGTCAACCTGTTCCTTATGAGGGTGAATATGTTGAAGATGAGGAATATCCATTG 996
|||| |||| ||| |||| | ||| |||||||||||| | || | | | || ||
Sbjct 502 CCTTGTGGTGTTCCTATTCCGTTTGAAGGTGAATATGTTTATGACAAATCACACCCCTTA 561
Query 997 TATATACAAAAGGTTCACTTCTCTTTCCAAGTAAAAGATAATATGTTACCAACTATTCAG 1056
||||| ||||| | ||| || || | ||||| ||| | || || |||||
Sbjct 562 TATATTCAAAAACTAACATTCGAGTTTGAATTGAAAGAGAATTATATTCCTACGATTCAA 621
Query 1057 CTAAAGAAACAG 1068
|||| ||||||
Sbjct 622 TTAAA-AAACAG 632
Задание 3
Последовательность белка получала из Uniprot для Saccharomyces cerevisiae - как ближайшего эволюционно из модельных организмов. Для поиска в геноме Amoeboaphelidium protococcarum пользовалась tblastn со стандартными настройками. Геном ради этого нужно превратить в базу данных командой
makeblastdb -in X5.fasta -dbtype nuclи только потом использовать blast, иначе читается только первый скэффолд. Команда tblastn
tblastn -query ###.fasta -db X5.fasta > ###.out
Первый белок: субъединица 1 цитохром c-оксидазы. Белок дыхательной цепи, жизненно важный для всех аэробов. Находится в митохондриях. Покрытие выравнивания 41.2%, что говорит, скорее всего, о наличии гомологичного домена, но вывод о гомологии белков сделать нельзя. Возможно, что в сборку генома не попала митохондрия, что объясняет отсутствие гомолога.
Query= sp|P00401|COX1_YEAST Cytochrome c oxidase subunit 1 OS=Saccharomyces
cerevisiae (strain ATCC 204508 / S288c) OX=559292 GN=COX1 PE=1 SV=2
Length=534
Score E
Sequences producing significant alignments: (Bits) Value
unplaced-887 245 2e-69
scaffold-17 42.4 6e-04
scaffold-170 31.2 1.6
unplaced-991 30.8 2.2
scaffold-104 28.5 9.7
> unplaced-887
Length=30515
Score = 245 bits (626), Expect = 2e-69, Method: Compositional matrix adjust.
Identities = 127/220 (58%), Positives = 156/220 (71%), Gaps = 11/220 (5%)
Frame = -2
Query 284 GFLVWSHHMYIVGLDADTRAYFTSATMIIAIPTGIKIFSWLATIHGGSIRLATPMLYAIA 343
G ++ HHMY VGLD DTR+YF++ATMIIA+PTGIKIFSW+AT+ GG I ++P+L+ I
Sbjct 24382 GKYIFRHHMYTVGLDVDTRSYFSAATMIIAVPTGIKIFSWIATLAGGRINFSSPILFIIG 24203
Query 344 FLFLFTMGGLTGVALANASLDVAFHDTYYVVGHFHYVLSMGAIFSLFAGYYYWSPQILGL 403
FL LFT+GGLTGV L+NA LDV+ HDTYYVV HFHYVLSMGA+F+LFAG+YYW P I
Sbjct 24202 FLILFTLGGLTGVVLSNAPLDVSLHDTYYVVAHFHYVLSMGAVFALFAGFYYWYPTITHK 24023
Query 404 NYNEKLAQIQFWLIFIGANVIFFPMHFLGINGMPRRIPDYPDAFAGWNYVASIGSFIATL 463
NE A+I F LIFIG NV F PMH LG+ G PRRI DYPD+F G N +AS GSFI+ +
Sbjct 24022 MSNELWAKIHFALIFIGVNVTFGPMHILGMAGHPRRILDYPDSFLGINQLASFGSFISFI 23843
Query 464 SLFLFIYILYDQlvnglnnkvnnkSVIYNKAPDFVESNTI 503
S+ F+ +Y + VIYN F NT+
Sbjct 23842 SIIPFLLSIY-----------FSNKVIYNNLSSFSLDNTV 23756
Следующий белок - ДНК-полимераза 3 (дельта), присутствующая у всех эукариот. Есть гомолог, даже в двух разных участках генома. Покрытие 87%.
Query= sp|P15436|DPOD_YEAST DNA polymerase delta catalytic subunit
OS=Saccharomyces cerevisiae (strain ATCC 204508 / S288c) OX=559292
GN=POL3 PE=1 SV=4
Length=1097
Score E
Sequences producing significant alignments: (Bits) Value
scaffold-359 1049 0.0
scaffold-81 1037 0.0
unplaced-816 181 6e-46
scaffold-423 154 1e-37
scaffold-424 145 6e-35
scaffold-17 40.8 0.004
scaffold-105 38.5 0.022
scaffold-22 33.5 0.69
> scaffold-359
Length=583372
Score = 1049 bits (2712), Expect = 0.0, Method: Compositional matrix adjust.
Identities = 531/954 (56%), Positives = 693/954 (73%), Gaps = 24/954 (3%)
Frame = +3
Query 145 EGHSVLCNVTGFKNYLYVPAPNSSDANDQEQINKFVHYLNETFD----HAIDSIEVVSKQ 200
+G+SVLC+V GF Y YVPAP + +++ + +F LN+ + A+ +E++ K+
Sbjct 102894 DGNSVLCHVHGFFPYFYVPAPPNFNSS---HLMEFTRSLNQNVEGAGGKAVLLVELMKKK 103064
Query 201 SIWGYSGDTKLPFWKIYVTYPHMVNKLRTAFERGHLSFNSWFSNGTTT---YDNIAYTLR 257
+I+GY G F KI V+ P +++K R E G F F + T NIAY LR
Sbjct 103065 TIYGYYG*EDSTFLKITVSSPKLISKARGILESGAFHFPG-FPHAVTLPGFESNIAYELR 103241
Query 258 LMVDCGIVGMSWITLPKGKYSMIEPNNRVSSCQLEVSINYRNLIAHPAEGDWSHTAPLRI 317
M+DC IVG +WI LP GK+ + + S +EV + Y +LI+H EG+WS APLRI
Sbjct 103242 FMIDCKIVGANWIELPAGKWCVRTGKDHTSHA*IEVDVFYEDLISHAPEGEWSKVAPLRI 103421
Query 318 MSFDIECAGRIGVFPEPEYDPVIQIANVVSIAGAKKPFIRNVFTLNTCSPITGSMIFSHA 377
+SFDIECAGR GVFP+P++D VIQIAN+V+I G KP IRN+FTL C I GS I S+
Sbjct 103422 LSFDIECAGRKGVFPDPQHDSVIQIANMVTIQG*SKPLIRNIFTLKECGHIVGSHILSYE 103601
Query 378 TEEEMLSNWRNFIIKVDPDVIIGYNTTNFDIPYLLNRAKALKVND-FPYFGRLKTVKQEI 436
E ++L W +FI +VDPD++ GYN NFD+PYL++RAKALK D F GR++ +I
Sbjct 103602 DEAQLL*KWSDFIKEVDPDIVTGYNINNFDMPYLVDRAKALKCKDSFFNLGRIRGKLSQI 103781
Query 437 KESVFSSKAYGTRETKNVNIDGRLQLDLLQFIQREYKLRSYTLNAVSAHFLGEQKEDVHY 496
K++ FSSKAYG RE + V ++GR+ LD++ IQR++KLRSY+LN+VSAHFLGE KEDV +
Sbjct 103782 KDTRFSSKAYGNREGREVALEGRVILDMI*IIQRDHKLRSYSLNSVSAHFLGE*KEDVPH 103961
Query 497 SIISDLQNGDSETRRRLAVYCLKDAYLPLRLMEKLMALVNYTEMARVTGVPFSYLLARGQ 556
SII+DLQNGD+ TRRRLAVYCLKDAYLP RL++KLM ++NY EMARVTGVP +YLL+RG
Sbjct 103962 SIITDLQNGDA*TRRRLAVYCLKDAYLP*RLLDKLMVVINYMEMARVTGVPLNYLLSRG* 104141
Query 557 QIKVVSQLFRKCLEIDTVIPNMQSQASDDQYEGATVIEPIRGYYDVPIATLDFNSLYPSI 616
IKVVS L+RK E + VIP M+S+ SD+ YEGATVIEP R +Y PIATLDF+SLYPSI
Sbjct 104142 *IKVVS*LYRKAKEENLVIPAMKSEGSDE*YEGATVIEPKRAFYKDPIATLDFSSLYPSI 104321
Query 617 MMAHNLCYTTLC-NKATVERLNLKIDEDYVITPNGDYFVTTKRRRGILPIILDELISARK 675
MMAHNLCY+TL N ++ L L D DY++TP+GD FV + R+GILP IL++LI ARK
Sbjct 104322 MMAHNLCYSTLVMNNQVIDALKLVKD*DYIVTPSGDKFVKSTVRKGILPTILEDLIGARK 104501
Query 676 RAKKDLRDEKDPFKRDVLNGRQLALKISANSVYGFTGATVGKLPCLAISSSVTAYGRTMI 735
RAK DL+ E DPFKR VL+GRQLALKISANSVYGFTGATVGKLPCL +S SVTAYGR MI
Sbjct 104502 RAKADLKKETDPFKRAVLDGRQLALKISANSVYGFTGATVGKLPCLQVS*SVTAYGREMI 104681
Query 736 LKTKTAVQEKYCIKNGYKHDAVVVYGDTDSVMVKFGTTDLKEAMDLGTEAAKYVSTLFKH 795
TK V++++C NGYKHDA VVYGDTDSVMVKFGT DLKEAMDLG EAA++++ F
Sbjct 104682 DFTKDLVEKQFCKANGYKHDADVVYGDTDSVMVKFGTEDLKEAMDLGREAAEFITQRFVK 104861
Query 796 PINLEFEKAYFPYLLINKKRYAGLFWTNPDKFDKLDQKGLASVRRDSCSLVSIVMNKVLK 855
PI LEFEK Y+PYLLINKKRYAGL+WT+ +K DK+D KGL +VRRD+C LVS V++ LK
Sbjct 104862 PIKLEFEKVYWPYLLINKKRYAGLYWTSVEKPDKMDTKGLETVRRDNCKLVSTVVDTCLK 105041
Query 856 KILIERNVDGALAFVRETINDILHNRVDISKLIISKTLAP---NYTNPQPHAVLAERMKR 912
IL+E++V+ A+ +V+ TI+++L N+VD+S L+I+K L+ +Y H LAERM++
Sbjct 105042 MILMEKDVEKAVNYVKSTISELLQNKVDLSLLVITKQLSKSGEDYAGK*AHVELAERMRK 105221
Query 913 RE-GVGPNVGDRVDYVII--GGNDKLYNRAEDPLFVLENNIQVDSRYYLTNQLQNPIISI 969
R+ G P +GDRV YVII Y RAEDP++VLENNI +D++YYL NQL+NP++ I
Sbjct 105222 RDAGSAPALGDRVAYVIIKAAKGSAAYERAEDPIYVLENNIPIDTKYYLDNQLKNPLMRI 105401
Query 970 VAPIIGDKQANGMFV---VKSIKINTGSQKGGLMSFIKKVEACKSCKGPLRKGEGPLCSN 1026
PI+ + ++ +F +SI++ T S GGLM F K C CK L+ +C +
Sbjct 105402 FEPILPNAESQ-LFSGEHTRSIQV-TSSSAGGLMKFTVKKMTCLGCKAVLKD*NAAVCQH 105575
Query 1027 CLARSGELYIKALYDVRDLEEKYSRLWTQCQRCAGNLHSEVLCSNKNCDIFYMR 1080
CL + +LY+K L +V +LE ++SRLWTQCQRC G+LH +VLC++K+C IFYMR
Sbjct 105576 CLPKINQLYMKQLDNVNELENRFSRLWTQCQRCQGSLHQDVLCTSKDCPIFYMR 105737
> scaffold-81
Length=367914
Score = 1037 bits (2682), Expect = 0.0, Method: Compositional matrix adjust.
Identities = 532/954 (56%), Positives = 689/954 (72%), Gaps = 24/954 (3%)
Frame = -2
Query 145 EGHSVLCNVTGFKNYLYVPAPNSSDANDQEQINKFVHYLNETFD----HAIDSIEVVSKQ 200
+G+SVLC+V GF Y YVPAP + +++ + +F LN + A+ +E++ K+
Sbjct 343361 DGNSVLCHVHGFFPYFYVPAPPNFNSS---HLMEFTCSLN*NVESAGGKAVLLVELMKKK 343191
Query 201 SIWGYSGDTKLPFWKIYVTYPHMVNKLRTAFERGHLSFNSWFSNGTTT---YDNIAYTLR 257
+I+GY G F KI V+ P ++K R E G F F + T NIAY LR
Sbjct 343190 TIYGYYG*DDSTFLKITVSSPKFISKARGILESGAFHFPG-FPHAVTLPGFESNIAYELR 343014
Query 258 LMVDCGIVGMSWITLPKGKYSMIEPNNRVSSCQLEVSINYRNLIAHPAEGDWSHTAPLRI 317
M+DC IVG +WI LP GK+ + + S +EV + Y +LI+H EG+WS APLRI
Sbjct 343013 FMIDCKIVGANWIELPAGKWCVRTGKDHTSHA*IEVDVFYEDLISHAPEGEWSKVAPLRI 342834
Query 318 MSFDIECAGRIGVFPEPEYDPVIQIANVVSIAGAKKPFIRNVFTLNTCSPITGSMIFSHA 377
+SFDIECAGR GVFP+P +D VIQIAN+V+I G KP IRN+FTL C I GS I S+
Sbjct 342833 LSFDIECAGRKGVFPDP*HDSVIQIANMVTIQG*NKPLIRNIFTLKECGHIVGSHILSYE 342654
Query 378 TEEEMLSNWRNFIIKVDPDVIIGYNTTNFDIPYLLNRAKALKVND-FPYFGRLKTVKQEI 436
E +L W +FI +VDPD++ GYN NFD+PYL++RAKALK D F GR++ I
Sbjct 342653 DEALLL*KWSDFIKEVDPDIVTGYNINNFDMPYLVDRAKALKCKDSFFNLGRIRGKMS*I 342474
Query 437 KESVFSSKAYGTRETKNVNIDGRLQLDLLQFIQREYKLRSYTLNAVSAHFLGEQKEDVHY 496
K++ FSSKAYG RE + V ++GR+ LD++ IQR++KLRSY+LN+VSAHFLGE KEDV +
Sbjct 342473 KDTRFSSKAYGNREGREVALEGRVILDMI*IIQRDHKLRSYSLNSVSAHFLGE*KEDVPH 342294
Query 497 SIISDLQNGDSETRRRLAVYCLKDAYLPLRLMEKLMALVNYTEMARVTGVPFSYLLARGQ 556
SII+DLQNGD+ TRRRLAVYCLKDAYLP RL++KLM ++NY EMARVTGVP +YLL+RGQ
Sbjct 342293 SIITDLQNGDA*TRRRLAVYCLKDAYLP*RLLDKLMVVINYMEMARVTGVPLNYLLSRGQ 342114
Query 557 QIKVVSQLFRKCLEIDTVIPNMQSQASDDQYEGATVIEPIRGYYDVPIATLDFNSLYPSI 616
IKVVS L+RK E + VIP M+S+ SD+ YEGATVIEP R +Y PIATLDF+SLYPSI
Sbjct 342113 *IKVVS*LYRKAKEENLVIPAMKSEGSDE*YEGATVIEPKRAFYKDPIATLDFSSLYPSI 341934
Query 617 MMAHNLCYTTLC-NKATVERLNLKIDEDYVITPNGDYFVTTKRRRGILPIILDELISARK 675
MMAHNLCY+TL N ++ L L D+DY++TP+GD FV + R+GILP IL++LI ARK
Sbjct 341933 MMAHNLCYSTLVMNNQVIDTLKLVKDKDYIVTPSGDKFVKSTVRKGILPTILEDLIGARK 341754
Query 676 RAKKDLRDEKDPFKRDVLNGRQLALKISANSVYGFTGATVGKLPCLAISSSVTAYGRTMI 735
RAK DL+ E DPFKR VL+GRQLALKISANSVYGFTGATVGKLPCL +S SVTAYGR MI
Sbjct 341753 RAKADLKKETDPFKRAVLDGRQLALKISANSVYGFTGATVGKLPCLQVS*SVTAYGREMI 341574
Query 736 LKTKTAVQEKYCIKNGYKHDAVVVYGDTDSVMVKFGTTDLKEAMDLGTEAAKYVSTLFKH 795
TK V++++C NGYKHDA VVYGDTDSVMVKFGT DLKEAMDLG EAA++++ F
Sbjct 341573 DFTKDLVEKQFCKANGYKHDADVVYGDTDSVMVKFGTEDLKEAMDLGREAAEFIT*RFVK 341394
Query 796 PINLEFEKAYFPYLLINKKRYAGLFWTNPDKFDKLDQKGLASVRRDSCSLVSIVMNKVLK 855
PI LEFEK Y+PYLLINKKRYAGL+WT+ DK DK+D KGL +VRRD+C LVS V++ LK
Sbjct 341393 PIKLEFEKVYWPYLLINKKRYAGLYWTSVDKPDKMDTKGLETVRRDNCKLVSTVVDTCLK 341214
Query 856 KILIERNVDGALAFVRETINDILHNRVDISKLIISKTLAP---NYTNPQPHAVLAERMKR 912
IL+E++V+ A+ +V+ TI+++L N+VD+S+L+I+K L+ +Y H LAERM++
Sbjct 341213 MILMEKDVEKAVNYVKNTISELLQNKVDLSQLVITKQLSKSGEDYAGK*AHVELAERMRK 341034
Query 913 RE-GVGPNVGDRVDYVII--GGNDKLYNRAEDPLFVLENNIQVDSRYYLTNQLQNPIISI 969
R+ G P +GDRV YVII Y RAEDP++VLENNI +D++YYL NQL+NP++ I
Sbjct 341033 RDAGSAPALGDRVAYVIIKAAKGSAAYERAEDPIYVLENNIPIDTKYYLDNQLKNPLMRI 340854
Query 970 VAPIIGDKQANGMFV---VKSIKINTGSQKGGLMSFIKKVEACKSCKGPLRKGEGPLCSN 1026
PI+ + ++ +F +SI++ T S GGLM F K C CK L+ +C +
Sbjct 340853 FEPILPNAESQ-LFSGEHTRSIQV-TSSSAGGLMKFTVKKMTCLGCKAVLKN*NAAVCQH 340680
Query 1027 CLARSGELYIKALYDVRDLEEKYSRLWTQCQRCAGNLHSEVLCSNKNCDIFYMR 1080
CL + LY+K L +V +LE ++SRLWTQC RC G+LH +VLC++K+C IFYMR
Sbjct 340679 CLPKIN*LYMKQLDNVNELENRFSRLWTQC*RCQGSLHQDVLCTSKDCPIFYMR 340518
А вот с последним белком красивой картинки не получилось. Это одна из субъединиц ДНК-зависимой РНК-полимеразы, которая встречается у всех эукариот, но, видимо, менее консервативна, чем я представляла. Тем не менее, покрытие 79.2% и консервативность домена позволяют предположить гомологию белков.
Query= sp|P38902|RPB11_YEAST DNA-directed RNA polymerase II subunit RPB11
OS=Saccharomyces cerevisiae (strain ATCC 204508 / S288c) OX=559292
GN=RPB11 PE=1 SV=1
Length=120
Score E
Sequences producing significant alignments: (Bits) Value
scaffold-287 87.4 2e-20
scaffold-44 86.7 4e-20
scaffold-282 43.5 9e-06
unplaced-804 28.5 0.91
> scaffold-287
Length=687983
Score = 87.4 bits (215), Expect = 2e-20, Method: Compositional matrix adjust.
Identities = 43/95 (45%), Positives = 59/95 (62%), Gaps = 0/95 (0%)
Frame = -1
Query 1 MNAPDRFELFLLGEGESKLKIDPDTKAPNAVVITFEKEDHTLGNLIRAELLNDRKVLFAA 60
MNAP R E+ +L +G K+ + D K PNA +EDHTLGN++R LL + KVLFAA
Sbjct 237308 MNAPARHEIIVLPDGVKKITVKQDIKIPNAATFEI*REDHTLGNVLREYLL*NPKVLFAA 237129
Query 61 YKVEHPFFARFKLRIQTTEGYDPKDALKNACNSII 95
YK+ HP +L++Q T P++A+ A N II
Sbjct 237128 YKMPHPLNPWIELKVQVTADTTPRNAVIQALNRII 237024
> scaffold-44
Length=398337
Score = 86.7 bits (213), Expect = 4e-20, Method: Compositional matrix adjust.
Identities = 43/95 (45%), Positives = 59/95 (62%), Gaps = 0/95 (0%)
Frame = +1
Query 1 MNAPDRFELFLLGEGESKLKIDPDTKAPNAVVITFEKEDHTLGNLIRAELLNDRKVLFAA 60
MNAP R E+ +L +G K+ + D K PNA ++EDHTLGN++R LL + KVLFAA
Sbjct 256414 MNAPARHEIIVLPDGVKKITVK*DIKIPNAATFEIQREDHTLGNVLREYLL*NPKVLFAA 256593
Query 61 YKVEHPFFARFKLRIQTTEGYDPKDALKNACNSII 95
YK+ HP +L++Q T P+ A+ A N II
Sbjct 256594 YKMPHPLNPWIELKVQVTADTTPRKAVI*ALNRII 256698
Задание 4
Для анализа мной был выбран геном Amanita muscaria Koide, скэффолд 15_c10 длиной 56,323 нуклеотидов.
С помощью инструмента blastx с использованием базы данных SwissProt был найден один хороший белок, который определяется точно, и ещё несколько возможных, что заметно из графической интерпретации выдачи BLAST.

Точно определённый белок - альфа-субъединица белкового транспортера белков SEC61 (Protein transport protein SEC61 subunit alpha). Совпадения найдены в основном среди грибов. Координаты из выравниваний определяются достаточно точно, видны возможные экзоны и интроны с точностью до 15 нуклеотидов. Ex1: 11890-11970, Ex2: 12033-12311, Ex3: 12359-13264, Ex4: 13326-13496.
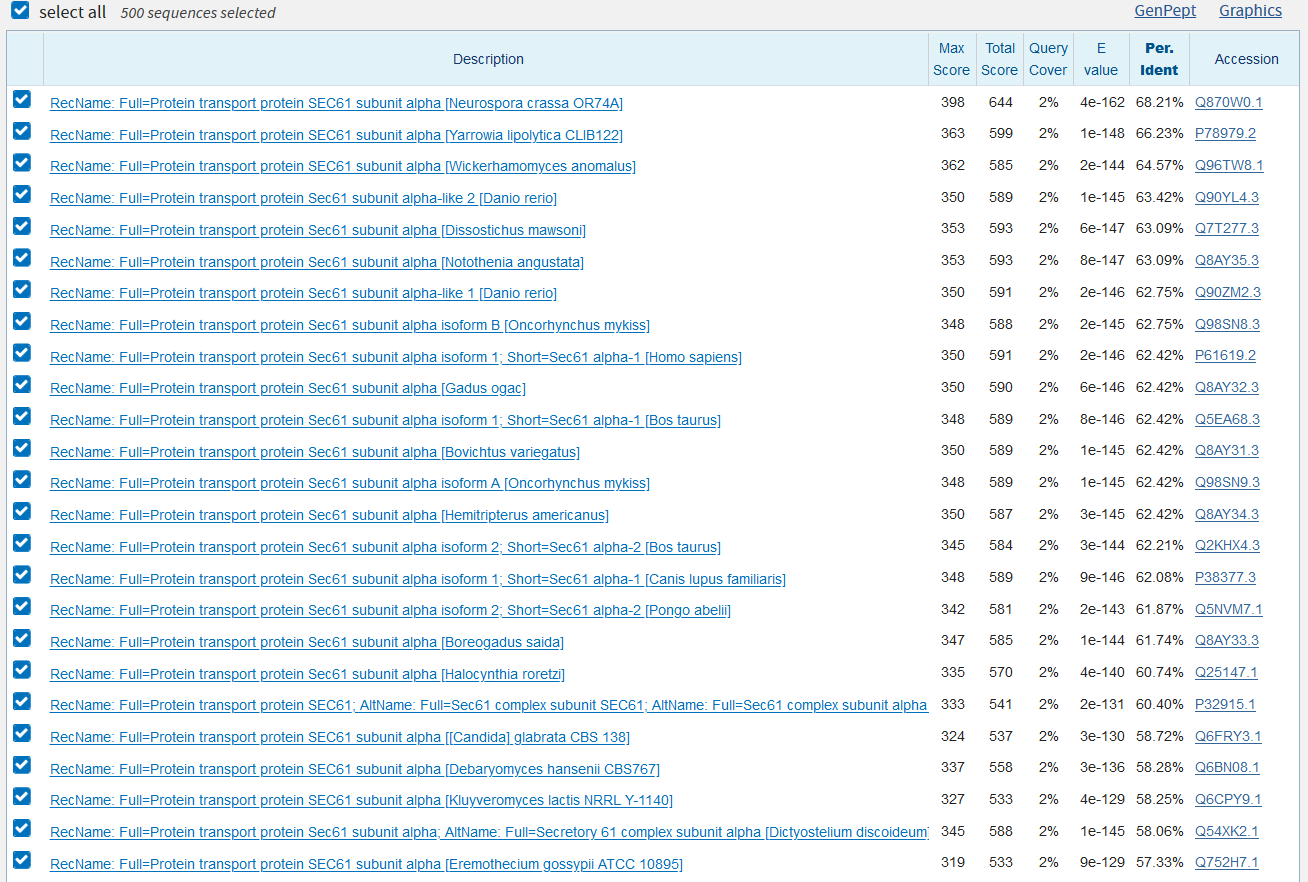
Примеры выравниваний:
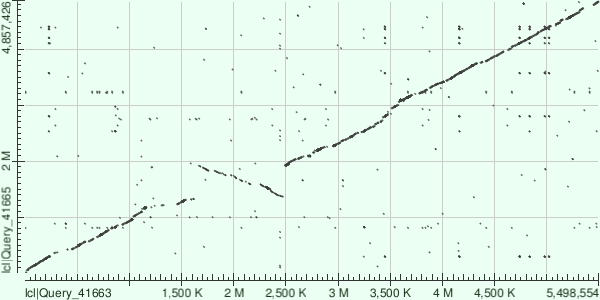
Задание 5
С помощью megablast для двух последовательностей я сравнила геномы Escherichia coli str. Sakai и Salmonella enterica subsp. enterica serovar Typhimurium str. LT2. Чтобы снизить уровень шума, я использовала именно megablast, а не blastn. Видно, что в геномах много совпадающих участков, а также видно две крупных перестройки - участкок генома с 1200 до 1450 тысяч п.н. у E. coli возник заново (возможно, в результате того, что в хромосому встроился вирус или плазмида), участок генома с 1510 до 2500 тысяч п.н. претерпел инверсию. Также видно много шумовых совпадающих последовательностей в разных участках генома и более короткие заново возникшие у E. coli участки, например с 35 до 55 тысяч п.н.