Практикум 4. Филогения белков
Задание 1. BLASTP
Используемые бактерии
- Streptococcus pyogenes serotype M1 - STRP1 (изучается STREQ)
- Streptococcus pneumoniae serotype 4 - STRPN
- Lactococcus lactis subsp. cremoris - LACLM (изучается LACLA)
- Lactobacillus acidophilus - LACAC
- Enterococcus faecalis - ENTFA
- Listeria monocytogenes serovar 1/2a - LISMO
- Bacillus subtilis - BACSU
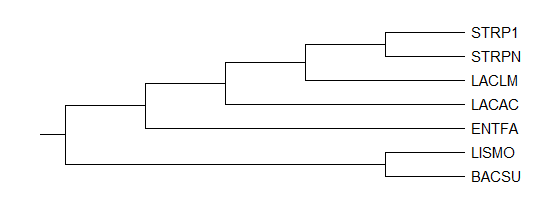
Скобочная формула
(((((STREQ,STRPN),LACLA),LACAC),ENTFA),(LISMO,BACSU))
Филогенетическое дерево бактерий
Результаты BLASTP для CLPX_ECOLI по протеомам бактерий (порог E-value = 0.00001)
Задание 2. Филогенетические деревья белков

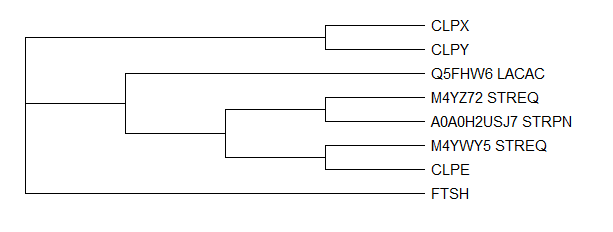
Первое: найдены гомологи белка CLPX - АТФ-связывающая субъединица АТФ-зависимой протеазы ClpP - во всех бактериях, кроме STREQ. Филогения, построенная по белкам, в этом случае частично не совпадает с реальной (ENTFA и LACAC). Второе: найдены гомологи CLPY (HSIU - другое название) у LISMO, BACSU, LACAC и ENTFA. Филогения, построенная по белкам, совпадает с реальной. Третье: найдены 4 ортолога и 2 паралога АТФ-зависимой протеазы Clp (мнемоника ClpE у BACSU) у LACAC, STRPN, LISMO, BACSU и STREQ (у которого два паралога). Паралог CLPC/MecB STREQ - negative regulator of genetic competence, белок теплового шока, АТФ-зависимый шаперон, что объясняет его похожесть. АТФ-связвающие домены одного происхождения похожи, но всё же это паралог, поэтому схлопываем только два ортолога. Четвёртое: АТФ-зависимая цинковая металлопротеаза FTSH у STREQ и STRPN.