Валидация РСА-модели
Задание 1. Анализ качества модели с помощью данных из PDB и анализа электронной плотности
В задании 1 необходимо провести изучение PDB-структуры, полученной методом РСА: 5EHR (разрешение 1.7 ангстрем). Стоит сразу уточнить, что эта структура имеет несколько версий, исследование проводится на основе текущей версии (1.2). Также в структуре имеются две цепи, А и В, аналогичного аминокислотного состава. Если не указано обратное, без упоминания цепи здесь и далее подразумеваются остатки цепи А. У 5 аминокислот в структуре есть альт-локи.
Белок, репрезентируемый изучаемой структурой - нерецепторная тирозин-фосфатаза SHP2 в комплексе с аллостерическим ингибитором SHP099. В оригинальной публикации [1] упоминается, что целью расшифовровки трёхмерной структуры было уточнение сайта связывания новой (на тот момент) молекулы ингибитора SHP099. Согласно результатам исследования, аллостерический ингибитор связывается близко к активному сайту тирозин-фосфатазы и является эффективным ингибиторов сигнальных каскадов в раковых клетках человека in vitro.
Отчёт по валидации структуры 5EHR из PDB.
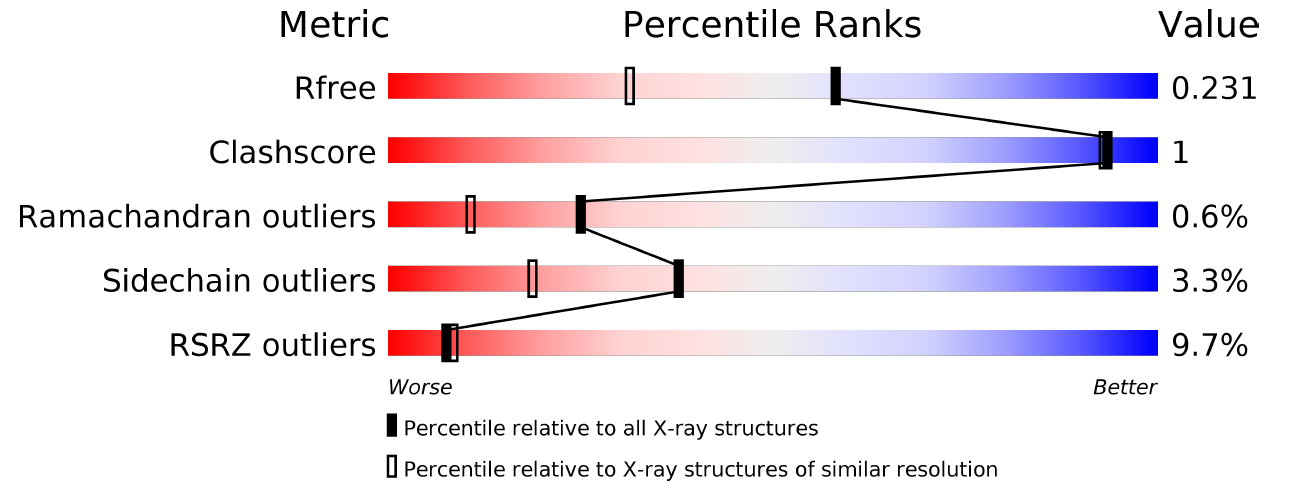
В отчёте по валидации можно найти большую часть информации, необходимой для анализа качества структуры, а также показатели критериев, по которым оно оценивается. Расшифровку критериев можно найти в соответствующей документации на сайте PDB [2].
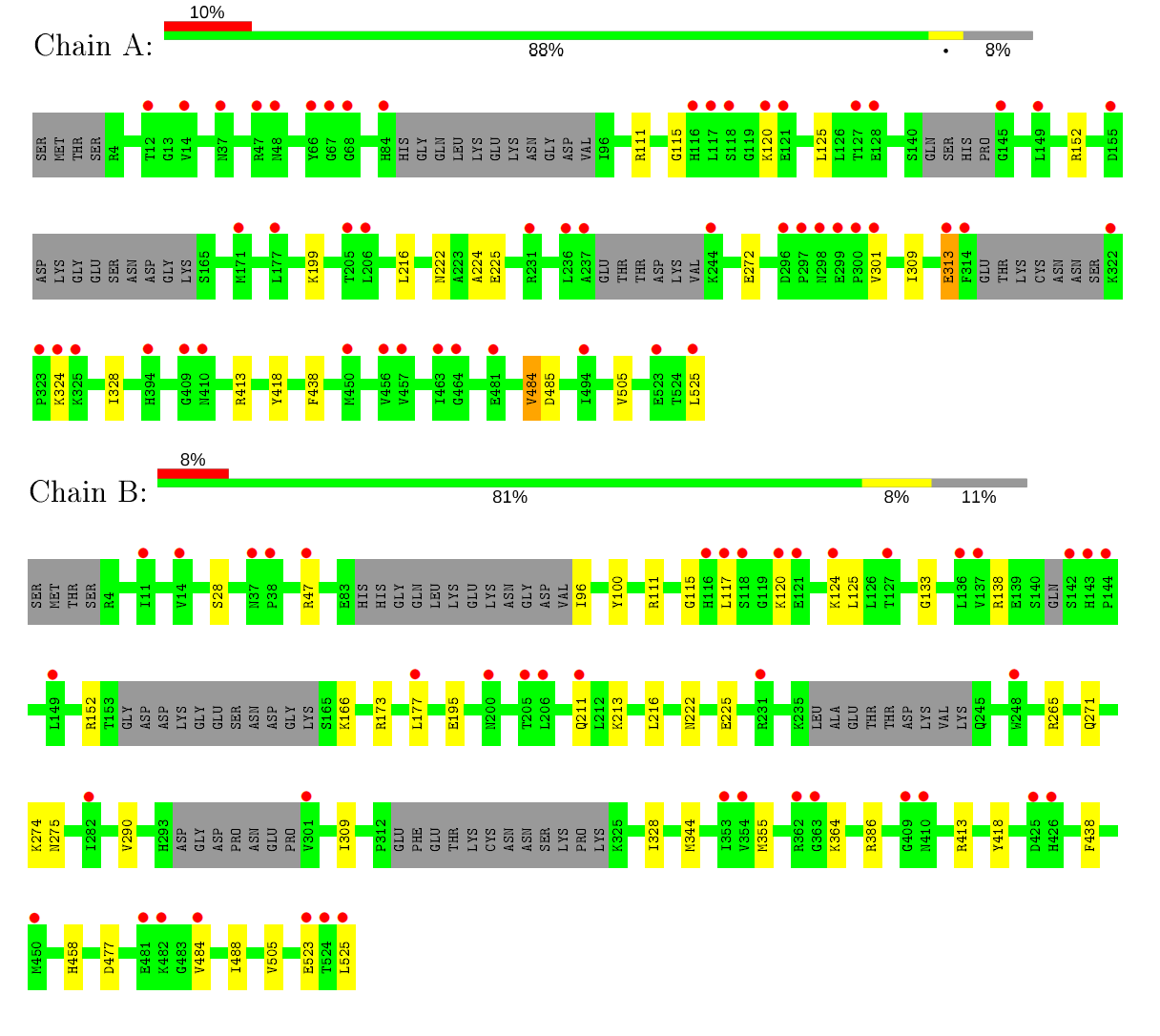
Работа с отчётом по валидации
Последовательность модели совпадает с референсной последовательностью за исключением SER-0, помеченного как тег экспрессии.
Разрешение 1.7 при полноте 97.8 % (данные, предоставленные депозиторами) - восстановленная из эксперимента ЭП модели должна быть довольно высокого качества.
R-фактор 0.195; Rfree 0.221 (данные, предоставленные депозиторами) - соответствие модулей структурных факторов модели экспериментальным довольно хорошее, значение показателя R-free не вызывает подозрения о переоптимизации модели. После пересчёта и R-фактор, и R-free несколько увеличились, но незначительно.
B-фактор Уилсона 22.6, что говорит о приемлемом уровне упорядоченности кристалла.
Clashscore у данной модели = 1. Все клеши, кроме двух, между атомами самого белка, что в целом неприятно. Оба оставшихся на цепи B, один клеш с молекулой аллостерического ингибитора, второй с молекулой воды.
Маргиналов по длине химических связей в структуре не наблюдается.
Маргиналов по углам химических связей в структуре не наблюдается.
Маргиналы по картам Рамачандрана: 4 остатка из 476 проанализированных на цепи А (всего 526 в цепи), 2 остатка из 458 проанализированных на цепи В (всего 526 в цепи). 0.6% маргиналов по картам Рамачандрана для структуры такого разрешения - довольно высокий результат!
Маргиналы по ротамерам боковых радикалов остатков: 10 остатков из 434 проанализированных на цепи А (всего возможно 468 в цепи), 19 остатков из 421 проанализированных на цепи В (всего возможно 468 в цепи). Здесь остатков описывается меньше, так как можно оценить только остатки, имеющие степени свободы по боковым цепям. 3% маргиналов по ротамерам для структуры такого разрешения - также довольно высокий результат.
Маргиналы по RSRZ (Z-score по RSR больше 2): 51 остаток из 485 проанализированных на цепи А (всего 526 в цепи), 42 остатка из 470 проанализированных на цепи В (всего 526 в цепи). По этому показателю маргиналов очень много в сравнении как со структурами в целом, так и со структурами похожего разрешения, то есть в модели много остатков, плохо вписанных в соответствующую им электронную плотность. К сожалению, оценить вклад всех неточно вписанных остатков сложно ввиду их количества, однако большинство из них находятся вне сайта связывания аллостерического ингибитора SHP099.
MolProbity выдал все те же ошибки, которые были указаны в отчёте с PDB. В задании 2 некоторые из остатков будут рассмотрены подробнее.
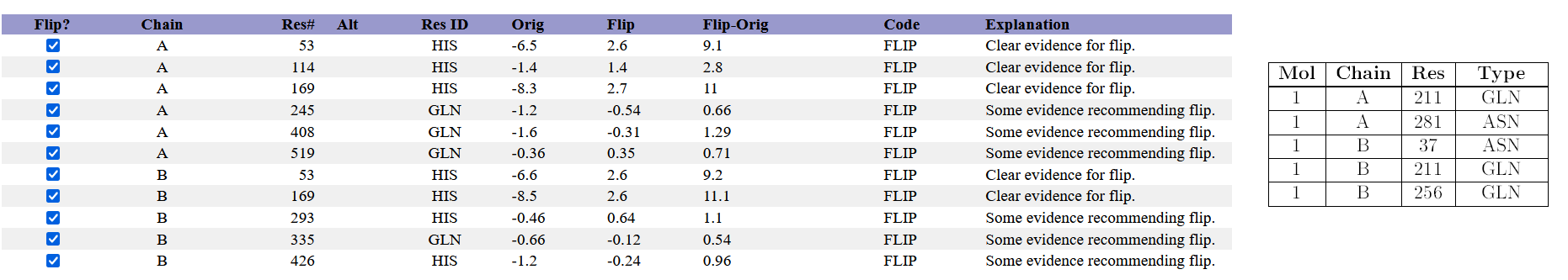
После добавления водородов в MolProbity было инвертировано 5 предложенных остатков (эти остатки также были указаны в отчёте по валидации) для оптимизации водородных связей и уменьшения числа клэшей. Пример такой инверсии будет приведен в задании 2. Визуализация данных из отчёта MyProbity также проводилась в задании 4.
Отчёт MolProbity по структуре 5EHR.
Отчёт MolProbity по структуре 5EHR после добавления водородов и инверсии предложенных остатков.
Карты Рамачандрана из MyProbity.
Задание 2. Исследование маргинальных остатков
В задании 2 рассматриваются и обсуждаются 5 маргинальных остатков из структуры 5EHR, определённые с помощью отчёта о валидации. Здесь и далее на рисунках голубым цветом показаны C-атомы цепи А, а зеленым - цепи В.
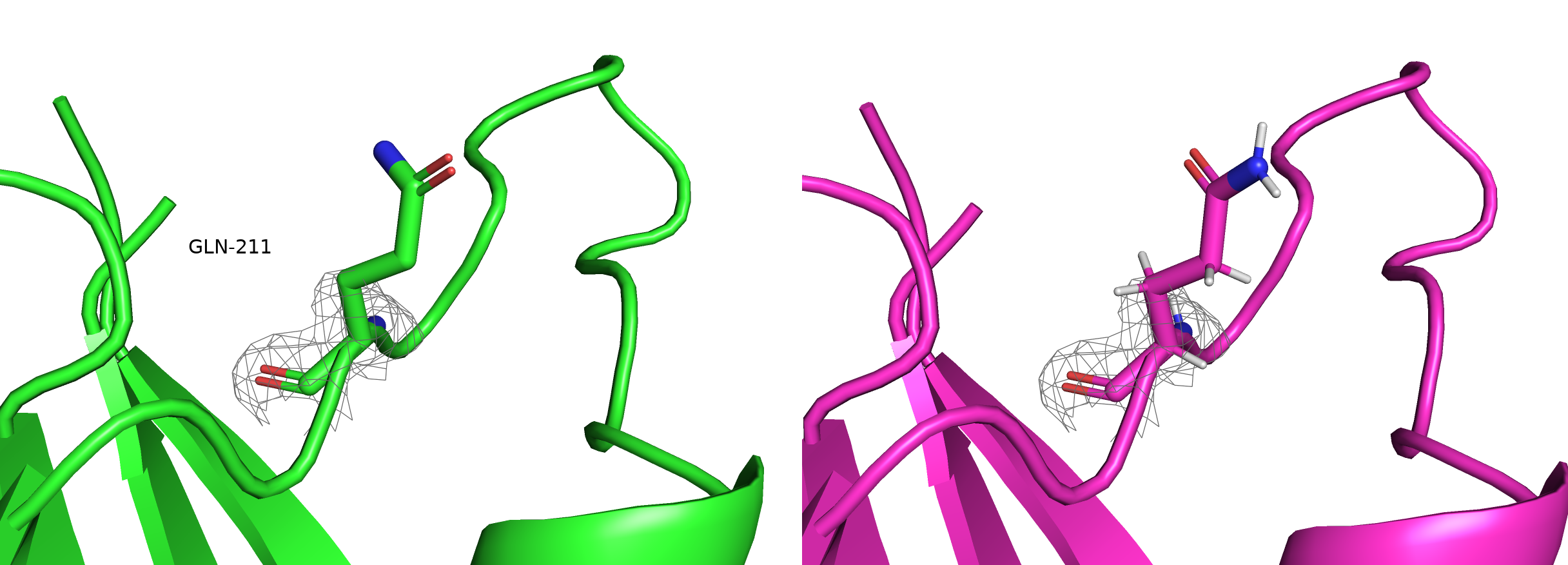
B:211:GLN - данный остаток является маргиналом по ротамерам, RSRZ, а также был целью инверсии MyProbity. Видно, что он действительно плохо вписан в ЭП - для боковой цепи ЭП практически нет, можно сделать инверсию и поправить маргинальность по ротамерам (но смысла в этом особого нет, так как подтверждающая ЭП для остатка отсутствует и работать с ним нежелательно). К счастью, это краевой остаток, который, скорее всего, ни в чём не задействован.
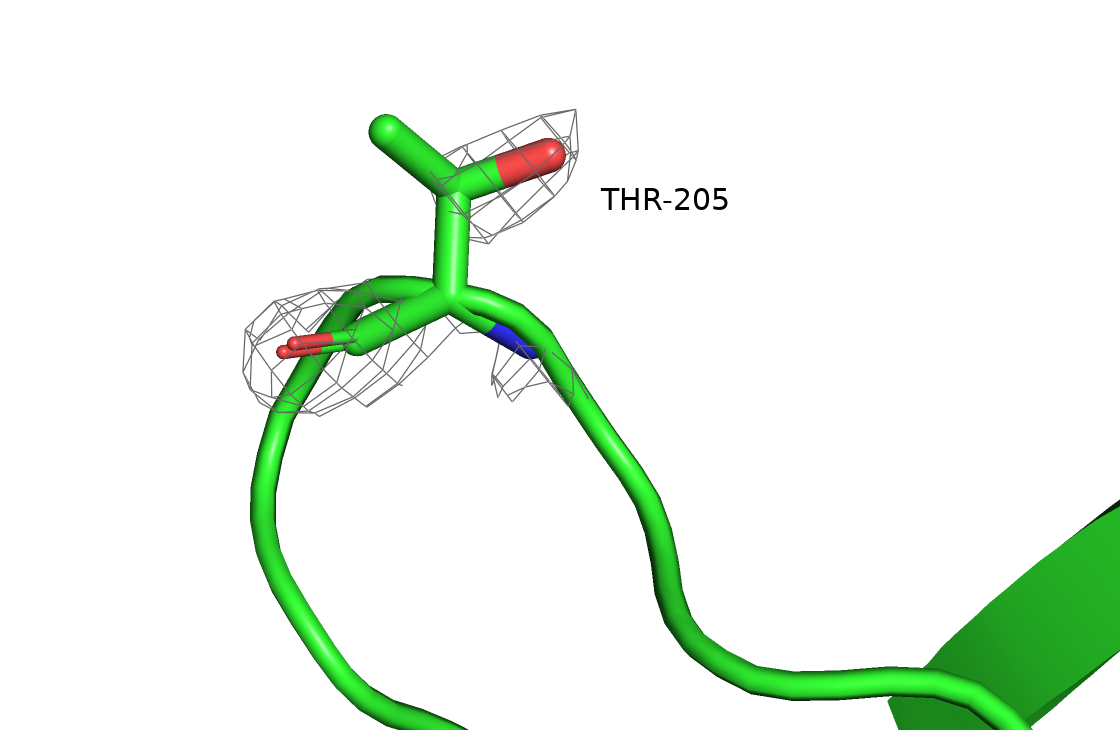
B:205:THR - данный остаток является маргиналом по RSRZ, причём его значение RSRZ (9.6) самое высокое в структуре, несмотря на сравнительно неплохую вписанность в ЭП. Скорее всего, играет роль краевое положение остатка в структуре.
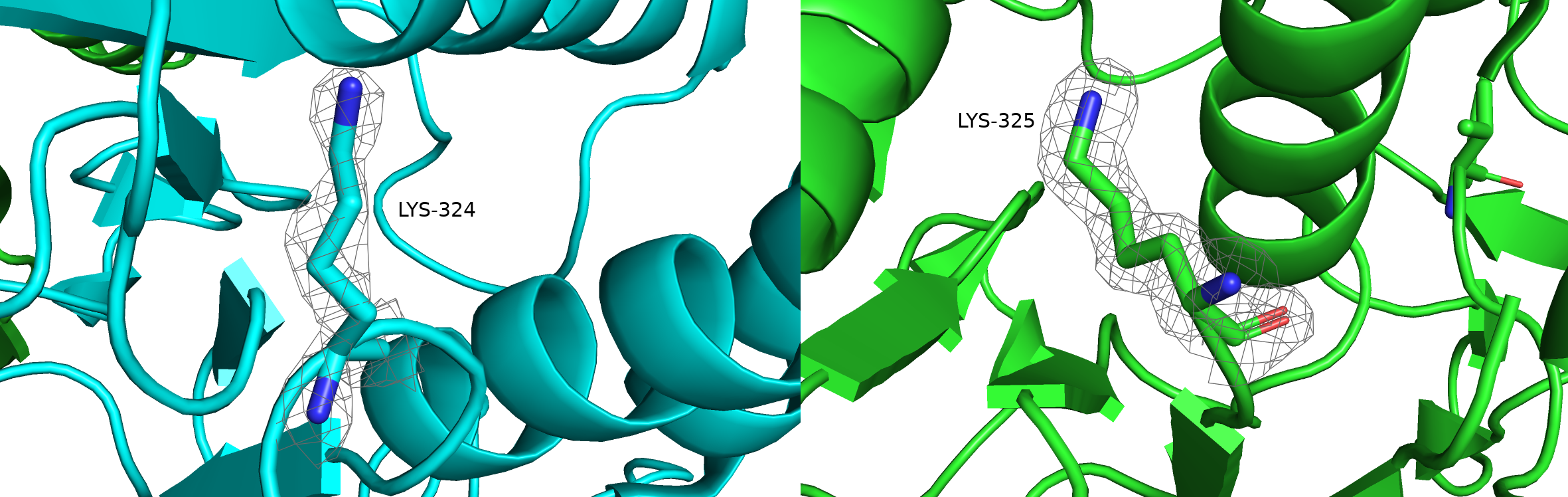
A:324:LYS - данный остаток является маргиналом по картам Рамачандрана. К сожалению, на взгляд определить её причину не удаётся. Покрытие ЭП хорошее, значит проблема, скорее всего, в расшифровке. Интересно заметить, что на цепи B участок белка рядом с соответствующим остатком попросту отсутствует. Этот остаток находится на краю белковой глобулы.
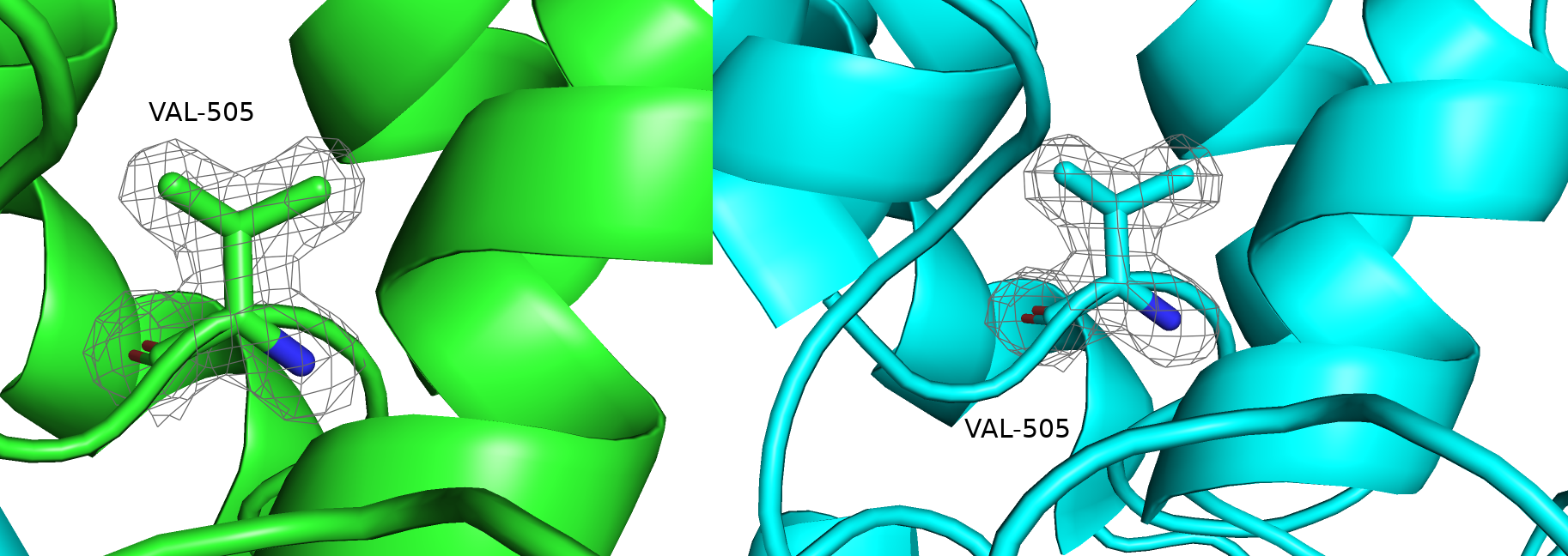
A:505:VAL - данный остаток является маргиналом по картам Рамачандрана, причём это наблюдается в обеих цепях модели (A и B). Этот остаток находится близко к центру белковой глобулы, однако ни с чем не взаимодействует. Покрытие ЭП хорошее, значит проблема, скорее всего, в расшифровке.

A:111:ARG - данный остаток является маргиналом по ротамерам, причём у него существуют 2 альт-локи с равной встречаемостью (0.5). У одной из альт-лок (А) также наблюдается клеш с атомами лиганда. Визуально при отображении сфер Ван-дер-Ваальсовых радиусов я его не заметила, но верю программам, что он есть. Это уже куда более интересный случай, поскольку остаток A:111:ARG напрямую взаимодействует с лигандом. Однако я бы сказала, что в данном случае это не ошибка в структуре, а важный для исследования факт. Такие свойства альт-локи А, как маргинальность по ротамерам и клеш с атомами лиганда, требуют более тщательного изучения как раз в рамках функционального анализа структуры.
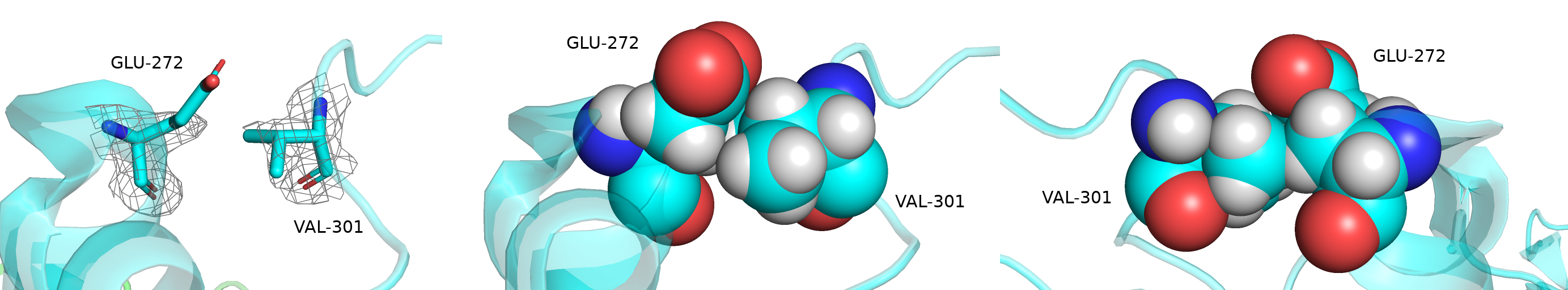
A:272:GLU и A:301:VAL - данные два остатка имеют тройной клеш атомов, сохраняющийся и после работы с MolProbity. На иллюстрации при отображении сфер Ван-дер-Ваальсовых радиусов чётко видно соответствующее перекрывание.
Задание 3. Обсуждение качества структуры для изучения особенностей исследуемого белка
Можно сказать, что структура 5EHR на самом деле имеет сравнительно низкое качество для анализа структуры белка целиком. Однако большая часть неточностей связана с остатками на краю белковой глобулы, не несущими функциональной нагрузки, в то время как сайт связывания аллостерического ингибитора (что и интересовало авторов оригинальной публикации [1]) находится в хорошем состоянии. Поскольку эта структура создавалась для определённой цели - исследовать взаимодействие с аллостерическим ингибитором - отсутствие ЭП для краевых остатков не должно быть проблемой. Для исследования строения белка она однозначно не подходит исходя как минимум из гэпов в аминокислотной последовательности структуры (см. рис. 5). Я бы также добавила, что аллостерические ингибиторы не стоит исследовать с помощью подобных структур без дополнительных данных, так как исследование механизма действия ингибитора, значительно изменяющего конформацию белка, потребует подробного анализа структуры целиком.
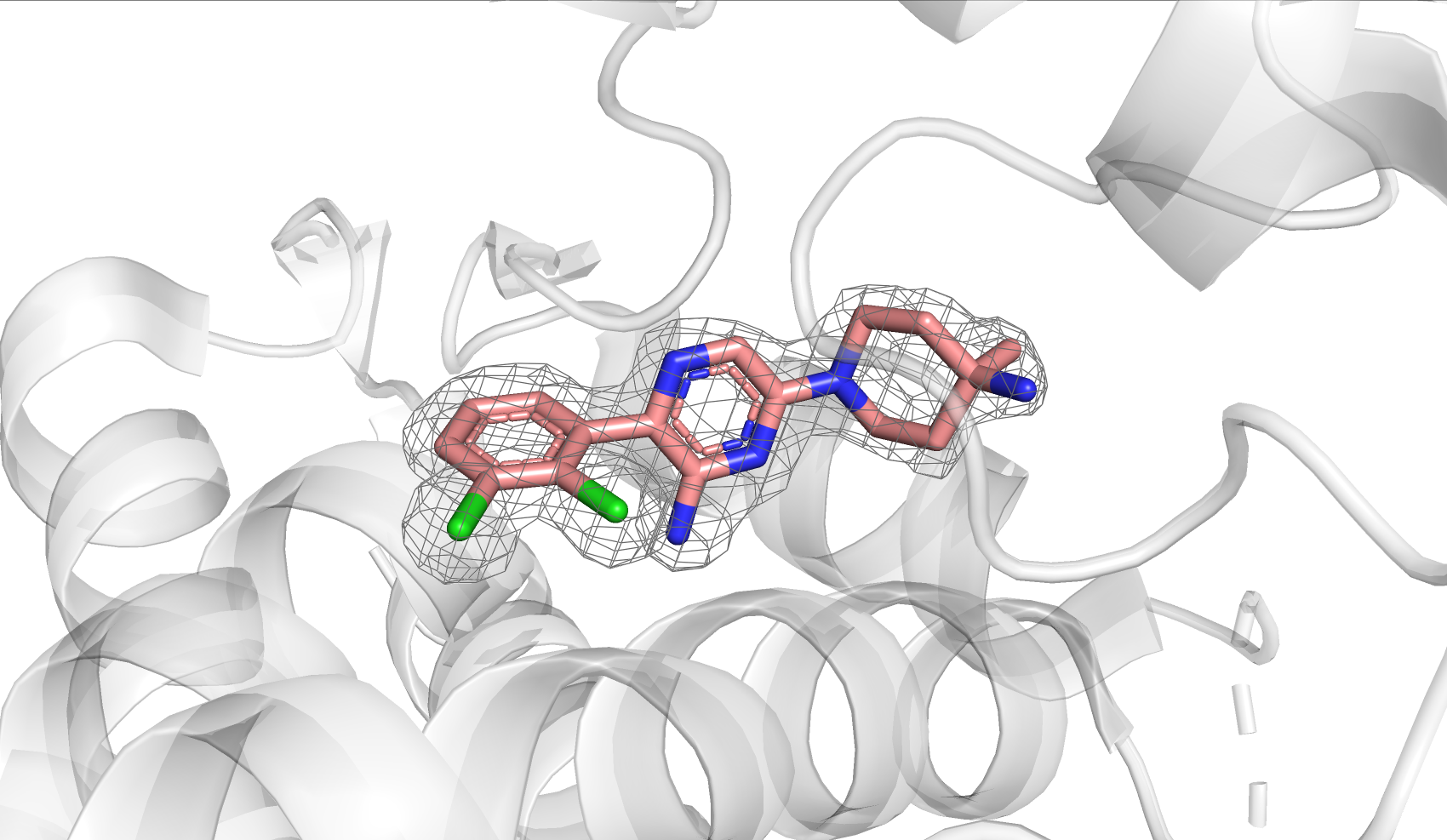
В структуре нет лиганда, но есть молекула аллостерического ингибитора SHP099. У ингибитора полное покрытие ЭП, всё очень чётко. Можно заметить, что электроотрицательные атомы хлора имеют более объёмное отображение ЭП, чем, к примеру, атомы кислорода.
Задание 4. PDB Redo
В задании 4 необходимо провести сравнение PDB-структуры 5EHR и аналогичной переделанной моделью из базы PDB Redo.
Отчёт об оценке качества структуры с сайта PDB Redo по поводу исправленной структуры говорит следующее: сильно улучшилось состояние маргиналов по ротамерам, несколько улучшилась ситуация с водородными связями и маргиналами по картам Рамачандрана.
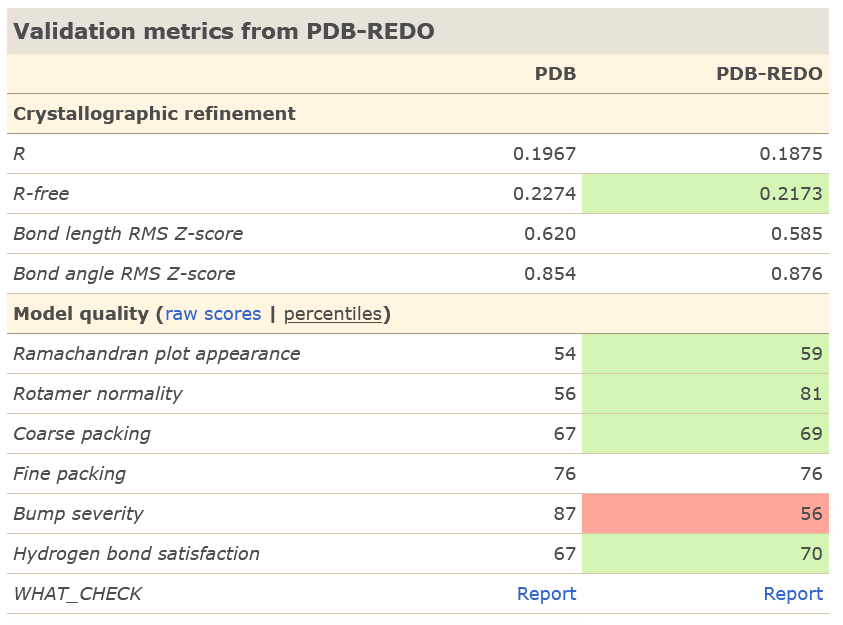
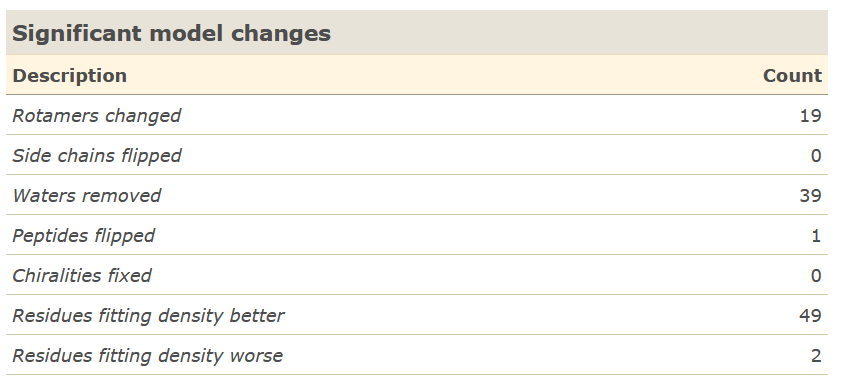
Согласно списку изменений в структуре (19 изменений ротамеров, удаление 39 молекул воды, переворот одного пептида) по данным PDB Redo, 49 остатков стали лучше соответствовать электронной плотности, а два - хуже.
Для сравнения по показателям я загрузила исправленную стуктуру в MolProbity. MolProbity предложил развернуть больше остатков, чем для оригинальной структуры.
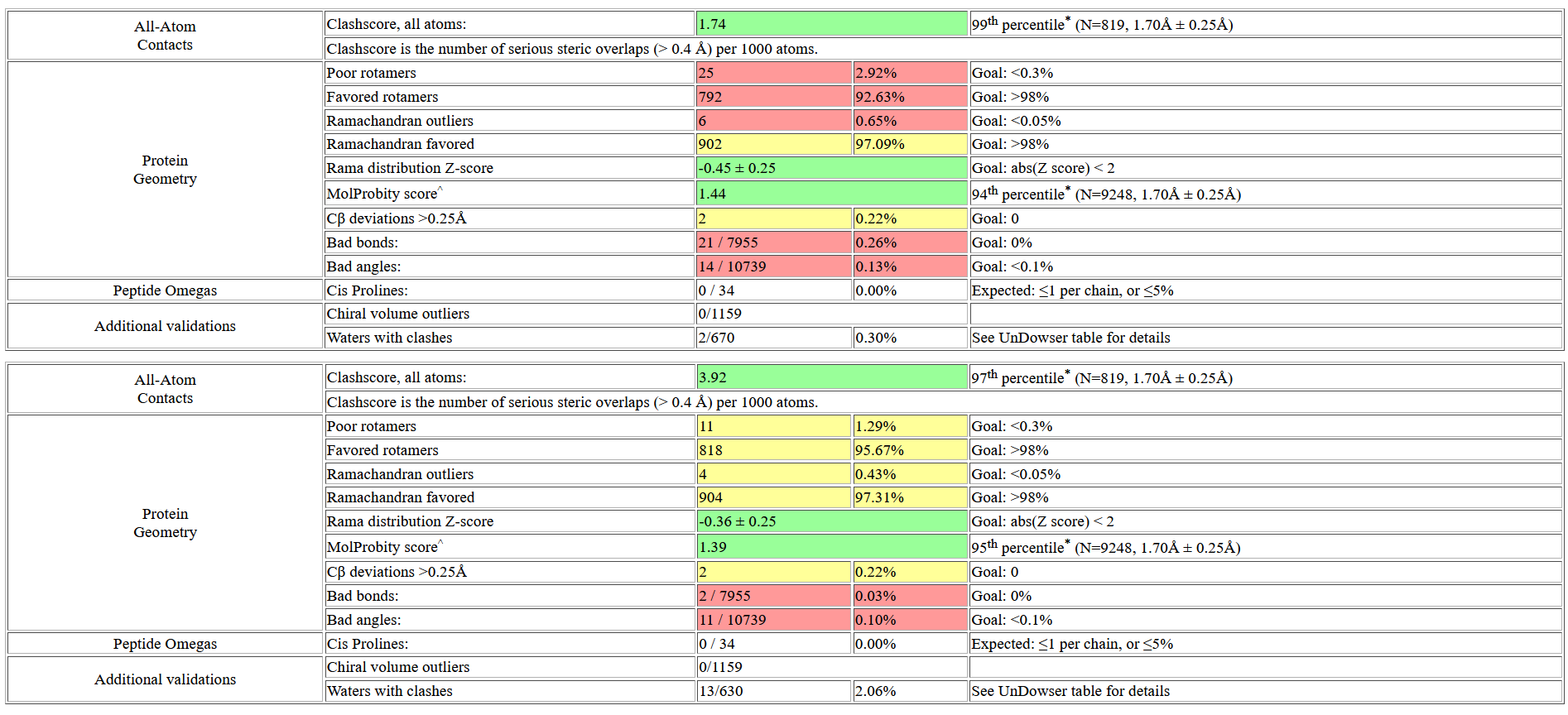
При сравнении характеристик из MolProbity для оригинальной структуры и структуры PDB Redo после модификаций MolProbity можно заметить, что в два раза увеличился клэш-скор, то есть после всех модификаций наблюдается в два раза больше клэшей на 1000 атомов. Все остальные показатели улучшились, так что общий MolProbity score вырос. Если судить по числовым показателям, модель из PDB Redo действительно выше по качеству.
Посмотрим на некоторые остатки, состояние которых улучшилось или ухудшилось в PDB Red мoдели 5EHR.
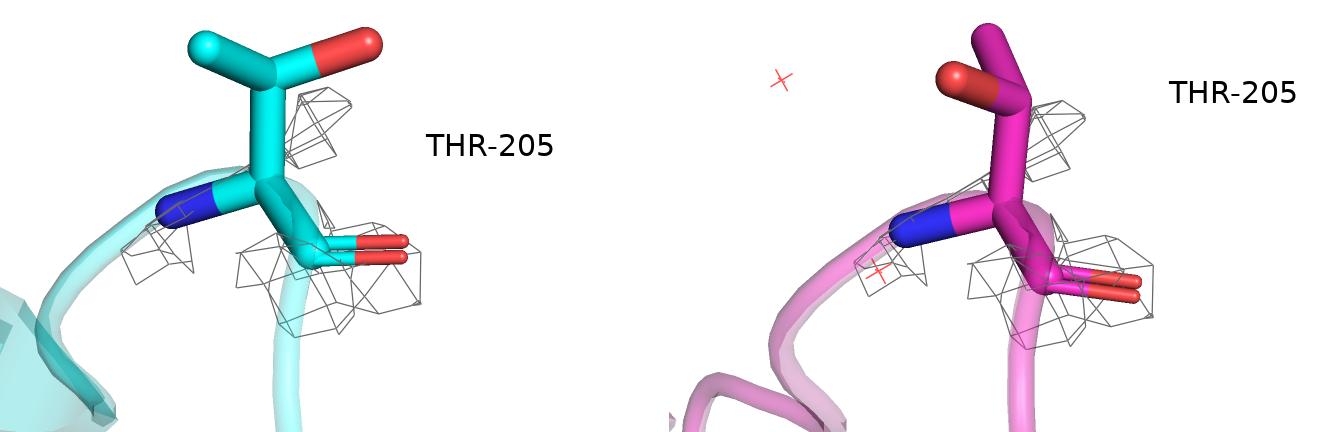
A:205:THR - данный остаток являлся RSRZ маргиналом. Не могу сказать, что новый вариант визуально лучше вписывается в плотность, но по расчётам это так.
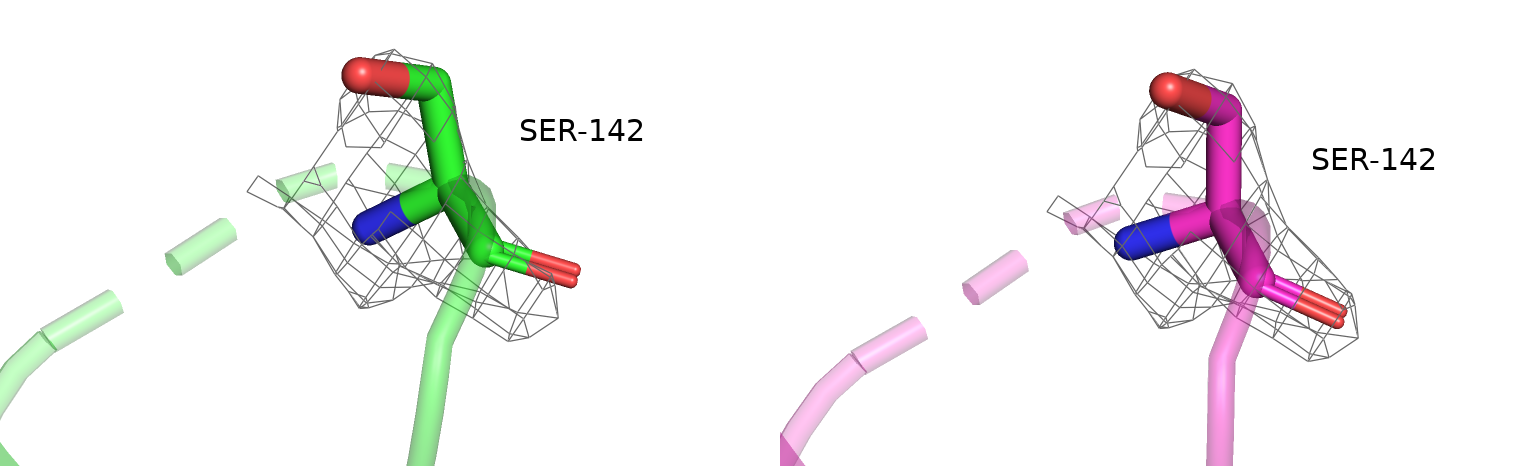
B:142:SER - данный остаток являлся RSRZ маргиналом. В PDB Redo модели он, как и предыдущий, намного лучше вписан в соответствующую ЭП по расчётам, хотя визуально ничего не изменилось.
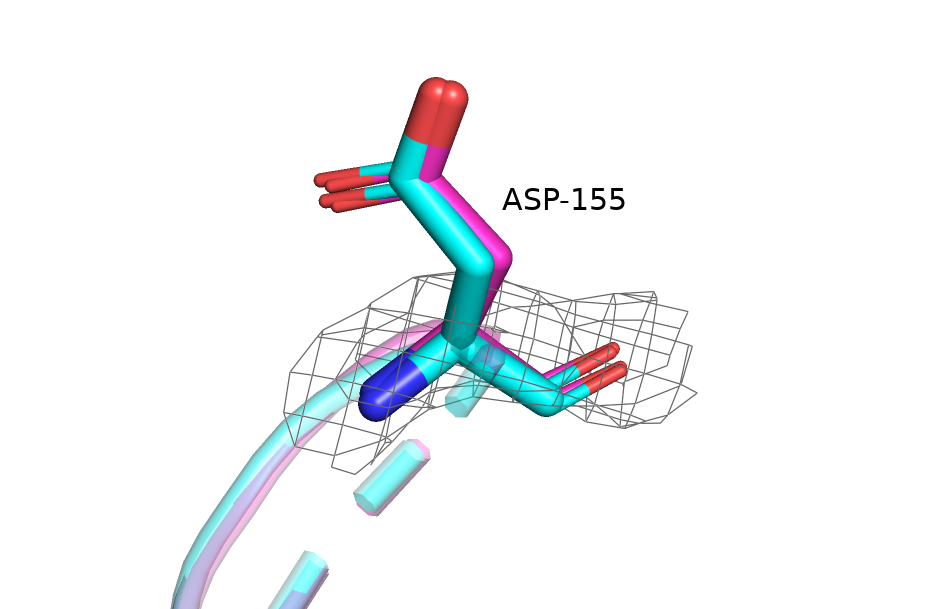
A:155:ASP - данный остаток являлся маргиналом по RSRZ, и остался им после изменений в модели, но качество его вписания в электронную плотность ухудшилось. Как мне кажется, здесь тоже ничего не изменилось - качество вписания боковой цепи в "отсутствующую" (при взятом уровне подрезки) электронную плотность вряд ли может стать сильно хуже, а остов и так вписывается.
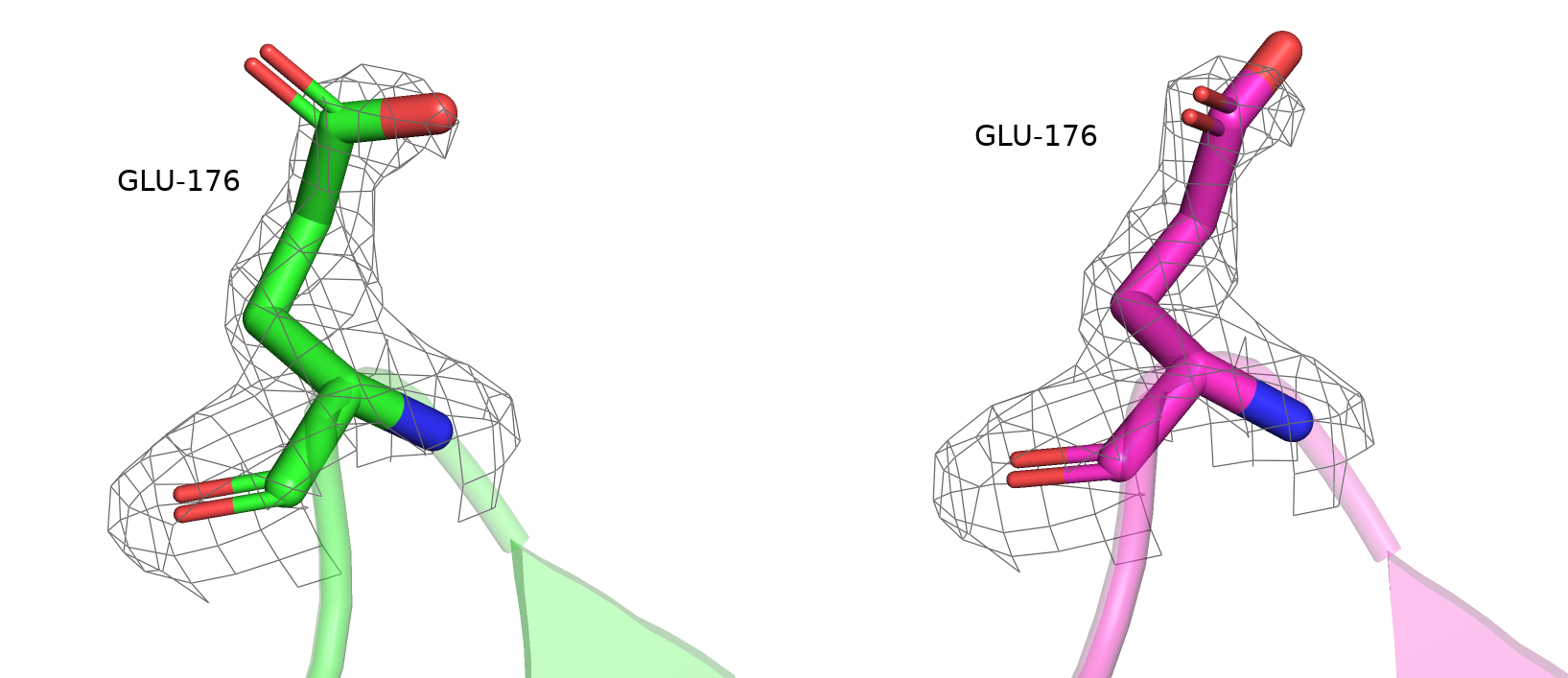
B:176:GLU - данный остаток не являлся маргиналом в исходной структуре, но стал маргиналом по RSRZ в исправленной. Он действительно стал хуже вписываться в электронную плотность. Не уверена, почему в исправленной структуре именно такой вариант, какого-то смысла в новом положении не заметно.
Как вывод из анализа исправленной структуры можно сказать, что многие мелкие недочёты были исправлены и качество структуры в целом возросло. Сайт связывания не изменился, а на периферии белковой глобулы всё так же присутствуют гэпы, так что эта модель может использоваться только для тех же целей, что и исходная. Основная часть исправлений относилась к краевым остаткам с отсутствующей ЭП, на которых в любом случае не стоит фокусироваться, но всё же общее качество модели в PDB Redo выросло.