Работа с Python в структурной биоинформатике
Задание 1. Работа с разметкой вторичной структуры в ручном режиме
В задании 1 для PDB-структуры 5EHR (ранее рассматриваемой в практикуме 6) небходимо было провести разметку вторичной структуры различными алгоритмами.
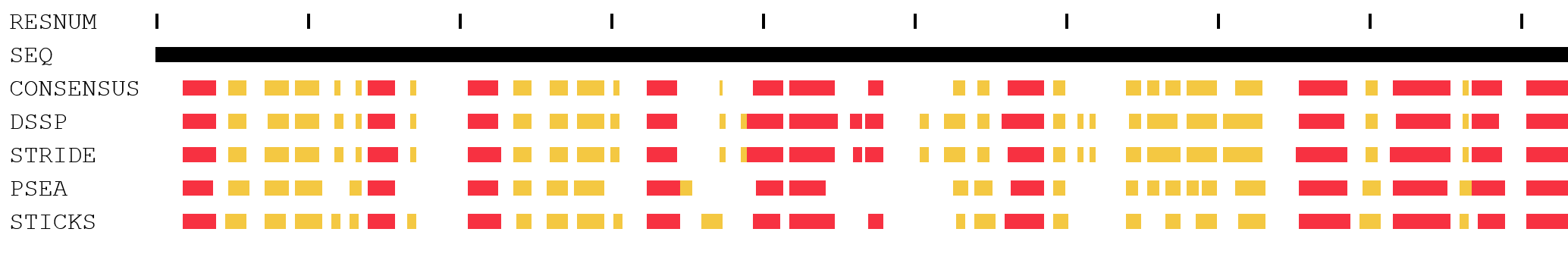
Поскольку мне было бы довольно сложно оценить и рассмотреть различия между, к примеру, поворотом и изломом, я в основном использовала упрощённую репрезентацию (H, E, O), обращаясь к полной репрезентации для оценки отдельных элементов. В упрощённой репрезентации большая часть противоречий между методами DSSP и STRIDE это принадлежность или же не-принадлежность единичных аминокислот к прилегающим элементам вторичной структуры. Противоречие в отсутствии или наличии вторичной структуры наблюдается на единственном участке (рис. 3-6 и обсуждение ниже), и ещё в одном случае есть различие в типе определяемой альфа-спирали (рис. 7-9 и обсуждение ниже). При добавлении в сравнение алгоритмов PSEA и STICKS, помимо противоречий, связанных с краевыми аминокислотами, и указанных выше, добавляются дополнительные (рис. 10-13 и обсуждение ниже). Различия между цепями A и B наблюдаются на четырёх участках (для последнего довольно незначительно) однако в целом вторичная структура очень похожа. Стоит заметить, что в цепи B наблюдаются гэпы (отсутствие отдельных аминокислот), что и обеспечивает различия, к примеру, на первом из участков.
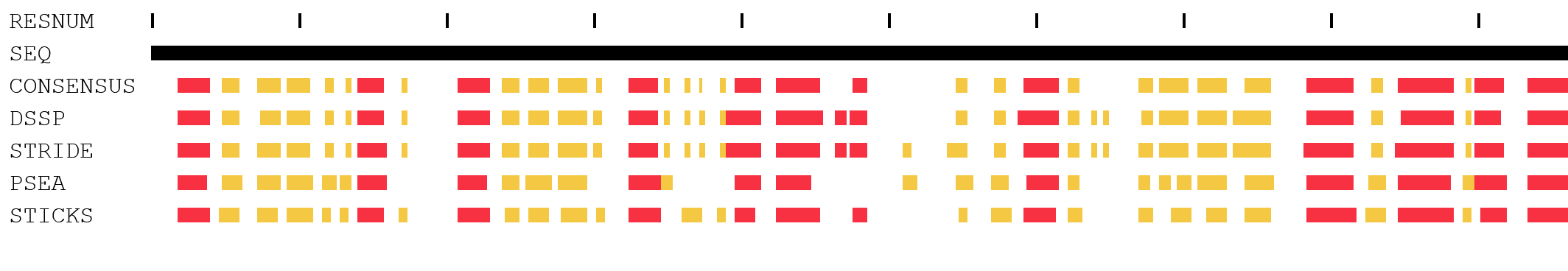
Скачать полную картинку результатов 2struc для цепи A.
Скачать полную картинку результатов 2struc для цепи B.
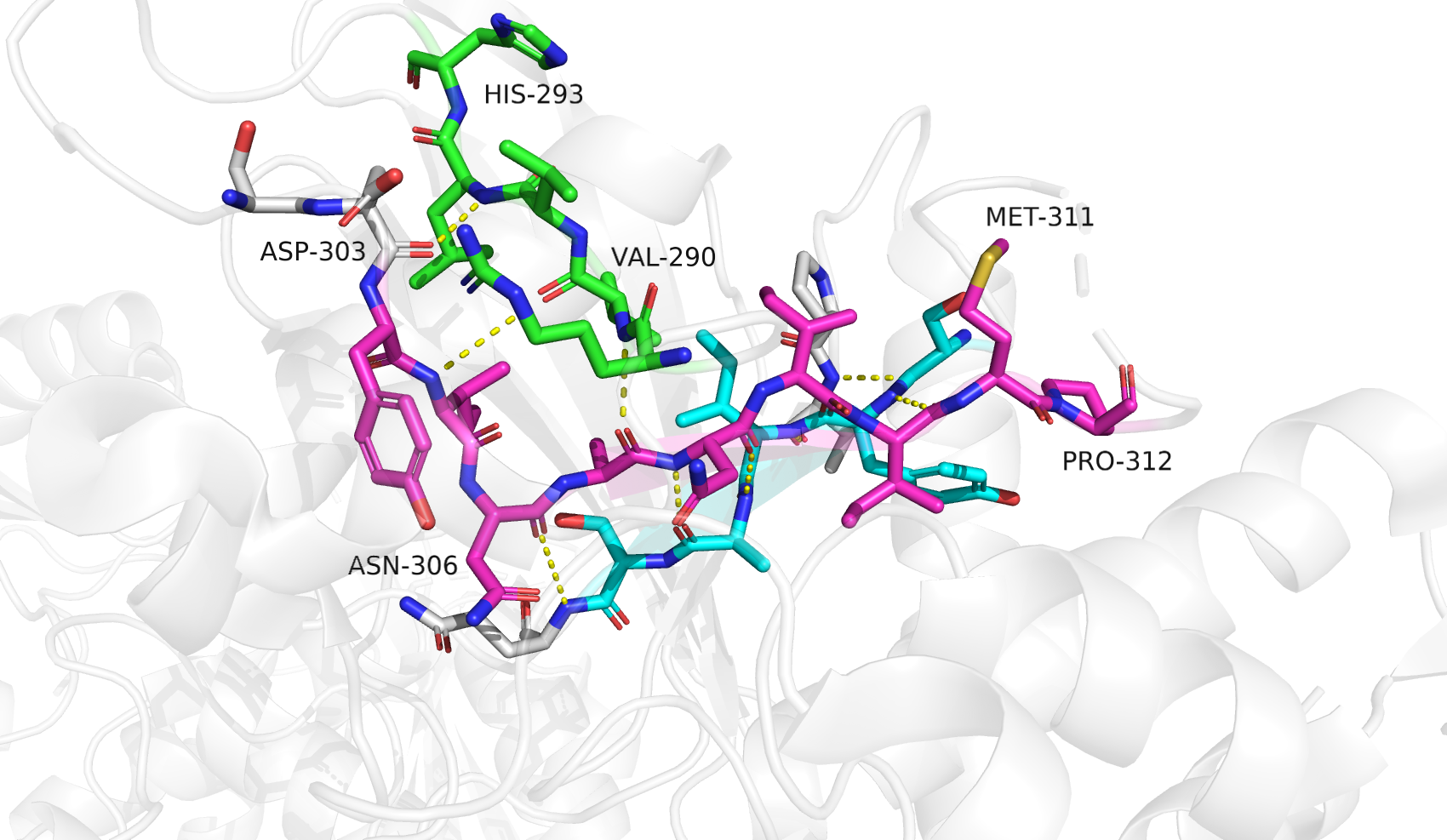
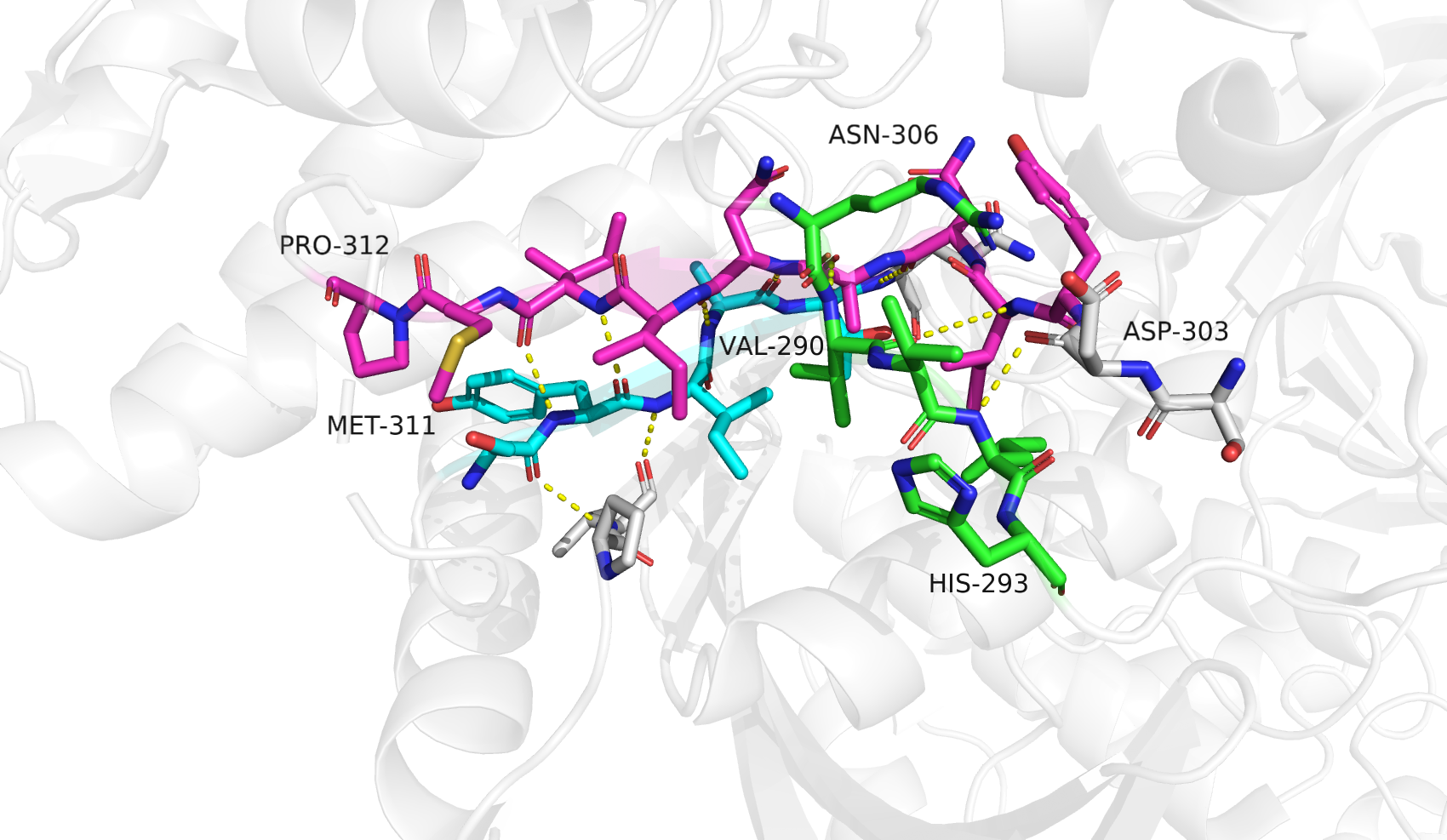
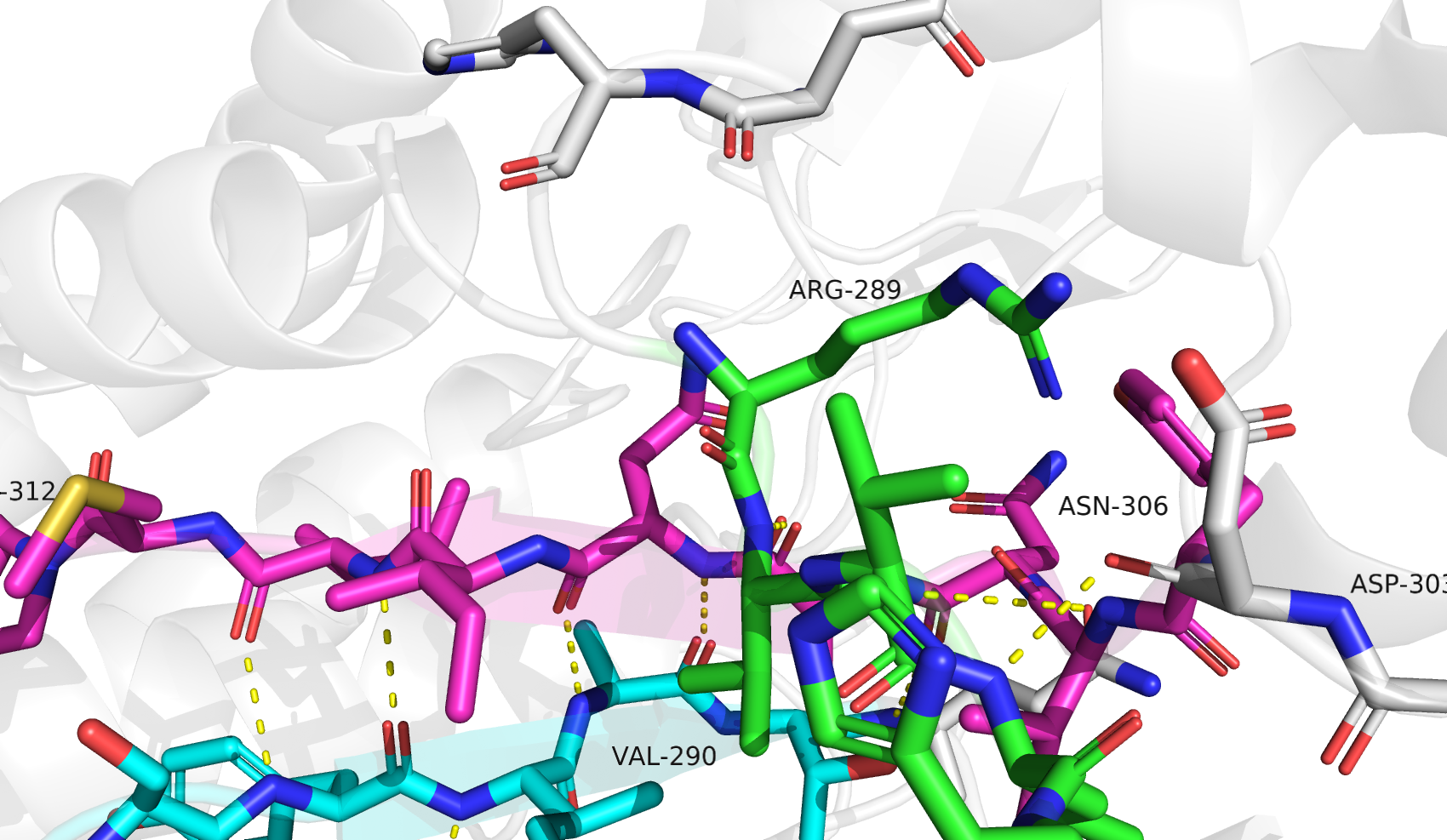
Первый участок (289-312) объединяет в себе несколько противоречий из-за сложности устройства. ASN-306 и VAL-290 представляют собой угловые аминокислоты, "ломающие" структуру соответствующих бета-тяжей, чтобы цепь аминокислот пошла в новом направлении. Я не согласна полностью с разметкой данного участка ни одной из программ. С моей точки зрения, к бета-тяжам относятся аминокислоты под номерами: 290-292 и 303-310. STRIDE определяет бета-тяжи как следующие наборы аминокислот: 289-291 и 304-310. PSEA определяет бета-тяжи как следующие наборы аминокислот: 289-293 и 307-312. DSSP определяет один бета-тяж: 307-310, как и STICKS: 308-310. Ближе всего к ручному анализу результат работы алгоритма STRIDE.
Я не вижу у ARG-289 (который отметили как входящий в бета-цепь и STRIDE, и PSEA) соответствующей схемы водородных связей, которая наблюдалась бы у остатка бета-тяжа. Такая же ситуация с MET-311, PRO-312 и HIS-293. Остаток ASP-303 (не отмеченный STRIDE) наоборот входит во вторичную структуру бета-листа через водородную связь с остовом.
Что любопытно, на цепи B никакого противоречия на этом участке между первыми двумя алгоритмами нет - DSSP и STRIDE по цепи B обе показывают результат, аналогичный выдаче STRIDE на цепи A, а вот PSEA уже не находит первый из бета-тяжей (289-293), который находил при работе с цепью А. Стоит повторно упомянуть, что в цепи B наблюдаются гэпы, в частности, здесь часть участка между бета-тяжами отсутствует. Возможно, это и объясняет такое отличие. .

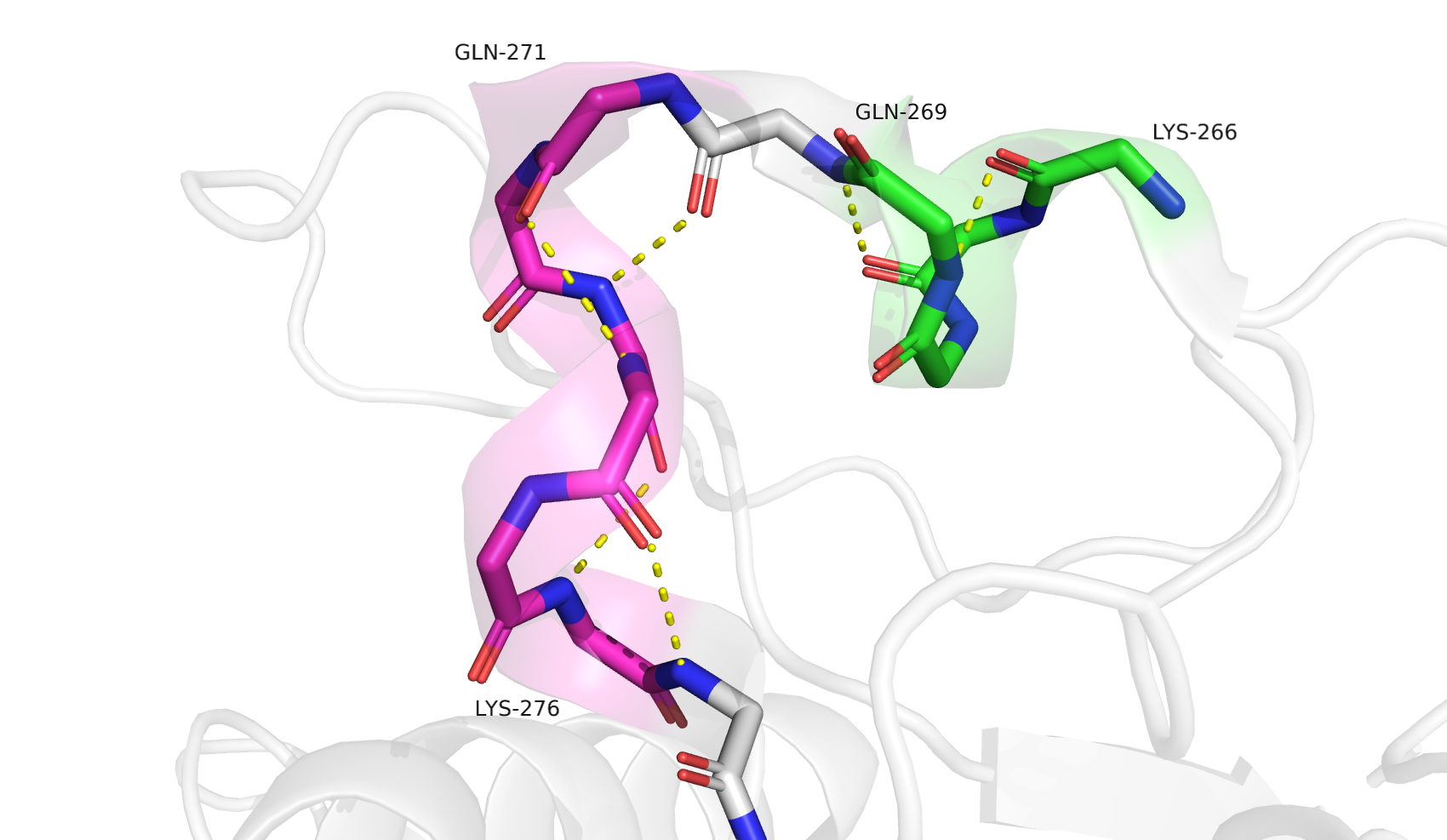
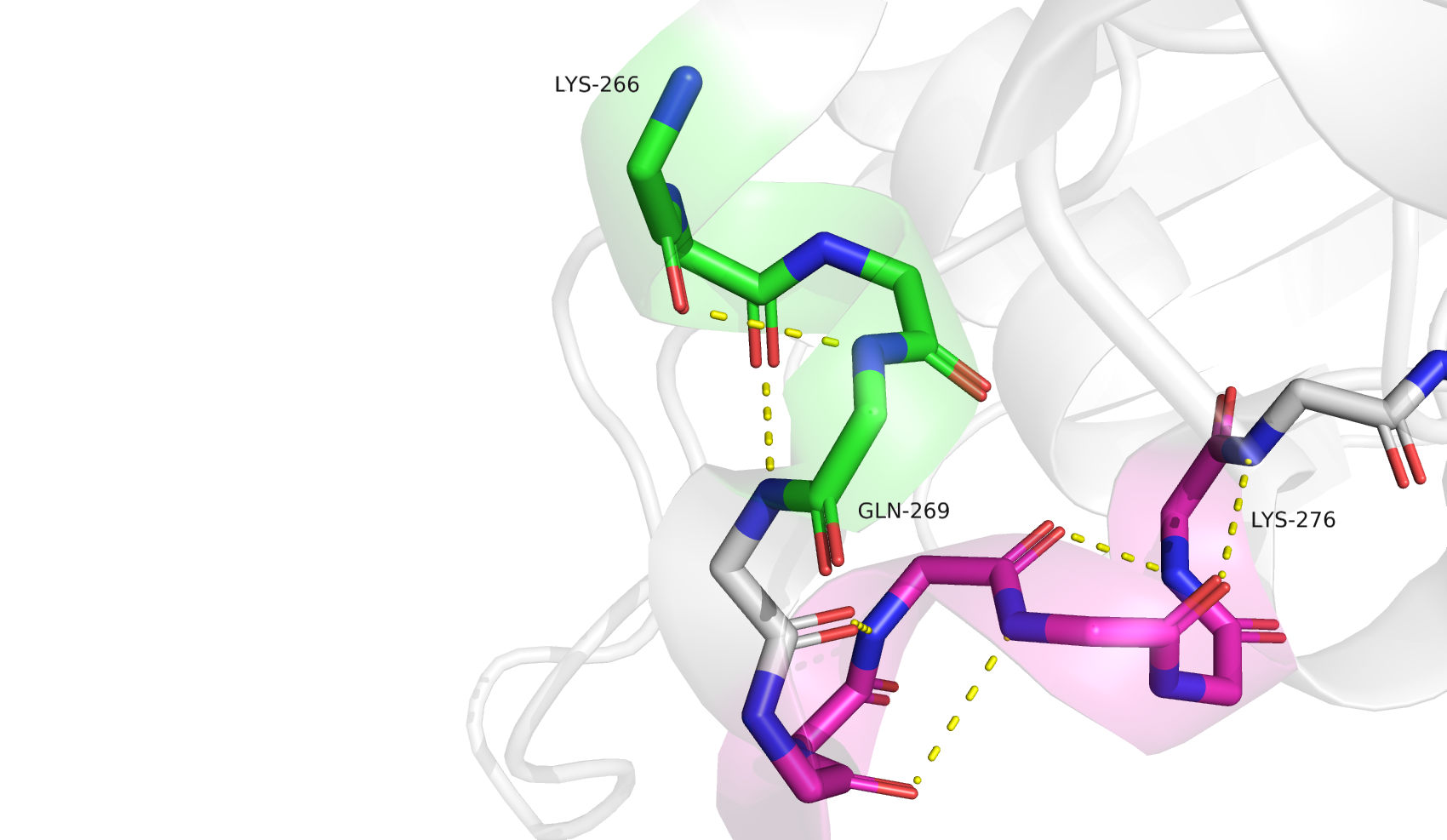
Второй участок противоречия между программами DSSP и STRIDE - несогласие в том, какая альфа-спираль образуется на участке 266-269. DSSP считает, что это классическая альфа-спираль, а STRIDE - 3-спираль, аналогично последующему участку (271-276), который обоими программами определяется как 3-спираль. В цепи B всё аналогично, кроме того что STRIDE для B не относит LYS-266 к 3-спирали. PSEA не умеет определять ничего кроме классических альфа-спиралей и бета-листов. На этом участке он ничего не нашёл. STICKS нашёл последующий участок 3-спирали 272-276 (но не включил в него GLN-271), но ничего не обнаружил на участке 266-269.
При рассмотре участка 266-269 можно видеть, что это действительно 3-спираль. GLN-271 также входит в 3-спираль 271-276. В целом, здесь я полностью соглашусь с разметкой STRIDE.

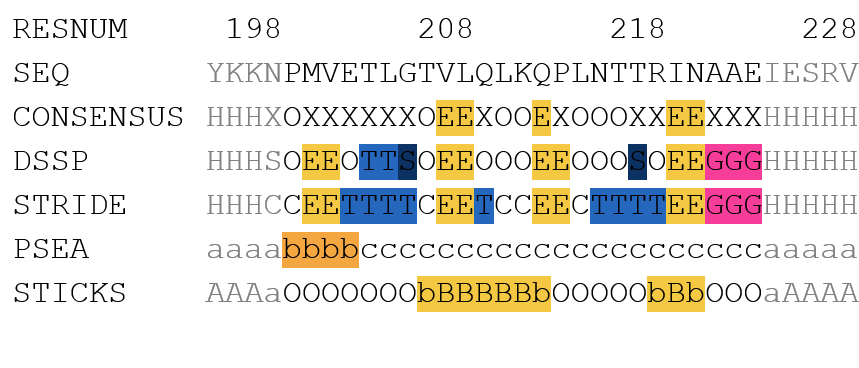
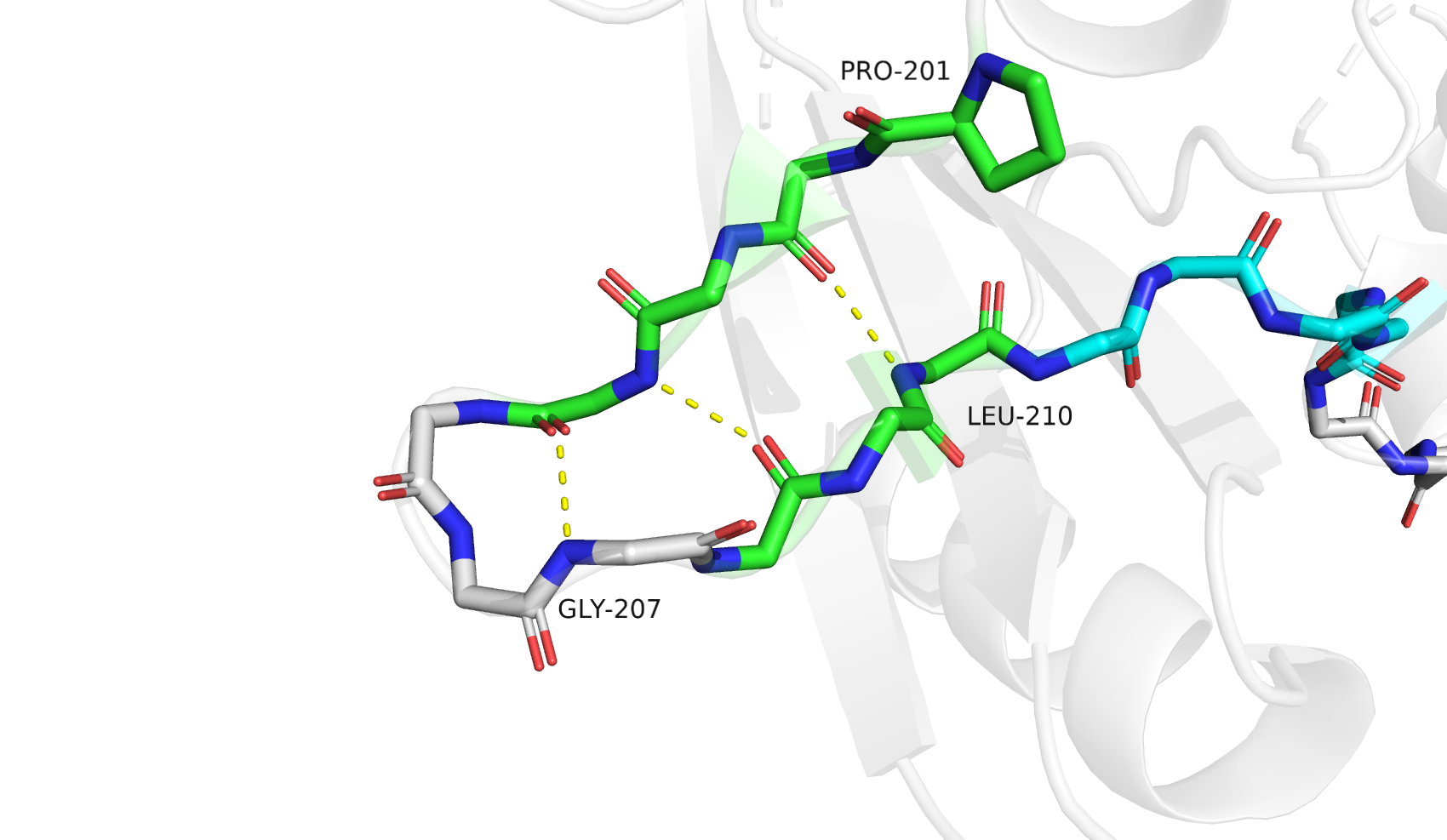
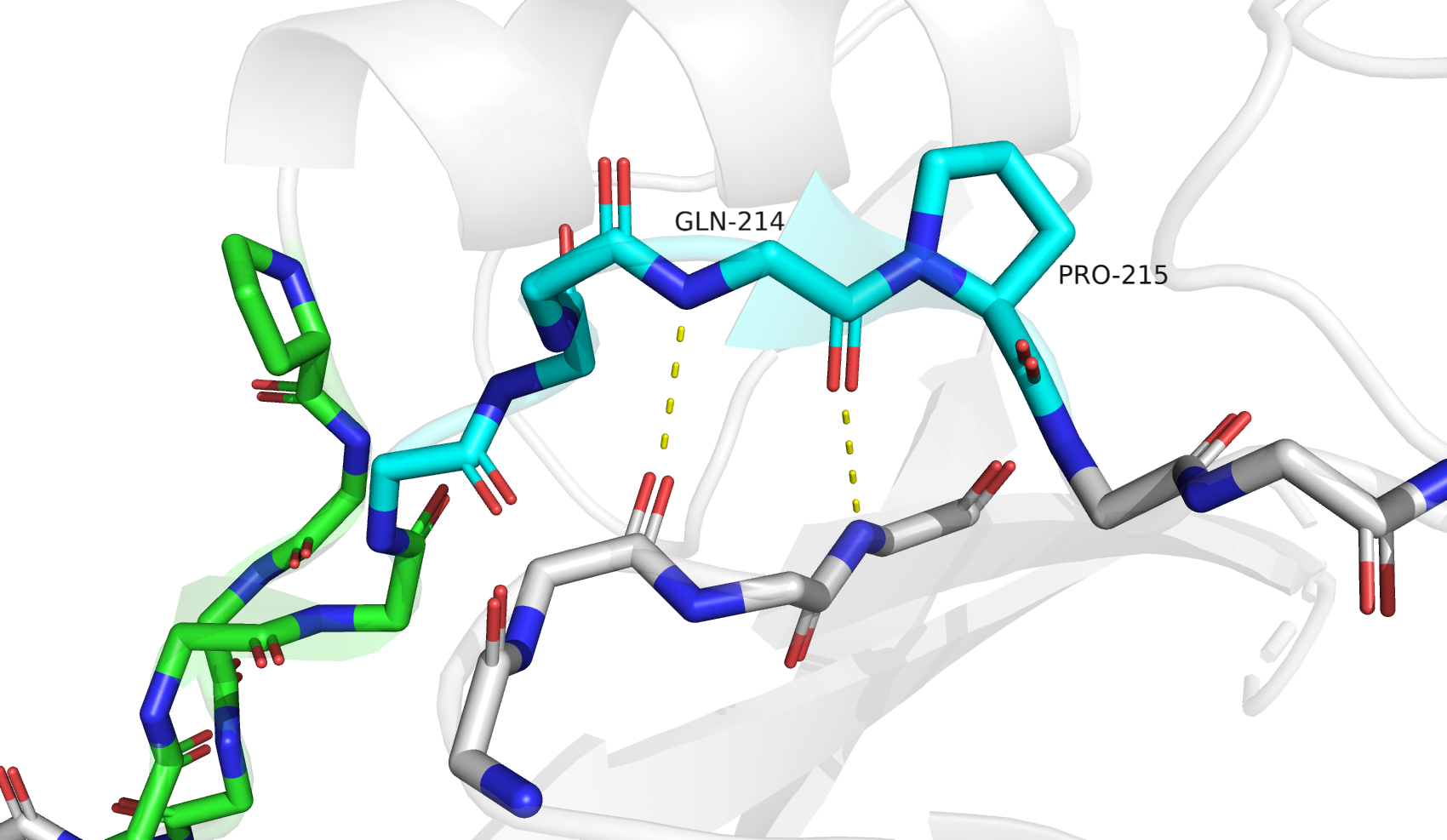
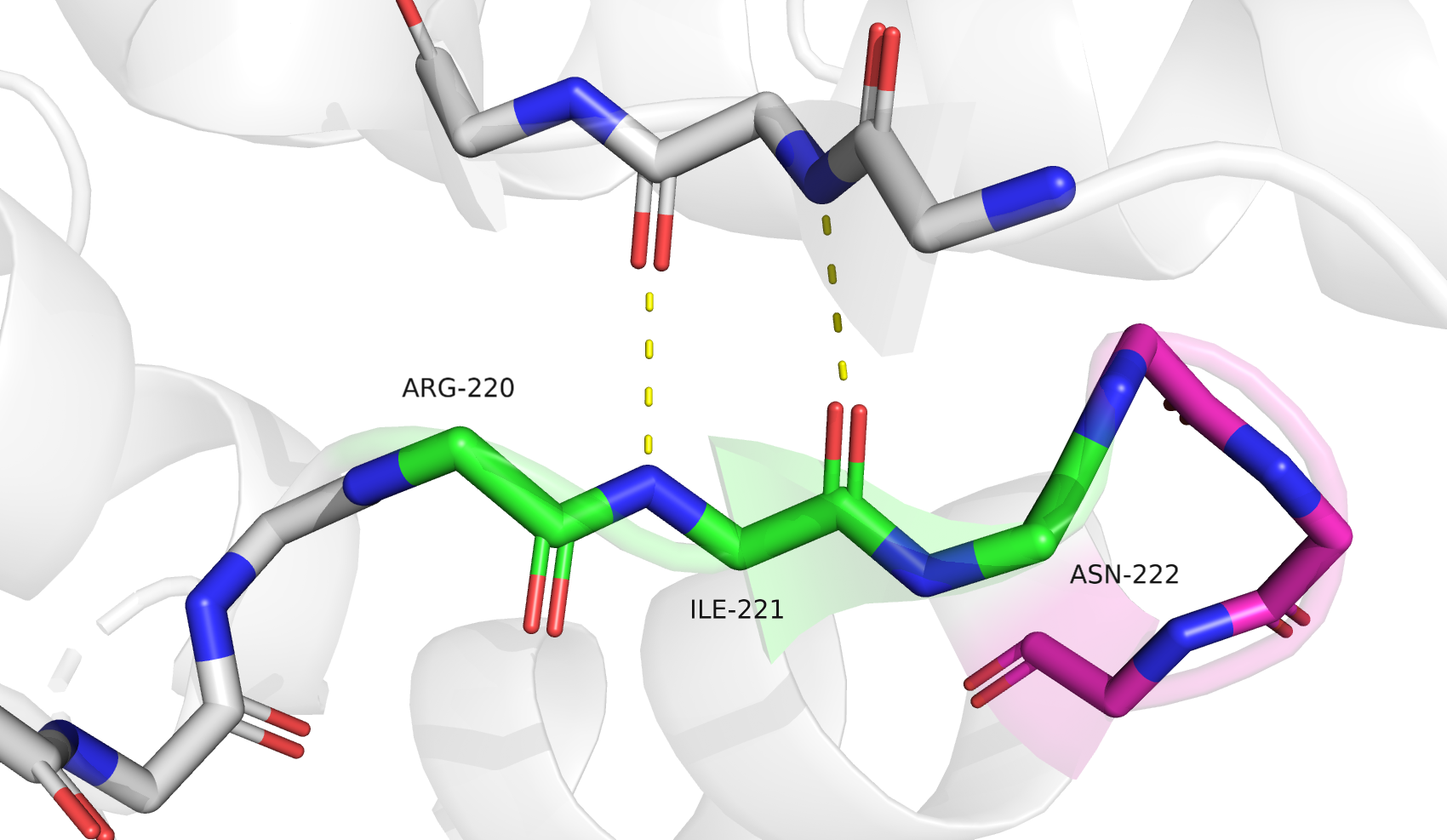
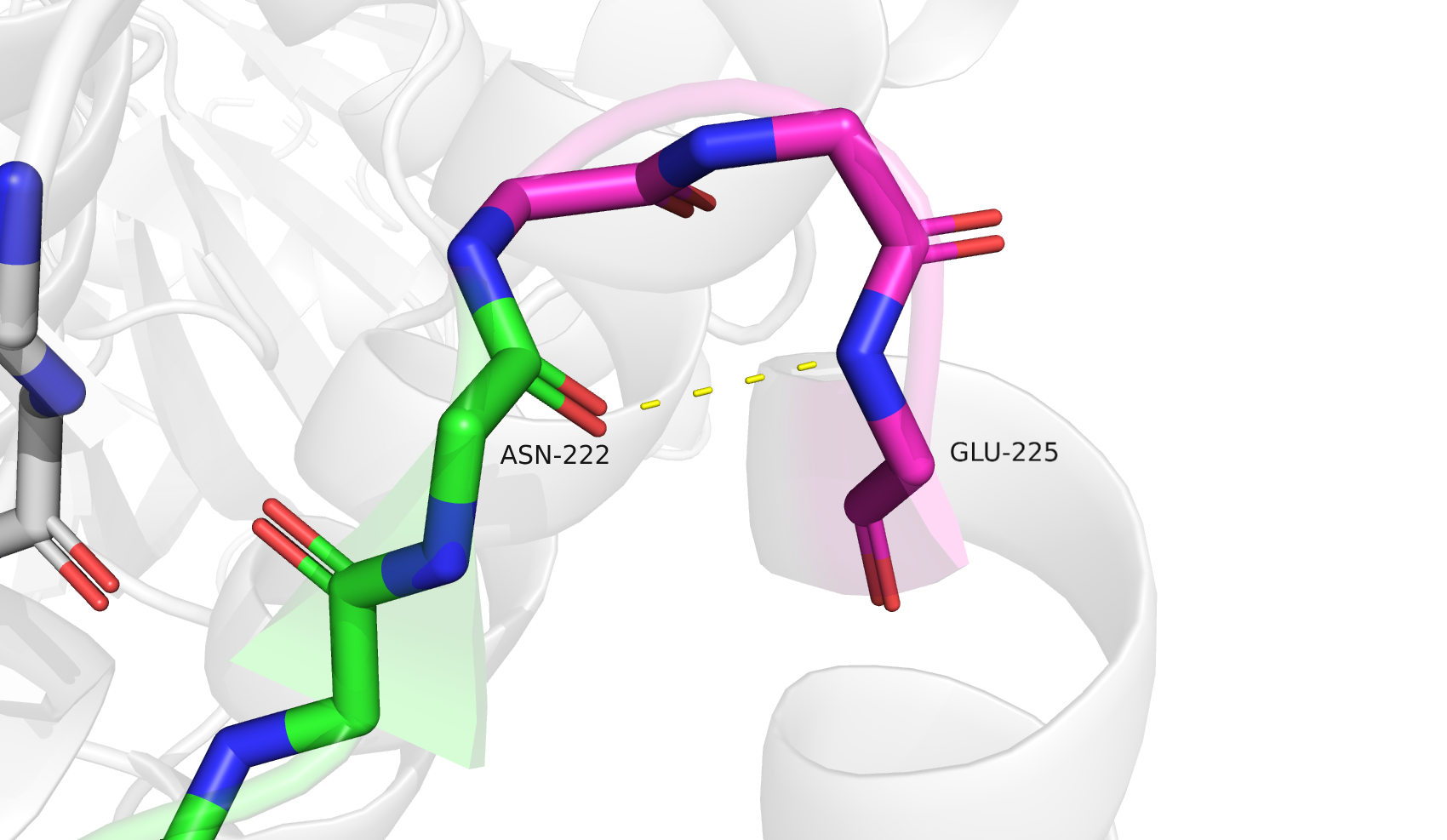
Для третьего участка я решила уделить больше внимания другим доступным алгоритмам - PSEA и STICKS. Исследуемый участок: 201-225, находится между двух альфа-спиралей и близко к сайту связывания лиганда. DSSP и STRIDE сходятся в разметке основной вторичной структуры - участки структуры бета-листа: 202-203, 209-210, 214-215, 221-222; и 3-спираль: 223-225, переходящая в последующую классическую альфа-спираль. PSEA на этом участке находит только один бета-тяж: 201-204; а STICKS два бета-тяжа: 208-214 и 220-222.
Посмотрев на стуктуру, я бы разметила её следующим образом - бета-тяжи: 202-204, 207-210, 214-215, 220-221; 3-спираль 222-225. Такая разметка не совпадает с выдачами программ, но выдача STRIDE довольно похожа. Любопытно, что из-за особенностей работы алгоритмов PSEA нашёл только первый бета-тяж, а STICKS все кроме него (слив второй и третий тяжи в единый).
Хотелось бы также сказать немного об особенностях работы алгоритмов. DSSP работает по принципу поиска водородных связей, которые затем объединяются в паттерны вторичной структуры. STRIDE же смотрит на отдельные остатки и определяет вероятность того, что они входят в состав какой-либо вторичной структуры, что позволяет лучше работать с короткими участками. P-SEA рассчитывает вторичную структуру, основываясь только на положении C-альфа атомов по определённым схемам, что объясняет его более низкую чувствительность и невозможность работы с неклассическими вторичными структурами. STICKS работает похоже на STRIDE, но смотрит не на остатки, а на отдельные линейные участки, для которых уже и определяется вероятность и характер вхождения в состав вторичной структуры.
Задание 2. Работа с разметкой вторичной структуры в автоматическом режиме
В задании 2 был проанализирован датасет 30 белков и выявлена склонность (propensity) различных аминокислот образовывать элементы вторичной структуры. Формула расчёта: Pik = (nik/ni)/(Nk/N)
Остатками, самыми склонными к образованию альфа-спиралей оказались глутаминовая кислота, аланин, лизин и глутамин (перечислены в порядке убывания propensity). Можно было бы предположить, что небольшой размер боковой цепи, либо её гибкость при наличии заряда помогает встраиванию в альфа-спираль. К образованию бета-листов склонны валин, тирозин, изолейцин и цистеин. Все эти аминокислоты (как и несколько следующих) гидрофобны, возможно, дело в том, что бета-листы удобны для образования гидрофобного ядра в белке. К образованию петель оказались более всего склонны пролин, глицин, аспарагин и аспарагиновая кислота (между propensity последних двух очень маленькая разница). Ригидная структура пролина упрощает повороты петель, но мешает образованию вторичных структур - вполне логично. Глицин очень маленький и в большей степени нейтрален к окружению, но во вторичной структуре не может образовывать стабилизирующие взаимодействия, кроме как остовом, и очень подвижен, так что он распространён именно в петлях. Аспарагин и аспарагиновая кислота
| Код аминокислоты | Число остатков в датасете | Альфа-спираль | Бета-лист | Петля |
|---|---|---|---|---|
| A | 468 | 1.3692231511380446 | 0.8427758082098917 | 0.8027125094030709 |
| C | 79 | 0.9054527190805133 | 1.547017243952406 | 0.8622264567548357 |
| D | 322 | 0.9811414427510634 | 0.39679987750727297 | 1.237188667997457 |
| E | 312 | 1.4138025095471902 | 0.5519588039684502 | 0.8798964045379816 |
| F | 203 | 0.9396487987830614 | 1.4777374748546719 | 0.8642858123762832 |
| G | 385 | 0.47996833528748417 | 0.5050180259183474 | 1.5440674021319183 |
| H | 118 | 0.782999191041023 | 1.0357149345105092 | 1.1370140871689987 |
| I | 305 | 1.123775676618369 | 1.639238838226767 | 0.6767599610892401 |
| K | 271 | 1.2427700527724082 | 0.47147439320052364 | 1.028250605086696 |
| L | 480 | 1.1673423423423421 | 1.354079581993569 | 0.7525429775653789 |
| M | 76 | 1.2157092703005357 | 1.0233259998871775 | 0.8419428200129954 |
| N | 193 | 0.9265643109761729 | 0.40296775125090933 | 1.2726944449603093 |
| P | 265 | 0.38239717035381426 | 0.3982972355356023 | 1.651294305057746 |
| Q | 221 | 1.2407297063116838 | 0.6786893832477339 | 0.9526697913794688 |
| R | 275 | 1.08379946677819 | 1.1312403780570983 | 0.8932001013721443 |
| S | 304 | 0.8921737386882963 | 0.9867786427483499 | 1.079587970823115 |
| T | 305 | 0.7426691428086615 | 1.1656809516279232 | 1.1166539357972463 |
| V | 350 | 0.8600722911361209 | 1.9998713826366556 | 0.725389998293224 |
| W | 72 | 0.9520877260238961 | 1.3887995712754555 | 0.8887174211248285 |
| Y | 179 | 1.0822857803720753 | 1.6448352464267966 | 0.7034144735232017 |