Данные в виде файлов с хроматограммами формата [ab1] были получены из капиллярного секвенатора по Сэнгеру. Для просмотра и редактирования автоматического прочтения этих хроматограмм использовалась программа Chromas.
Файлы данного задания:
В следующей таблице приведены нечитаемые участки в соответствии с рекомендацией программы Chromas. Для обратной последовательности — после замены на комплеменатрную.
| Нечитаемый участок | 5'-конец | 3'-конец |
| Прямая цепь | 1-98 (98 нуклеотидов) | 674-715 (45 нуклеотидов) |
| Обратная цепь | 1-42 (42 нуклеотидов) | 576-711 (136 нуклеотид) |
После работы с обеими хроматограммами можно сказать, что качество второй значительно лучше. Там я встретился с меньшим количеством проблемных участков. Причиной тому можно считать хорошее соотношение сигнал-шум, а также более короткий нечитаемый участок.
Отчёт о проблемах при редактировании автоматического прочтенияОпишу некоторые из проблемных ситуаций, возникших в ходе редактирования автоматического прочтения последовательностей ДНК. Я работал как с прямой, так и с обратной-комплементарной последовательностью. В ходе работы возникали, помимо прочих, такие случаи, когда однозначный выбор нуклеотида был невозможен. Тогда на этой позиции в [FASTA]-файле ставился один из так называемых вырожденных кодов (ambiguity codes[1]):

В Таблице 1 приведены несколько проблемных ситуаций и иллюстрации, их поясняющие. В верхней части двойных изображений всегда показана прямая, а снизу — обратная-комплеменатрная цепочка. Участки выровнены. (Выравнивание после удаления нечитаемых участков такое, что 1-ый нуклеотид прямой цепи соответствует 38-ому нуклеотиду обратной-комплементарной.
| Проблема | Описание | Изображение |
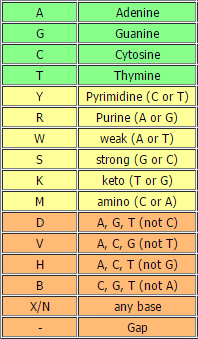
| Полиморфизм нуклеотидов | В 156-ой позиции прямой цепи нуклеотид был неопознан из-за перекрывания пиков сигнала и шума(пики А и Т). Но при обращении к обратной-комплементарной цепи(нуклеотид 524) всё становится — виден четкий пик аденина и несильный сигнал тимина между 2 пиками тимина, а также скорее всего шумовой сигнал гуанина(одиночный несильный пик). В FASTA-файл в этой позиции поставлен вырожденный код w(A или T). | 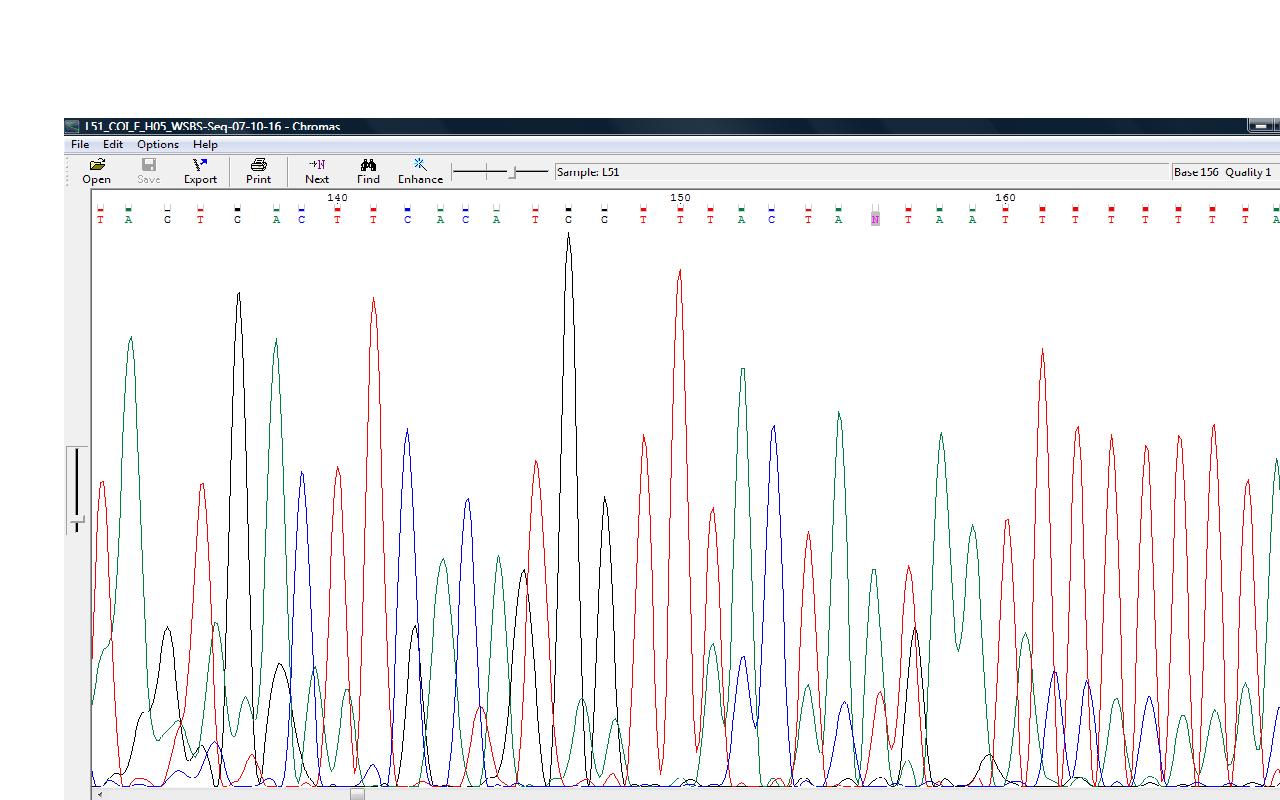 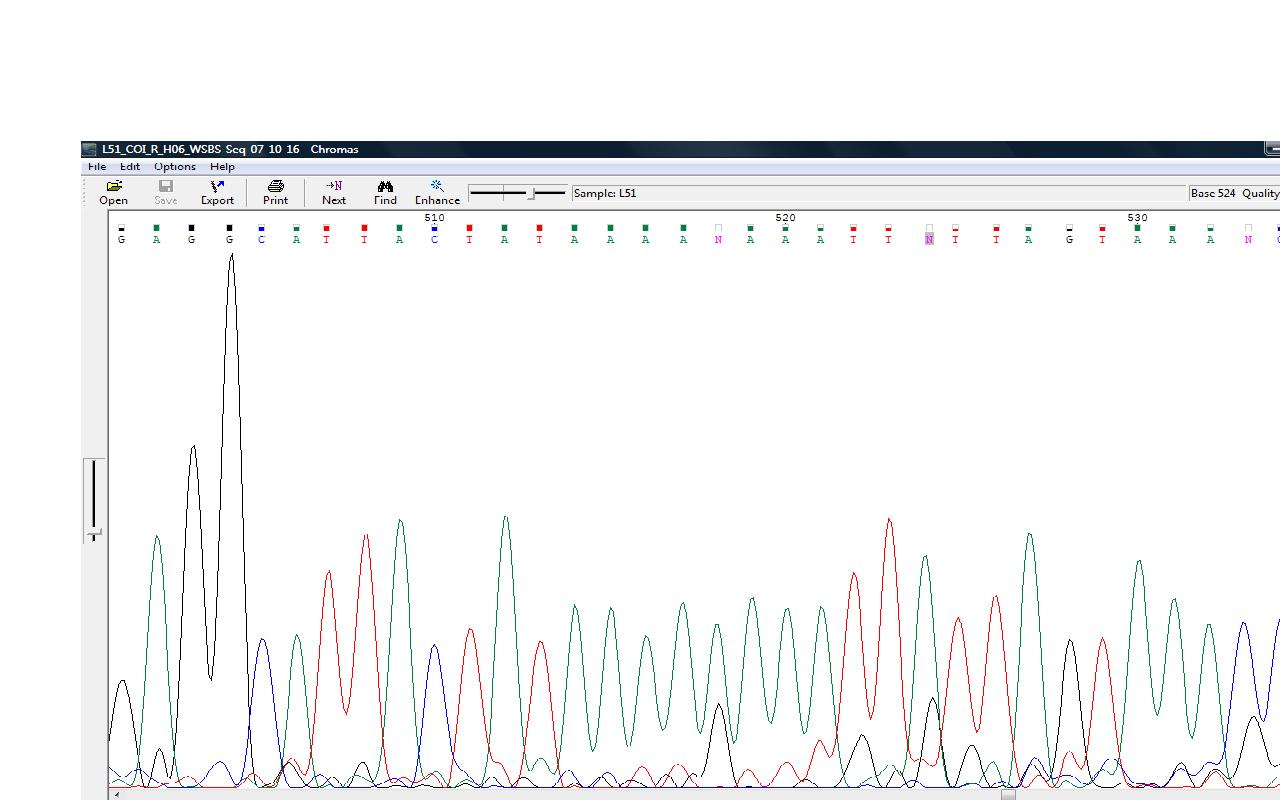 |
| Слишком высокий шум затрудняет автоматическое определение нуклеотида | При рассмотрении обратной-комплементарной цепи в позиции 190 нуклеотид не распознан программой. Имеется высокий пик гуаниеа и невысокий пик аденина. В комплементарной цепи четко распознан программой цитозин. В FASTA-файл в этой позиции поставлен гуанин g. |  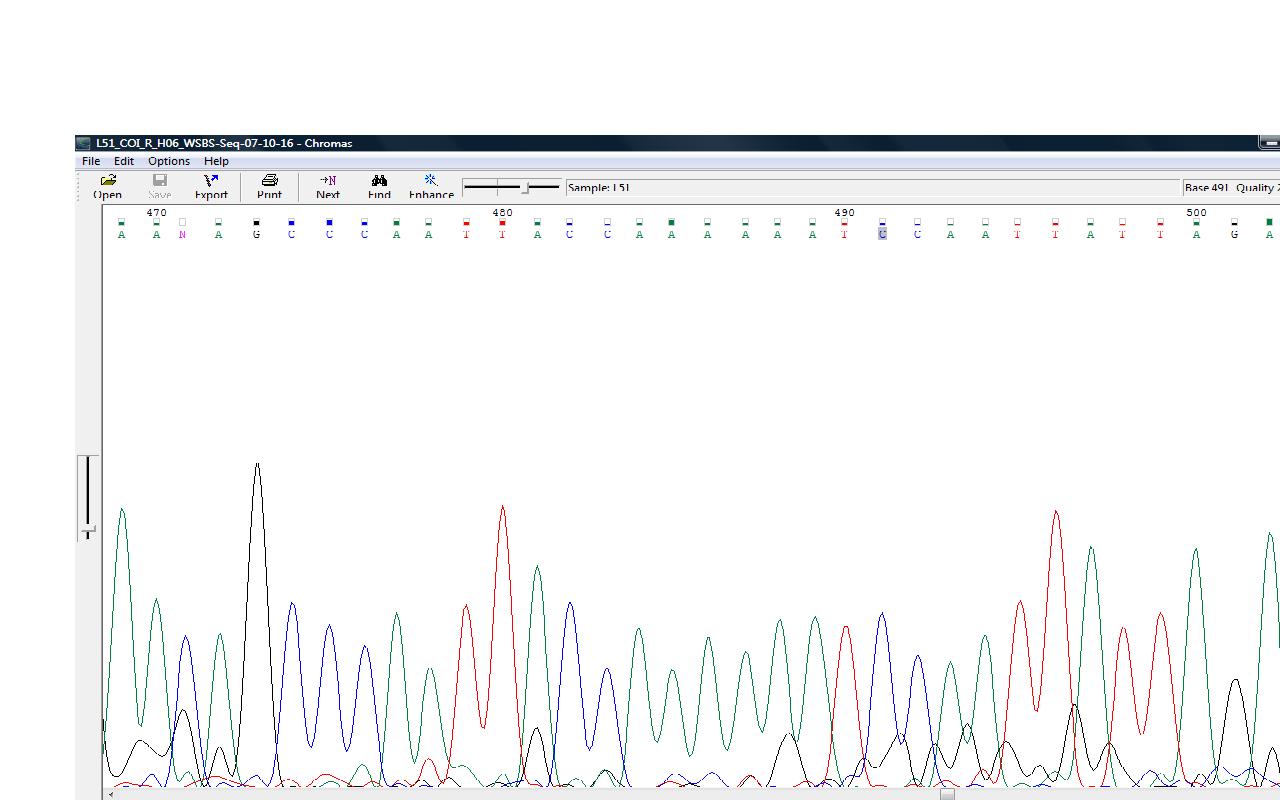 |
| Слишком высокий шум затрудняет автоматическое определение нуклеотида | В 240-ой позиции при рассмотрении замечаем высокий пик гуанина и невысокий аденина. На обратной цепи нуклеотид определен программой(441 нуклеотид на комплементарной цепи) как цитозин. В FASTA-файле записан гуанин g. | 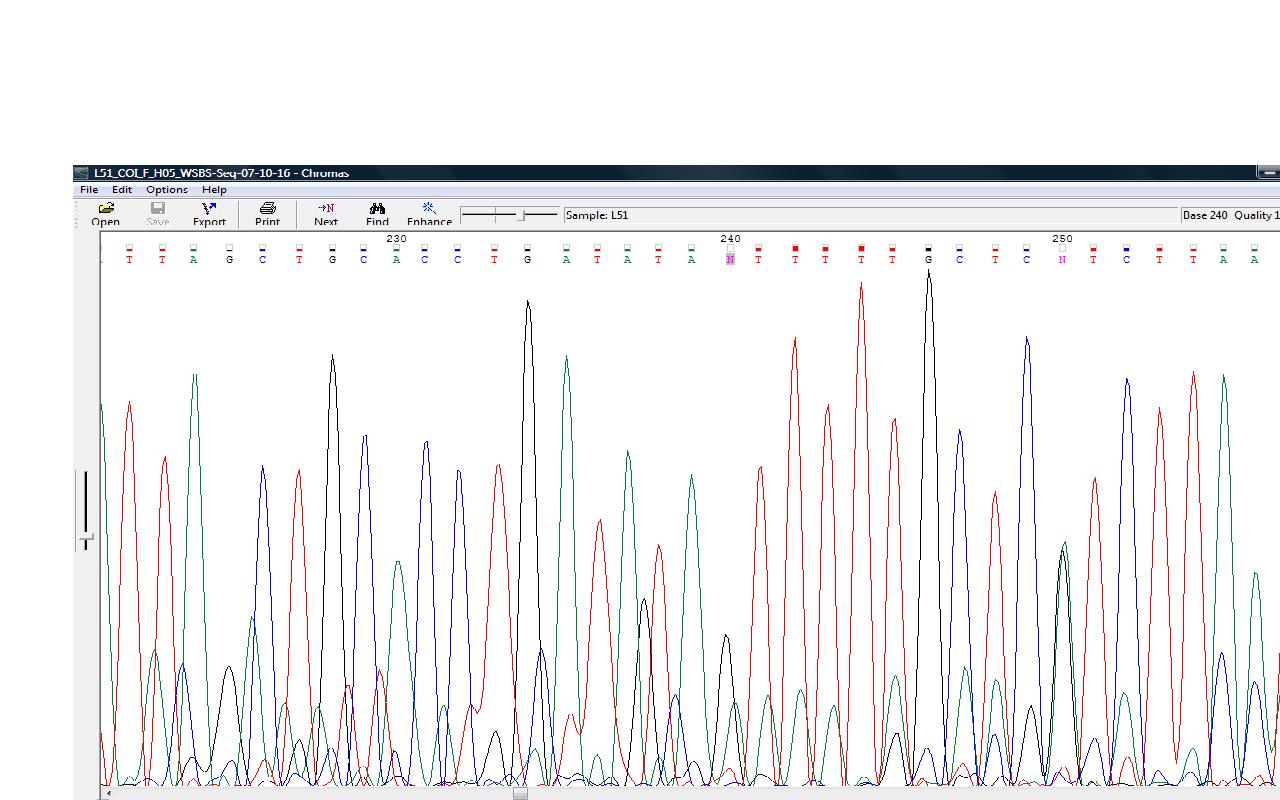 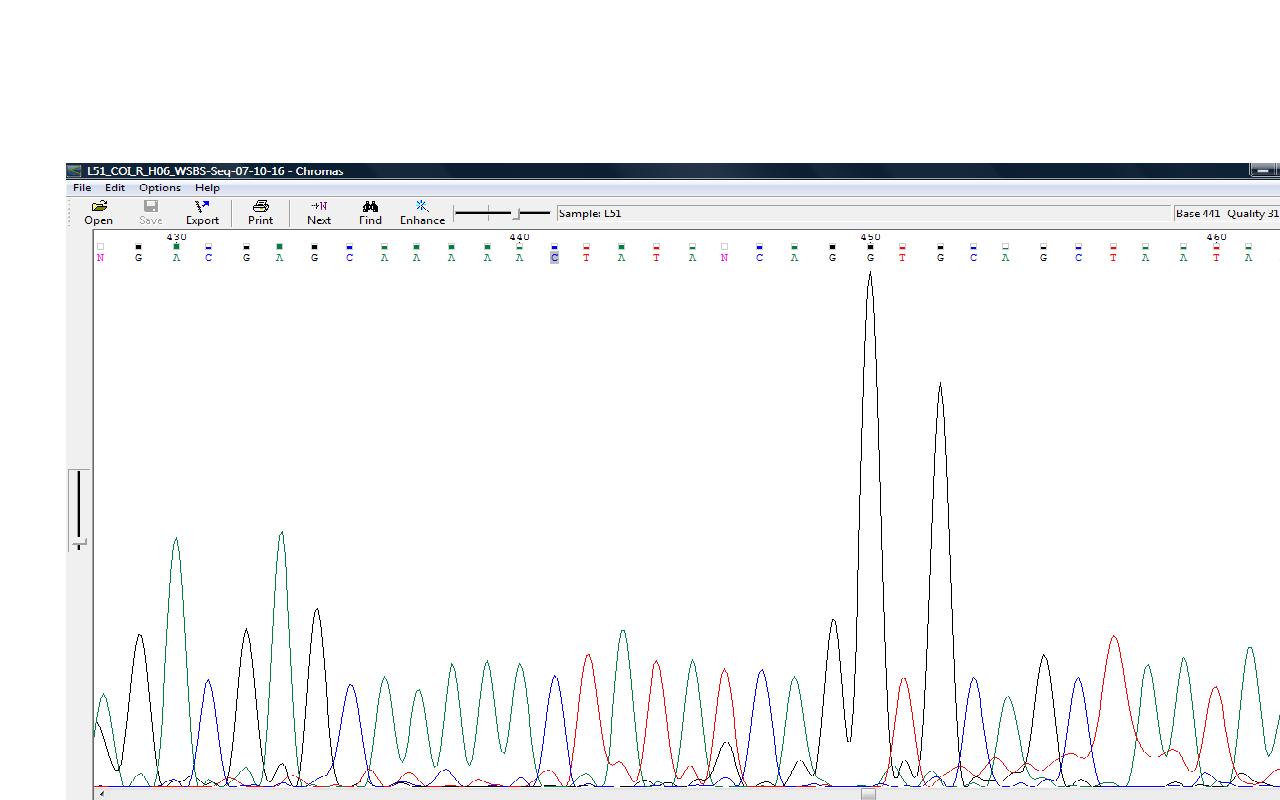 |
| Полиморфизм нуклеотидов | В 250 нуклеотиде мы видим примерно одинаковые пики гуанина и аденина. На обратной цепи(431 нуклеотид) мы видим большой пик цитозина и очень маленький смещенный пик тимина. ПОскольку на прямой цепи пики примерно равны в FASTA-файле стоит вырожденный код — r (A или G). Можно отметить, что маленький пик тимина на обратной цепи располагался ровно между 431 и 432 нуклеотидом. | 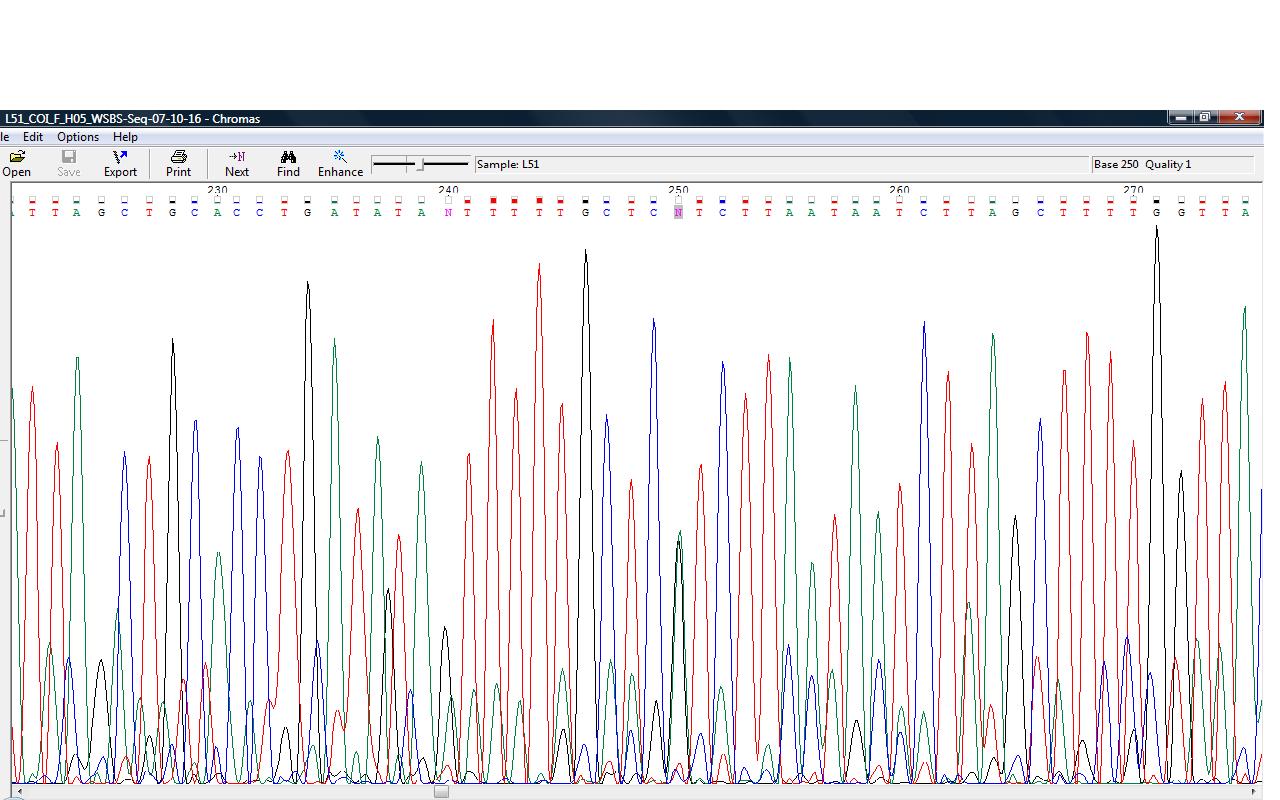 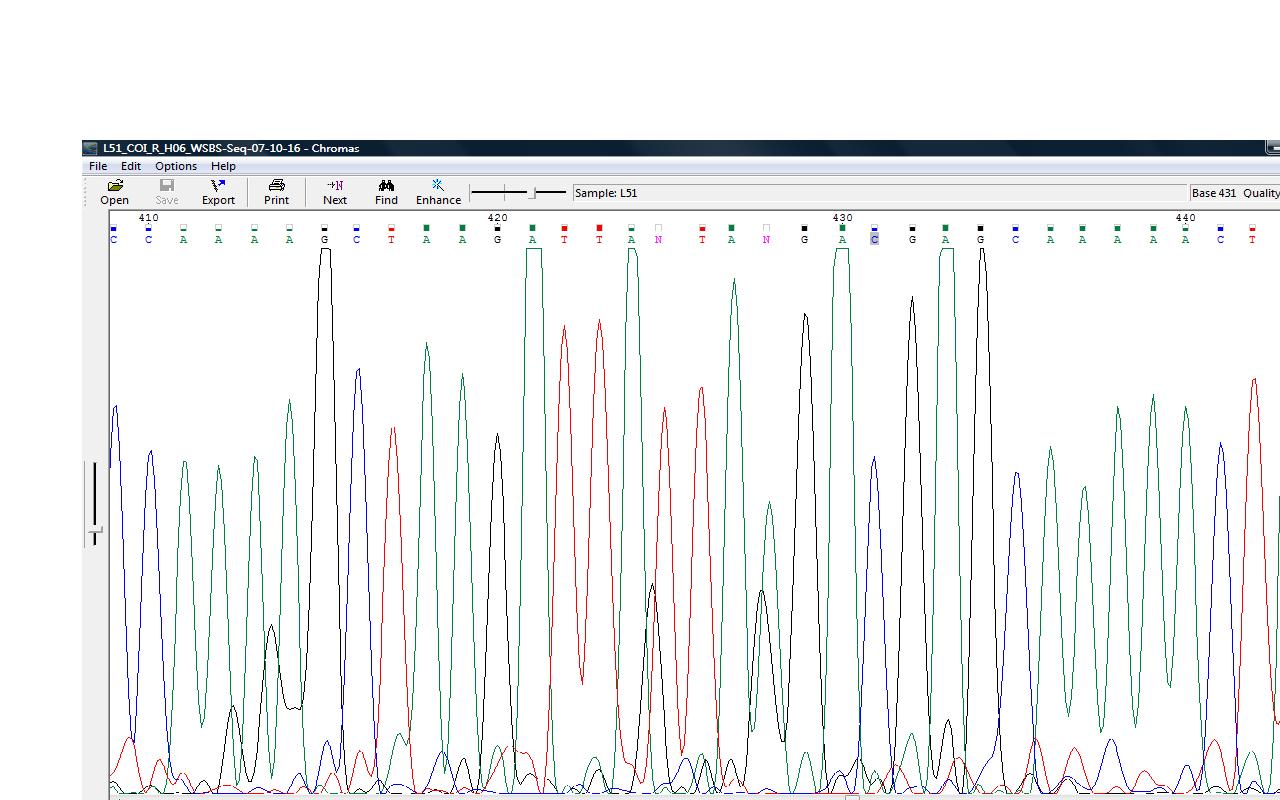 |
После проведенного редактирования полученные [FASTA]-последовательности прямой и обратной-комплементарной цепей были выровнены с ипользованием Muscle with defaults, а полученное выравнивание раскрашено по нуклеотидам.

Консенсусная последовательность:
>Consensus/1-663 Percentage Identity Consensus
TGGGAGGDTATATTYTGTTTTAVGTTTATGGTCTGGCTTAGTGGGGTTGGTATACAGGACTATAATGCGTAC
AGAGTTAATACATCCMGGTTCTTTTTATGGTGAGTCMGTTTATAATGTTTTAGTGACTTCACMTGGTTTACT
AWYAATTTYTTTTATAGTAATGCCTCTAATAATTGGATTTTTTGGAAATTGGGCTGTTCCCCTTTTATTAGC
TGCACCTGATATAGTTTTTGCTCRTCYTAMTAATCTTAGCTTTTGGTTACTTCCTGCGGCTACTATTTTMTT
GCTAATATCTAATGAAGTGRAGGAAGGAGTTGGGACGGGTTGAACACTTTACCCCCCTTTATCTGCTYGATT
AGGTCATCCTGCCCCADCGATGGAGYTTATAATTTTMGGGCTACATATTGCTGGAATAACTTCTATTTTTGC
AAGAATTAATTTCGTAACTACAGGTGCTAATATGCGACCTGAGGGGGTGGCTCCTCRSCGAMCTACCTTGTT
TGTGGTCTCAGTGGTAAYYACATCATTTYTACTGGTGGTTGCCATACCCGTACTABCTGCCGGCTTAACTAT
ACTTCTTACTGACMRAAATTTTAATACTTCTTTTTTTGATCCGGTAGGAGGAGGGGACCCTGTTTTATTTAT
TCATTTGTTTTGDTA
На рисунке ниже приведён пример действительно плохой по качеству хроматограммы. Её файл доступен для скачивания.
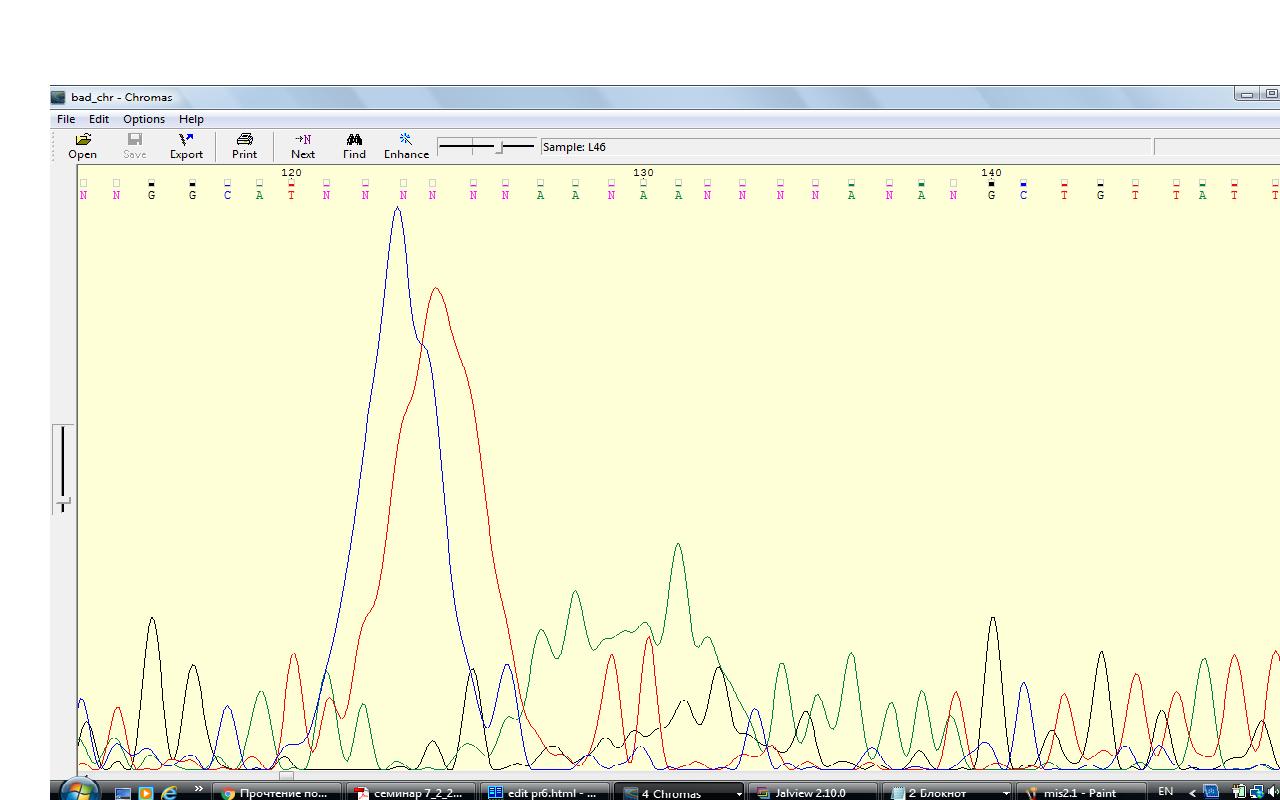
Как видим, автоматического прочтения не произошло. И это неудивительно, при просматривании хроматограммы на всем протяжении шум неотличим от сигнала, встречаются огромные пики, размытые и нечёткие. Возможно, в образце содержались разные цепочки ДНК.