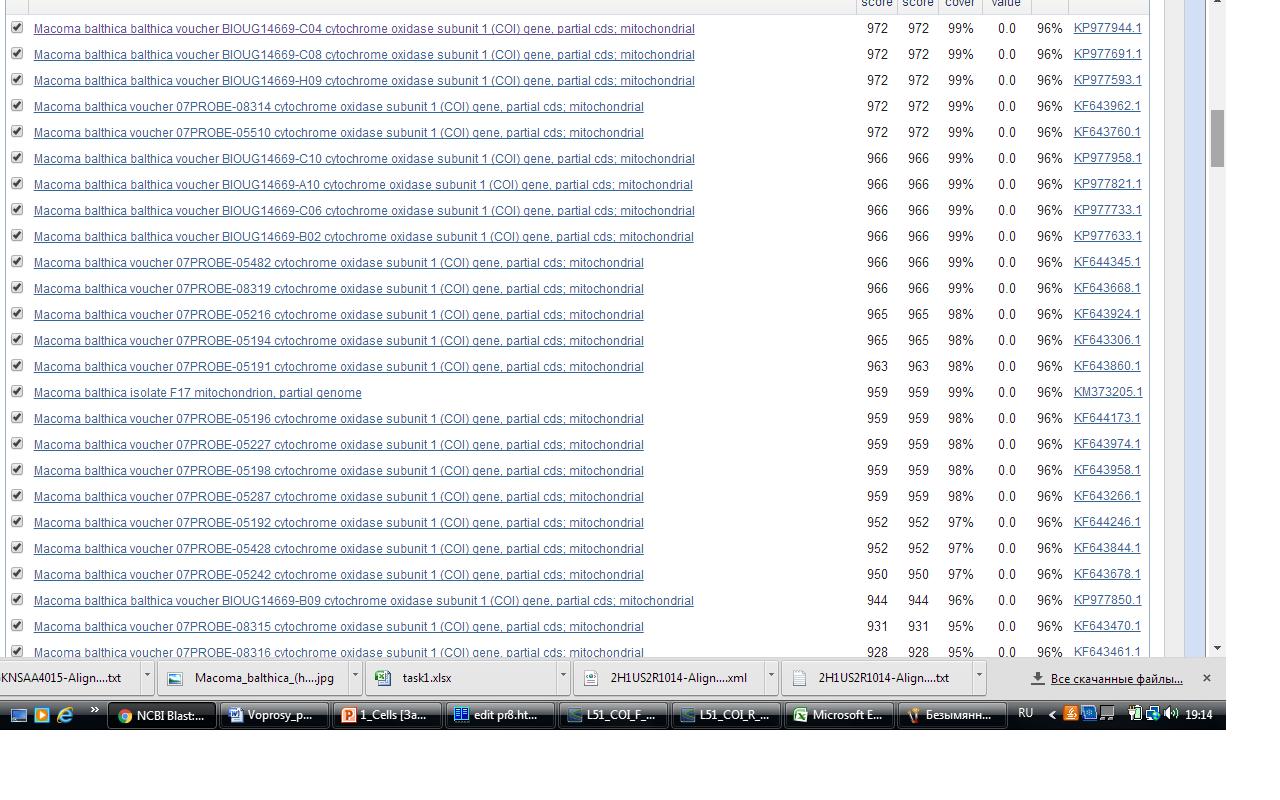
В первом задании необходимо было определить таксономию и функцию прочтенной в практикуме 6 последовательности. Последовательность в формате fasta была подана на вход в blastn. Поиск осуществлялся по базе данных Nucleotide collection со стандартными параметрами алгоритма blastn (за исключением количества находок: 1000). Так было построено 108 выравниваний (скачать таблицу в Excel: task1.xlsx). На рис.1 представлена выдача blastn, а на рис.2 представлена суммарная статистика по всем находкам. Предположительно последовательность - ген BIOUG14669-C04 субъединицы 1 цитохром c-оксидазы(COI) митохондриальный геном балтийской макомы (Macoma balthica), так как при поиске гомологов при помощи BLAST все найденные гомологи - митохондриальные гены субъединицы 1 цитохром -оксидазы(COI) балтийской макомы (Macoma balthica).
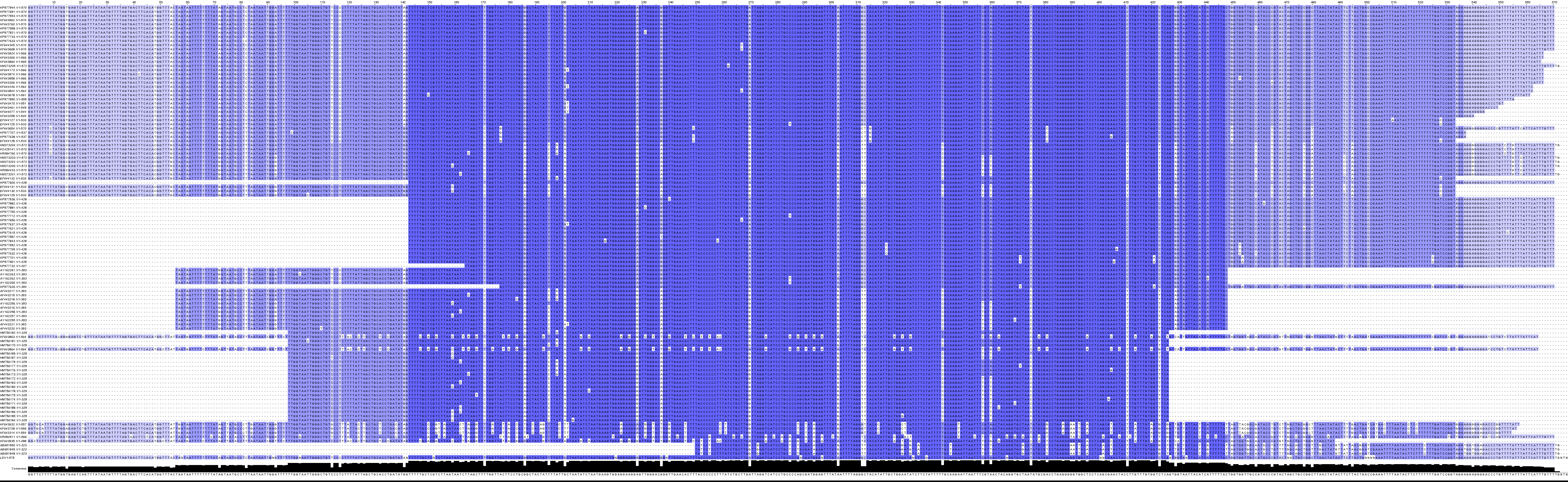
Выравнивание в формате msf.
Выравнивание в формате fasta.
Весь проект в формате jvp.
Так как при построении дерева последовательностей в Jalview исходная последовательность оказалась в одной из средних ветвей дерева, а все анализируемые последовательности принадлежат одному виду, то можно заключить, что исходная последовательность тоже принадлежит к виду Macoma balthica. Первые 42 находки имели следующие характеристики: E-value = 0, Identity >= 90%, Query cover >= 92%, Score = [721-972]. Первые 67 находок имели E-value = 0. Далее шли находки с меньшим покрытием, идентичностью и весом. Все находки относятся к организму Macoma balthica. Наибольшего внимания заслуживают первые пять находок, имеющие одинаковый максимальный Score = 972 , Identity = 96% , Query cover = 99%. Для наглядности в Jalview было построено выравнивание (Mafft with defaults) пяти последовательностей этого видов (Macoma balthica 5 сверху) и исходной последовательности (рис. 4).
Таксономия до вида: Eukaryota; Opisthokonta; Metazoa; Eumetazoa; Bilateria; Protostomia; Lophotrochozoa; Mollusca; Bivalvia; Heteroconchia; Euheterodonta; Veneroida; Tellinoidea; Tellinidae; Limecola; Limecola balthica

Рис. 4. Выравнивание исходной последовательности с 5 гомологами.
Выравнивание в формате msf.
Выравнивание в формате fasta.
Весь проект в формате jvp.
Балтийская макома |
Балтийская макома (лат. Macoma balthica) — вид морских двустворчатых моллюсков из семейства теллинид.
Раковина треугольной формы красного, жёлтого, зелёного или бело-коричневого цвета длиной от 2 до 3 см.
Внутренняя сторона раковин часто красного цвета.
Моллюск обитает в ваттах, зарываясь в песок на глубину 4—10 см. Питается водорослями.
Вид распространён в северной части Атлантического и Тихого океана.

Рис. 5. Фотография балтийской макомы.
Цитохром с-оксидаза
Рис. 6. Цитохром с-оксидаза.
Задание 2. Сравнение списков находок разными алгоритмами BLAST.
Было призведено сравнение списков находок нуклеотидной последовательности тремя алгоритмами BLAST:
blastn, megablast и discontiguous megablast.
Чтобы результат сравнения был показателен, необходимо
было ограничить область поиска. Поиски по виду (Limecola balthica (taxid:1903275)) и по роду (Limecola (taxid:1903274)) давали слишком
малую разнообразность находок (почти все находки имели E-value>2e-130 99% сходство со входной более 90%). Когда область была
расширена до семейства (Tellinidae (taxid:52798)), а максимальное число находок
(Max target sequences) увеличено до 5000, число разнообразных находок оказалось
достаточным.
| Параметры запуска BLAST | |||||||
|---|---|---|---|---|---|---|---|
| Database | Max Target Sequences | Expect Threshold | Word Size | Max matches | Match/Mismatch Scores | Gap Costs | |
| megablast | Nucleotide collection (nr/nt) | 5000 | 10 | 28 | 0 | 1, -2 | Linear |
| discontiguous megablast | Nucleotide collection (nr/nt) | 5000 | 10 | 11 | 0 | 2, -3 | Existence:5, Extention: 2 |
| blastn | Nucleotide collection (nr/nt) | 5000 | 10 | 11 | 0 | 2, -3 | Existence:5, Extention: 2 |
| Результаты выдачи megablast |
|---|
| Результаты выдачи discontiguous megablast |
|---|
| Результаты выдачи blastn |
|---|
| Сравнение алгоритмов | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Алгоритм | Число находок | Score лучшей находки | Score худшей находки | E-value лучшей находки | E-value худшей находки | Ident лучшей находки | Ident худшей находки | Query cover лучшей находки | Query cover худшей находки |
| megablast | 108 | 949 | 259 | 0.0 | 3e-70 | 96% | 56% | 99% | 80% |
| discontiguous megablast | 258 | 949 | 93.3 | 0.0 | 2e-20 | 96% | 49% | 99% | 68% |
| blastn | 266 | 949 | 24.7 | 0.0 | 9.0 | 96% | 2% | 99% | 94% |
Больше всего находок выдал алгоритм blastn.
Пример находки, найденной им и не найденной другими:
Tellinella staurella mitochondrial gene for 16S ribosomal RNA, partial sequence (Max Score 24.7, Query Cover 100%, E-value 9.0, Ident 2%).
Меньше всего находок дает алгоритм megablast.
Пример находки, найденной discontiguous megablast и blastn, но не найденной megablast:
Macoma petalum voucher 11BFMOL-0234 cytochrome oxidase subunit 1 (COI) gene, partial cds; mitochondrial (Max Score 495, Query Cover 85%, E-value 2e-141, Ident 74%).
Нужно заметить, что при поиске с помощью discontiguous megablast и blastn все находки были одинаковы за исключением 8 последовательностей, найденных blastn и не найденных discontiguous megablast. Эти 8 последовательностей имели E-value 9.0 , Ident 2% , Max Score 24.7, 7 из них имели Query Cover 94% , одна имела Query Cover 100%. Проведенное сравнение позволяет сделать вывод, что алгоритмы blastn и discontiguous megablast при использованных параметрах работают примерно одинаково и выдают почти не отичающееся число находок почти одинакового качества. Megablast работает с намного более высокими требованиями к качеству находки и выдает последовательности наиболее близкие к исходной и отсеивает большее количество находок. У megablast относительно высокие требования к E-value, а Query cover не может быть ниже 28 нуклеотидов, поэтому он может не находить короткие последовательности. Лучшие 30 находок всех трех алгоритмов одинаковы, что позволяет сделать вывод, что все алгоритмы одинаково ищут последовательности с высокой степенью сходства.
Задание 3. Проверка наличия гомологов белков
В этом задании было необходимо проверить наличие гомологов определенных белков в геноме организма X5 (Amoboaphelidium protococarum)
c помощью локального BLAST.
Для начала я создал локальную базу данных с помощью
операции
makeblastdb -in X5.fasta -dbtype nuclа затем для каждого из выбранных белков запустил по ней алгоритм tblastn, находящий гомологи белка в формальной трансляции нуклеотидного банка с помощью операции
tblastn -query xxx.fasta -db X5.fasta > xxx.out
Белок HSP7C_HUMAN
HSP7C_HUMAN - консервативный шаперон HSP70, белок теплового шока. Имеется у большинства организмов из всех царств.
Играет роль репрессора активации транскрипции. Является компонентом PRP19-CDC5L комплекса, формирующего интегральную часть
сплайсосомы, и необходим для активации сплайсинга пре-мРНК. Связывая бактериальные ЛПС (липополисахариды), является посредником в процессах
ЛПС-индуцированного воспаления, включая секрецию моноцитами TNF (фактора некроза опухоли) .
Результаты tblastn по базе данных X5.fasta для данного белка: HSP7C_HUMAN.out.
| Всего находок - 16 |
|---|
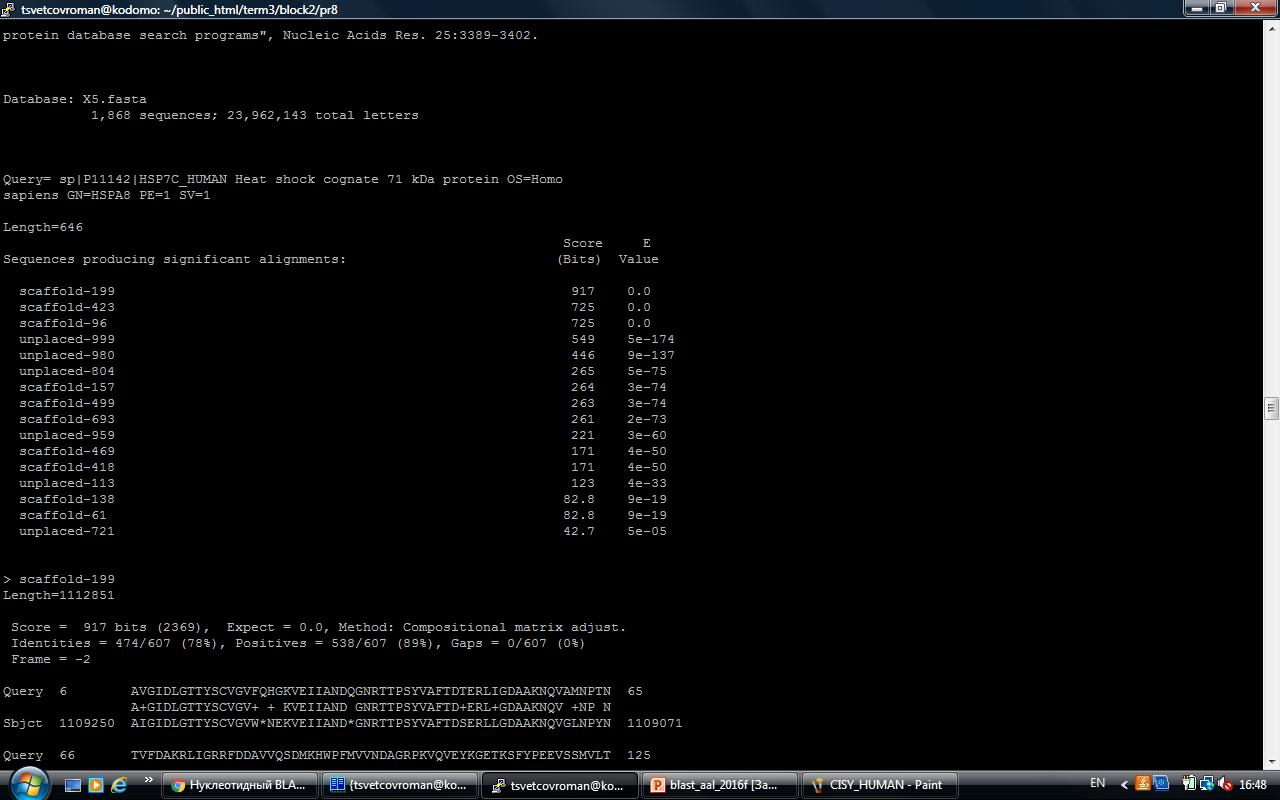 |
Лучшая находка имеет следующие параметры:
> scaffold-199
Length=1112851
Score = 917 bits (2369), Expect = 0.0, Method: Compositional matrix adjust.
Identities = 474/607 (78%), Positives = 538/607 (89%), Gaps = 0/607 (0%)
Frame = -2
У находки хороший E-value, достаточно высокие проценты Positives и Identities. Параметры сходства достаточны, чтобы назвать ее гомологом исследуемого белка, вероятно выполняющим ту же функцию.
Белок TERT_HUMAN
TERT_HUMAN - теломераза, восстанавливающая длину хромосомы при репликации. Имеется у большинства,
но не у всех эукариот. Активна в прогениторных и раковых клетках, в нормальных же соматических не
активна или проявляет очень низкую активность. Играет важную роль в процессах старения и предотвращении
апоптоза.
Результаты tblastn по базе данных X5.fasta для данного белка: TERT_HUMAN.out.
| Всего находок - 3 |
|---|
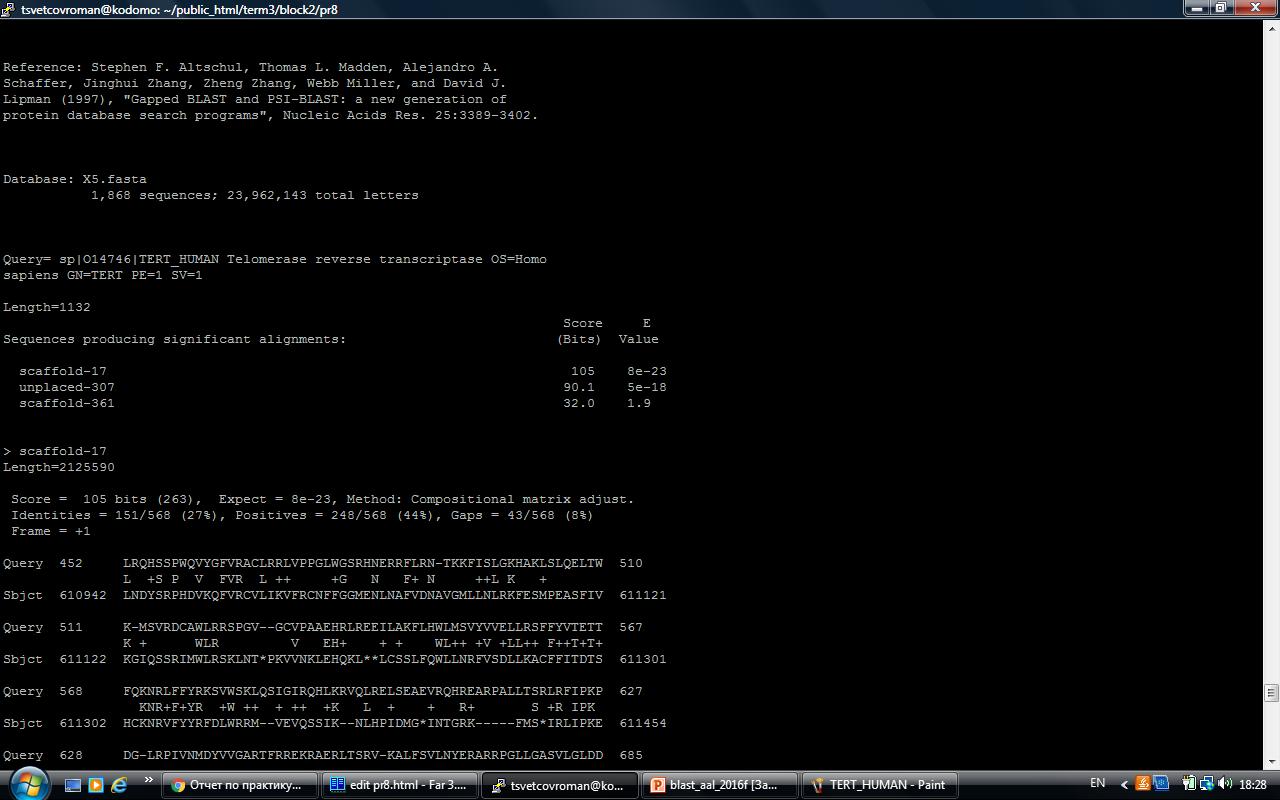 |
Лучшая находка имеет следующие параметры:
> scaffold-17
Length=2125590
Score = 105 bits (263), Expect = 8e-23, Identities = 151/568 (27%),
Positives = 248/568 (44%), Gaps = 43/568 (8%), Frame = +1
Этот результат является положительным. BLAST выдал три находки, однако даже у самой лучшей из них параметры сходства(Identities и Positives) слишком низкие, чтобы наверняка утверждать сохранение функций. Можно предположить, что теломераза TERT_HUMAN либо отсутствует, так как она не является жизненно важной либо ее функцию выполняет другая теломераза. Гомология отдельных доменов маловероятна, потому что совпадения распределены по всей длине последовательности относительно равномерно.
Белок CISY_HUMAN
CISY_HUMAN - митохондриальная цитратсинтаза. Участвует в цикле трикарбоновых кислот.
Проявляет каталитическую активность в реакции Acetyl-CoA + H2O + oxaloacetate = citrate + CoA.
Результаты tblastn по базе данных X5.fasta для данного белка: CISY_HUMAN.out.
| Всего находок - 4 |
|---|
 |
Лучшая находка имеет следующие параметры:
> scaffold-693
Length=1268102
Score = 565 bits (1457), Expect = 2e-180, Method: Compositional matrix adjust.
Identities = 262/377 (69%), Positives = 315/377 (84%), Gaps = 3/377 (1%)
Frame = +1
Скорее всего она действительно является гомологом интересующего нас белка с сохранением функций ввиду относительно хороших параметров сходства.
Задание 5. Поиск гена белка, закодированного в одном контиге ''Amoboaphelidium''
Для поиска я выбрал контиг scaffold-258. Его длина составляет 99209 п.н., следовательно, на нем
вполне может поместиться ген.
Информация о длинах контигов была получена с помощью команды
infoseq X5.fasta -only -name -lengthС помощью операции
seqret X5.fasta: unplaced-982 -out
unplaced982.fasta я извлек последовательность выбранного контига в отдельный файл
(c.fasta).Далее был запущен blastn с параметрами по умолчанию и ограничению по таксону Amoeboaphelidium (taxid:1243176).
Результаты работы blastn:
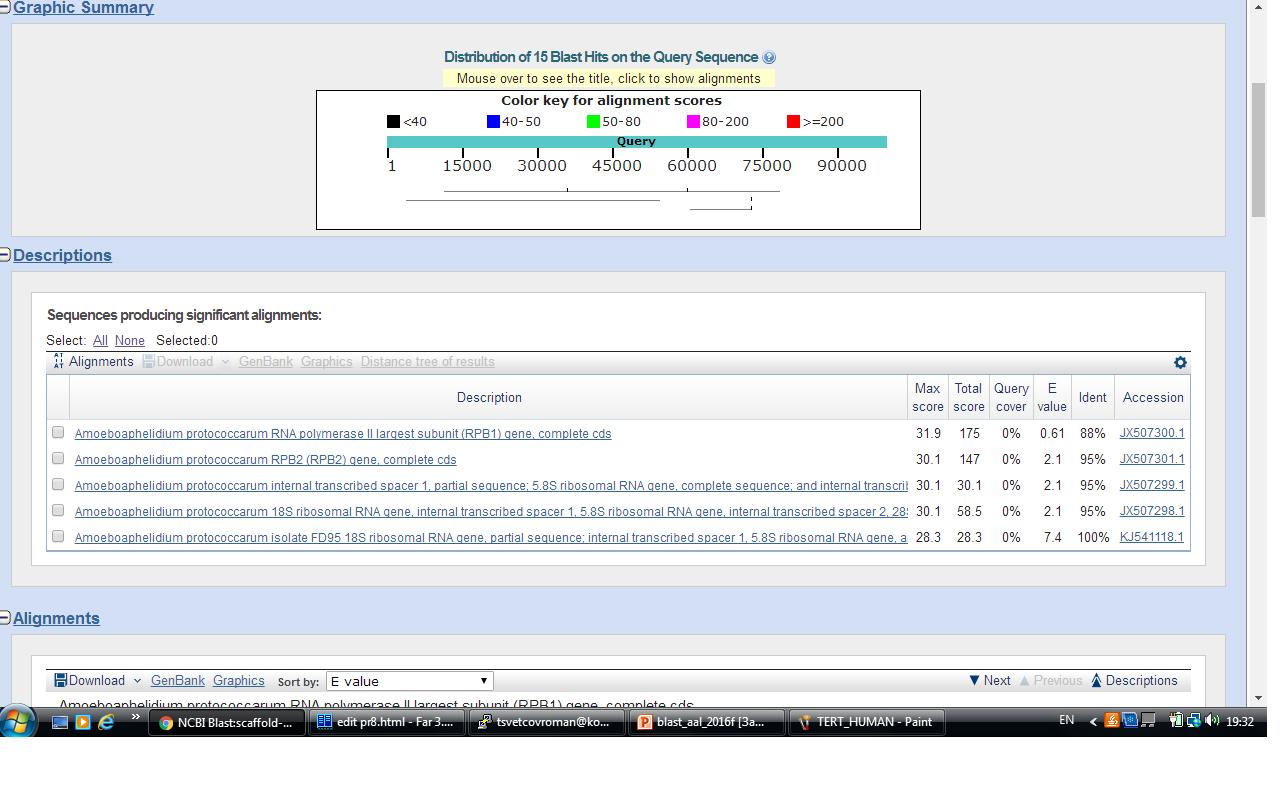
Как видно из рисунка, blastn выдает 5 находкок,сходных по всем параметрам. Ident находок довольно высокий:от 88% до 100%, однако покрытие составляет 0% и E-value очень высок. Несмотря на это, мы можем предположить, что в контиге закодированы гены, указанные в данных находках, так как находки и аназизируемый геном принадлежат одному роду.
Найдено 5 генов, 1 из которых кодирует белки больших субъединиц РНК-полимеразы II(RPB1) , 1 ген кодирует внутренний спейсер1, 5.8S рибосомальную РНК и внутренний спейсер2, 1 ген кодирует 18S рибосомальную РНК и внутренний спейсер1 5.8S рибосомальной РНК и внутренний спейсер2 28S рибосомальной РНК, межгенный спейсер1, 5S рибосомальную РНК и межгенный спейсер2. 1 ген ген, кодирует 18S рибосомальную РНК и внутренний спейсер1 5.8S рибосомальной РНК и внутренний спейсер2, 1 ген не указанной функцией, скорее всего функция этого гена неизвестна.
Находки:
- Находка Amoeboaphelidium protococcarum RNA polymerase II largest subunit (RPB1) gene, complete cds - ген , который кодирует белки больших субъединиц РНК-полимеразы II(RPB1).
- Находка Amoeboaphelidium protococcarum RPB2 (RPB2) gene, complete cds - ген с не указанной функцией.
- Находка Amoeboaphelidium protococcarum internal transcribed spacer 1, partial sequence; 5.8S ribosomal RNA gene, complete sequence; and internal transcribed spacer 2, partial sequence - ген, кодирующий внутренний спейсер1, 5.8S рибосомальную РНК и внутренний спейсер2.
- Находка Amoeboaphelidium protococcarum 18S ribosomal RNA gene, internal transcribed spacer 1, 5.8S ribosomal RNA gene, internal transcribed spacer 2, 28S ribosomal RNA gene, intergenic spacer 1, and 5S rRNA ribosomal RNA gene, complete sequence; and intergenic spacer 2, partial sequence - ген, кодирующий 18S рибосомальную РНК и внутренний спейсер1 5.8S рибосомальной РНК и внутренний спейсер2 28S рибосомальной РНК, межгенный спейсер1, 5S рибосомальную РНК и межгенный спейсер2.
- Находка
Amoeboaphelidium protococcarum isolate FD95 18S ribosomal RNA gene, partial sequence; internal transcribed spacer 1, 5.8S ribosomal RNA gene, and internal transcribed spacer 2, complete sequence; and 28S ribosomal RNA gene, partial sequence - ген, кодирующий 18S рибосомальную РНК и внутренний спейсер1 5.8S рибосомальной РНК и внутренний спейсер2.
Источники