1. С помощью команды
seqret -sequence embl:CP013715 -outseq gff::a.gff -featureбыл скачан файл a.gff, содержащий плазмиду CP013715 в формате gff. При этом в итоговом файле a.gff были сохранены особенности (features) с помощью квалификатора -features, так как они понадобятся для дальнейшей работы.
Затем с помощью команды
seqret -sequence embl:CP013715 -outseq fasta::a.fasta -featureбыл скачан файл a.fasta, содержащий плазмиду CP013715 в формате fasta.
2. Выданная мне плазмида принадлежит Staphylococcus equorum и относится к цепи C2014 (strain C2014 plasmid pC2014-1). Staphylococcus equorum - это Грамм-положительная, коагулаза-отрицательная бактерия, принадлежащая к роду Staphylococcus (Стафилококк).
Систематика:
Царство: Bacteria
Отдел: Firmicutes
Класс: Bacilli
Порядок: Bacillales
Семейство: Staphylococcaceae
Род: Staphylococcus
Вид: Staphylococcus equorum
Представители данного рода — неподвижные грамположительные кокки, диаметр клетки которых составляет от 0,6 до 1,2 мкм. Для представителей рода характерно деление в нескольких плоскостях, результатом чего есть расположение микробных клеток «виноградными гроздьями» в чистой культуре. Стафилококки — факультативные анаэробы, хемоорганотрофы с окислительным и ферментативным типом метаболизма, каталазопозитивные и оксидозонегативные. Не образуют спор или капсул. Некоторые стафилококки синтезируют характерные пигменты. Формы колоний на твердых средах округлые, выпуклые, пигментированные (белые, желтые, золотистые). На жидких - равномерное помутнение. Широко распространены в почве, воздухе, представители нормальной кожной микрофлоры человека и животных. В состав этого рода входят патогенные и условно-патогенные для человека виды, колонизирующие носоглотку, ротоглотку и кожные покровы. Патогенные стафилококки продуцируют эндо- и экзотоксины, ферменты, нарушающие жизнедеятельность клеток. Существует стафилококковый бактериофаг, обладающий способностью специфически лизировать стафилококковые бактерии. Известна достаточно высокая чувствительность стафилококков к водным растворам солей серебра и его электролитическим растворам.

В плазмиде 85 генов и 79 белок- кодирующих генов. В плазмиде я обнаружил много участков, не входящих в состав генов. Длина плазмиды составляет 80362 пар оснований. Средняя длина гена составляет 732,47 пар оснований(подсчитано с помощью Excel).
Ссылка на файл с расчетами:count.xlsx.
3. С помощью программы g.py из файла a.gff были получены координаты генов и записаны в файл AnyFileName.txt. Затем с использованием программмы Prodigal было получено предсказание координат генов и записано в файл genes.cdi. С помощью команды
prodigal -i cp015748.fasta -o prodigal.fasta -f scoбыло получено предсказание генов программой prodigal для данной плазмиды. При этом был выбран (-f sco) минималистичный формат sco для записи результата: prodigal.fasta. Из обоих файлов координаты генов (начало, конец, ориентация, разделенные символом '_') были записаны в отдельные файлы с помощью команд:
grep CDS a.gff | cut -f 4,5,7 --output-delimiter='_'> a.out grep '>' prodigal.fasta | cut -f 2,3,4 -d '_' > prodigal.outПолученные файлы с координатами: prodigal.out, a.out.
В записи Genbank записан 81 ген, программа Prodigal предсказала 87 генов. 4. Далее был создан скрипт a.py, с помощью которого были оценены количественно следующие показатели: - Число и % генов, которые аннотированы в GenBank и для которых предсказание обоих концов гена с помощью Prodigal точно такое же; - Число и % генов, для которых аннотация только N-конца белка не совпадает с аннотацией Prodigal; - Число и % генов, для которых аннотация только С-конца белка не совпадает с аннотацией Prodigal; - Число и % генов, для которых аннотация обоих концов не совпадает с аннотацией Prodigal. Примечание: Процент считался от общего числа предсказанных Prodigal генов, так как их было больше аннотированных в GenBank'е генов.
4. Краткое описание алгоритма работы скрипта.
1. Скрипт читает файлы prodigal.out и a.out . Для каждого файла он создает 2 списка: в одном начало гена с учетом ориентации гена, в другом конец гена с учетом ориентации гена.2. Далее скрипт для каждой пары начало-конец из файла a.out каждое начало и каждый конец сравнивает со всеми парами начало-конец из файла prodigal.out, в случае если есть совпадение для начала и для конца индикатор увеличивается на 1. После окончания работы цикла значение индикатора, отражающее число совпадений выводится на экран.
3. Число генов, у которых не совпадает N- или C-конец считается также, только сравниваются тоько концы в первом случае, и только начала во втором. В случае совпадения значений из первого и второго файла индикатор увеличивается на 1. После окончания работы цикла значение индикатора равно искомому числу генов плюс число полностью совпадающих генов. Для того, чтобы получить искомое число генов, вычитаем число полностью совпадающих генов, полученное в п. 2 и результат выводим на экран.
4. Чтобы получить число генов, у которых предсказание полностью не совпадает с записью в Genbank, каждую пару начало-конец из файла a.out сравниваем со всеми парыми из файла prodigal.out и считаем число несовпадений, с помощью индикатора x. Зная, что всего в файле prodigal.out 87 генов, в случае если число несовпадений равно 87 увеличиваем индикатор y. После окончания работы цикла выводим на экран значение индикатора y.
5. Результаты.
| Число генов, которые аннотированы в БД GenBank и для которых предсказание обоих концов гена с помощью Prodigal точно такое же | 47 |
| Процент генов, которые аннотированы в БД GenBank и для которых предсказание обоих концов гена с помощью Prodigal точно такое же | 58,02% |
| Число генов, для которых аннотация только N-конца белка не совпадает с аннотацией Prodigal | 7 |
| Процент генов, для которых аннотация только N-конца белка не совпадает с аннотацией Prodigal | 8,64% |
| Число генов, для которых аннотация только С-конца белка не совпадает с аннотацией Prodigal | 21 |
| Процент генов, для которых аннотация только С-конца белка не совпадает с аннотацией Prodigal | 25,93% |
| Число генов, для которых аннотация обоих концов не совпадает с аннотацией Prodigal | 6 |
| Процент генов, для которых аннотация обоих концов не совпадает с аннотацией Prodigal | 7,41% |
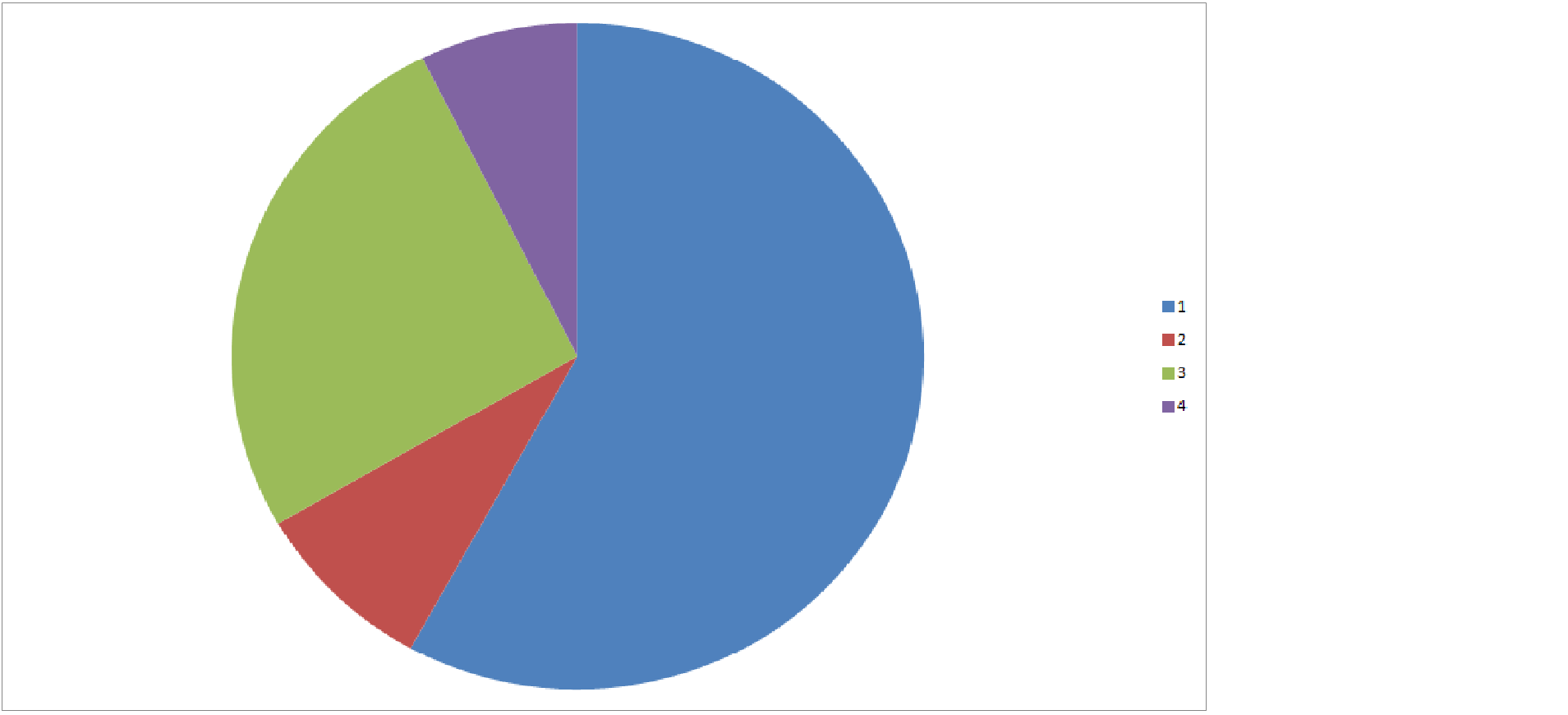
Рисунок 1. Круговая диаграмма, отображающая результаты сравнения предсказания Prodigal с записью Genbank. Цифрой 1 обозначены гены, которые аннотированы в БД GenBank и для которых предсказание обоих концов гена с помощью Prodigal точно такое же, цифрой 2 - гены, для которых аннотация только N-конца белка не совпадает с аннотацией Prodigal, цифрой 3 - гены, для которых аннотация только С-конца белка не совпадает с аннотацией Prodigal, цифрой 4 - гены, для которых аннотация обоих концов не совпадает с аннотацией Prodigal.
Пример 1. Не найденный Prodigal ген, аннотированный в Genbank. Ген имеет координаты 74746_74946_+. Данный ген не был предсказан Prodigal, и в границах данного гена, анотированного Genbank нет участков генов, предсказанных Prodigal. На рис. 2 данный ген выделен коричневым. В строке Product для данного гена указано "hypothetical protein". Это означает, что продукт данного гена неизвестен, что дает основания предположить, что данный ген отсутствует. Дополнительным аргументом в пользу этой версии является то, что он не был предсказан Prodigal. Для того, чтобы это проверить, был запущен BLAST с этим аннотированным геном (discontiguous megablast). В итоге была найдена 1 находка с E-value 7e-97. Эта находка является аннотацией самого анализируемого гена в Genbank, которая вводилась в поиск в BLAST. То есть BLAST не нашел гомологов аннотированного в Genbank гена. Результаты поиска можно посмотреть: s.txt. В качестве продукта указан "hypothetical protein". Можно сделать вывод, что в Genbank возможно допущена ошибка и предсказание Prodigal скорее всего верно.

Рис. 2Аннотированный в Genbank ген, не предсказанный Prodigal.
Пример 2. Не найденный Prodigal ген, аннотированный в Genbank. Ген имеет координаты 71311_71526_-. Данный ген не был предсказан Prodigal, и в границах данного гена, анотированного Genbank нет участков генов, предсказанных Prodigal. На рис. 2 данный ген выделен коричневым. В строке Product для данного гена указано "hypothetical protein". Это означает, что продукт данного гена неизвестен, что дает основания предположить, что данный ген отсутствует. Дополнительным аргументом в пользу этой версии является то, что он не был предсказан Prodigal. Для того, чтобы это проверить, был запущен BLAST с этим аннотированным геном (discontiguous megablast). Было найдено 4 находки со статистически значимым E-value:7e-105 , 7e-67 , 2e-56 , 2e-29. В з случаях в качестве продукта указан "hypothetical protein" , в 1 случае продукт не был указан(отсутствовала запись CDS и строка product). Результаты поиска можно посмотреть: s1.txt.
В данном случае, в отличие от примера 1 нельзя утверждать, что в Genbank допущена ошибка, однако вероятность этого все равно есть, хотя и намного ниже, чем в примере 1. Наличие 4 гомологов анализируемого гена позволяет предположить, что ген аннотирован в Genbank скорее всего верно. Можно предположить, что в данном случае Prodigal допустил ошибку при предсказании.

Рис. 3 Аннотированный в Genbank ген, не предсказанный Prodigal.
Дополнительное задание.
[1] Эпидермальный стафилококк: // http://lechimsya-prosto.ru/epidermalnyj-stafilokokk. [URL]