Укоренение в среднюю точку.
Для реконструкции филогенетического дерева мною был выбран белок с мнемоникой RL1, являющийся рибосомным белком L1. Из Swiss-Prot были получены последовательности белков с данной функцией из отобранных ранее бактерий. Для этого был запущен JalView. В меню File было выбрано Fetch sequences. Далее я щёлкните по "Select Database" и выбрал Uniprot. Через точку с запятой я записал следующие выражения:RL1_BACAN ; RL1_CLOBA ; RL1_CLOTE ; RL1_FINM2; RL1_LACAC ; RL1_STAAR; RL1_STAES ; RL1_STRPN. После нажатия OK появилось окно с последовательностями. В этом окне я выбрал Web Service, затем Alignment, затем Muscle with Defaults. Программой Muscle было построено их выравнивание (ссылка на выравнивание в fasta-формате). Затем для удобства от названия белка в выравнивании была оставлена только мнемоника вида. Полученное выравнивание в fasta-формате.

Методом "Neighbor Joining Using % Identity" было реконструировано филогенетическое дерево (ссылка на дерево в Newick-формате).
Ссылка на проект JalView
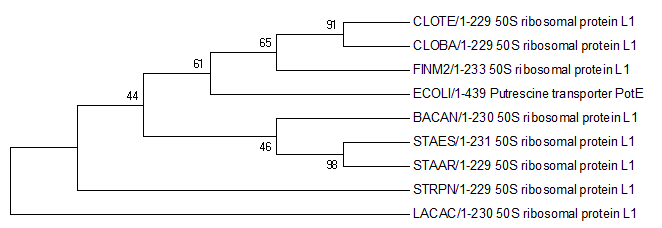
Полученное с помощью JalView дерево было сохранено. Изображение данного дерева можно увидеть ниже.
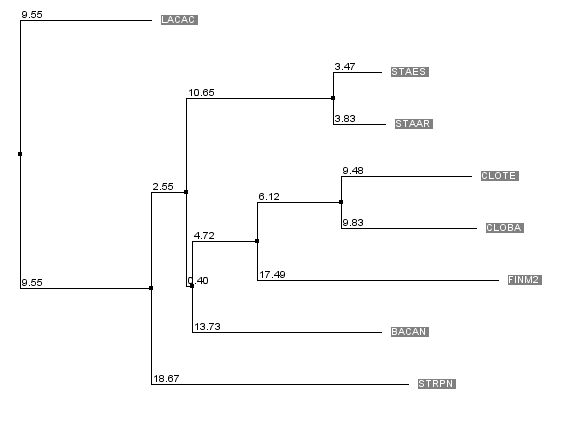
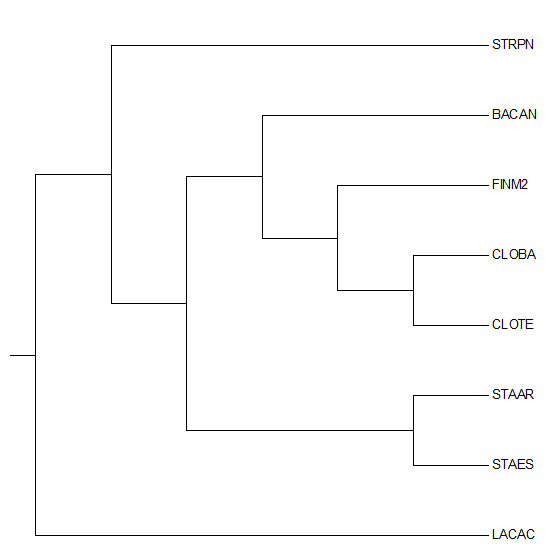
Затем данное дерево было переукоренено в среднюю точку с помощью программы retree пакета PHYLIP (выходной файл outtree). Ниже можно увидеть изображение полученного дерева.
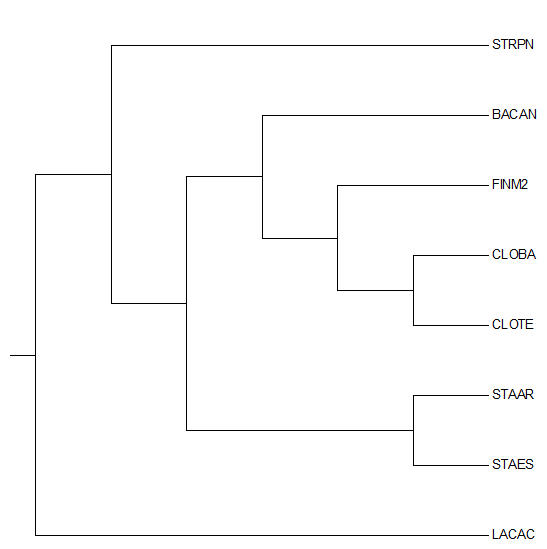
Можно заметить, что дерево было укоренено в нетривиальную ветвь, отделяющую STRPN и LACAC от остальных белков. Данное укоренение можно считать правильным, так как эта ветвь отделяет класс Bacilli, другие ветви отделяют порядки Bacillales, Lactobacillales и Staphylococcaceae.

Бутстрэп анализ
Бутстрэп — практический компьютерный метод исследования распределения статистик вероятностных распределений, основанный на многократной генерации выборок методом Монте-Карло на базе имеющейся выборки[1]. Позволяет просто и быстро оценивать самые разные статистики (доверительные интервалы, дисперсию, корреляцию и так далее) для сложных моделей. Предложен в 1977 году Брэдли Эфроном (первая публикация относится к 1979 году[2]). Суть метода состоит в том, чтобы по имеющейся выборке построить эмпирическое распределение. Используя это распределение как теоретическое распределение вероятностей можно с помощью датчика псевдослучайных чисел сгенерировать практически неограниченное количество псевдовыборок произвольного размера, например, того же как у исходной. На множестве псевдовыборок можно оценить не только анализируемые статистические характеристики, но и изучить их вероятностные распределения. Таким образом, например, оказывается возможным оценить дисперсию или квантили любой статистики независимо от её сложности. Данный метод является методом непараметрической статистики.