Отчет по практикуму 4. Построение и анализ дерева по нуклеотидным последовательностям.
Построение дерева по нуклеотидным последовательностям.
Последовательности 16S рибосомальной РНК каждой из бактерий из практикума были получены из файлов .frn (была выбрана одна копия последовательности 16S рибосомальной РНК одного из штаммов) из базы полных геномов NCBI. Так как последовательности 16S рибосомальной РНК Streptococcus pneumoniae serotype 4 в каталоге не было, то вместо нее была взята последовательность 16S рибосомальной РНК Streptococcus_pneumoniae_CGSP14. Все последовательности были записаны в общий файл 16srna.fasta с соответствующими мнемониками для последовательности каждой бактерии.

Выравнивание в формате mfa
Выравнивание в формате msf
Весь проект
Дерeво в формате Newick
Выравнивание было построено программой Jalview с помощью программы Muscle. Дерево было построено с помощью программой MEGA с помощью метода наибольшего правдоподобия.
Построение и анализ дерева, содержащего паралоги
Выравнивание было построено программой Jalview с помощью программы Muscle. Дерево было построено с помощью программой MEGA с помощью метода наибольшего правдоподобия. Дерево приведено на рис. 2.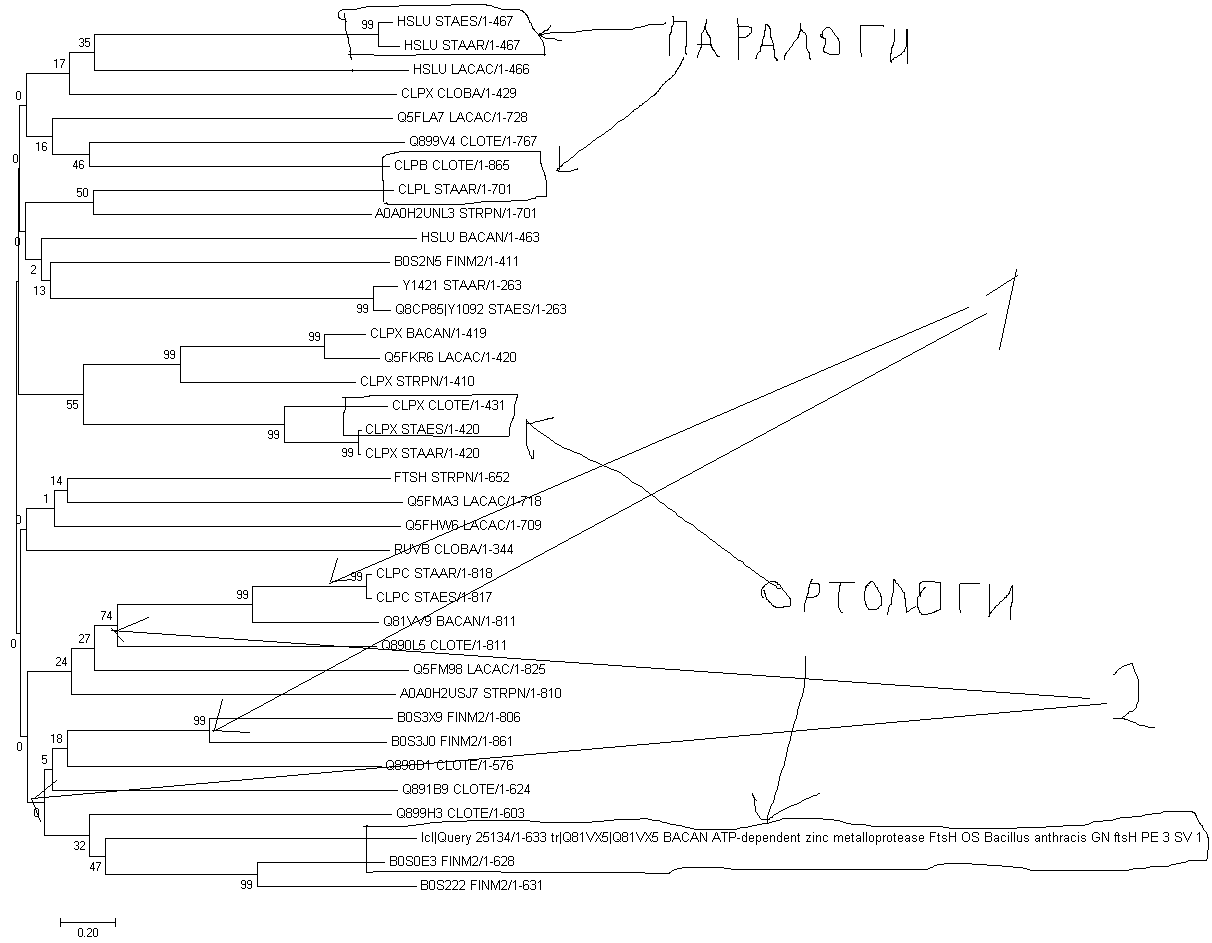
1 - дупликация гена
2- разделение в результате видообразования
Построение и анализ дерева, содержащего паралоги.
Ссылки:
[1] http://lib.stat.cmu.edu/S/bootstrap.funs
[2] Эфрон, 1979.