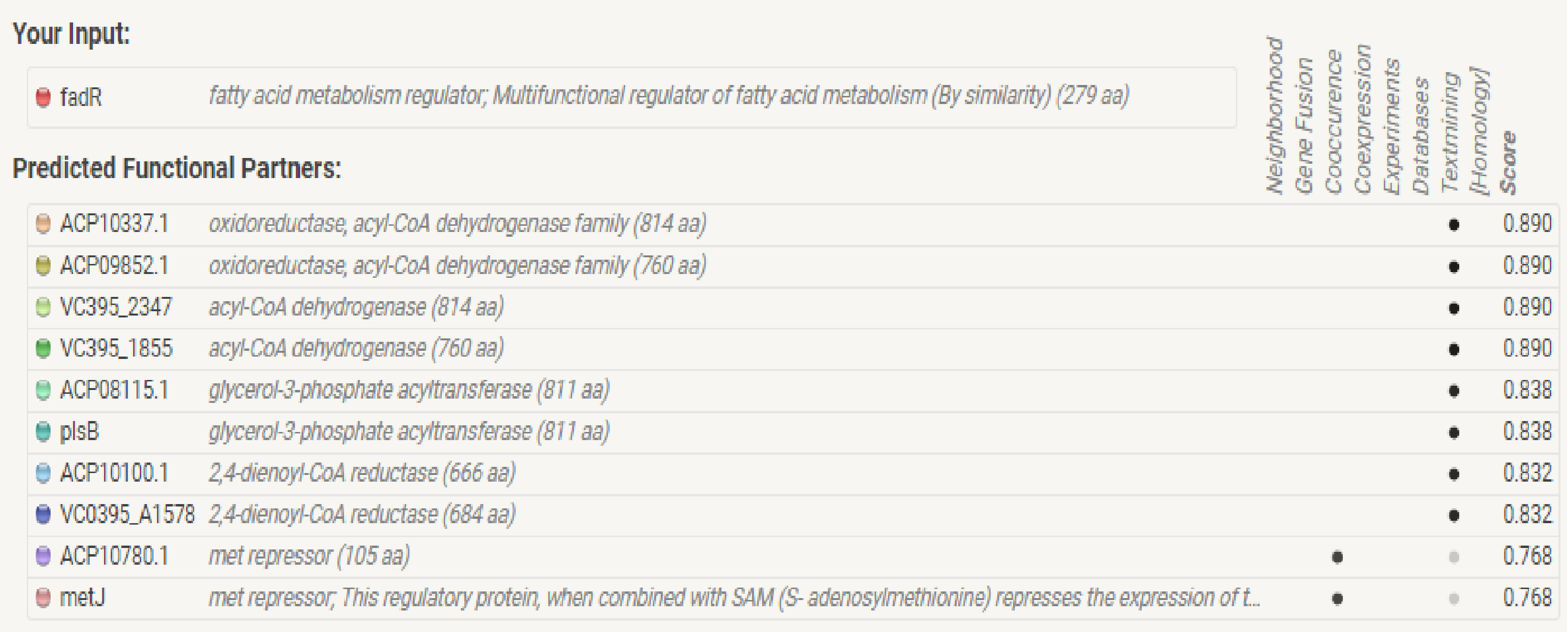
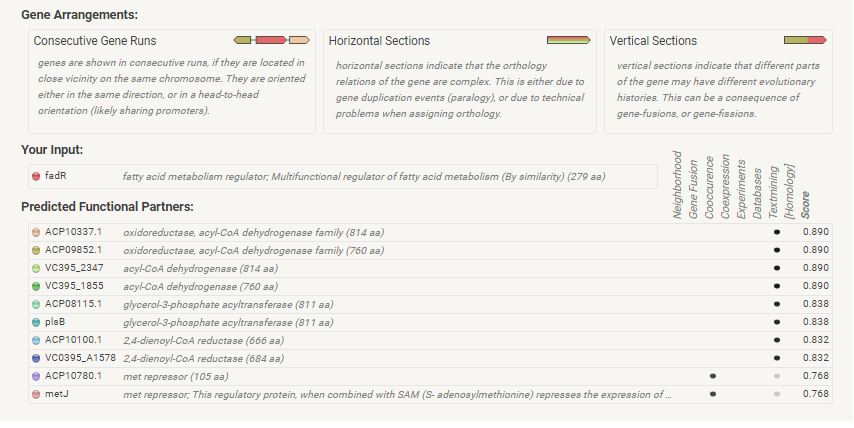
| Аспект | Идентификатор GO | Название термина | Перевод названия термина | Код типа достоверности |
| Биологический процесс (Biological process) | GO:0071072 | negative regulation of phospholipid biosynthetic process | Отрицательная регуляция процесса биосинтеза фосфолипида | IMP |
| Биологический процесс (Biological process) | GO:0019217 | regulation of fatty acid metabolic process | | ISS |
| Биологический процесс (Biological process) | GO:0006355 | regulation of transcription, DNA-templated | | ISS |
| Функция молекулы (Molecular function) | GO:0003700 | transcription factor activity, sequence-specific DNA binding | | ISS |