| Параметр | Белок 4cz8 с альфа-спиралями(Антипортер протонов и одновалентных катионов), 2 цепи | Белок 2erv с бета-тяжами(Липид А 3-О-деацилаза), 1 цепь |
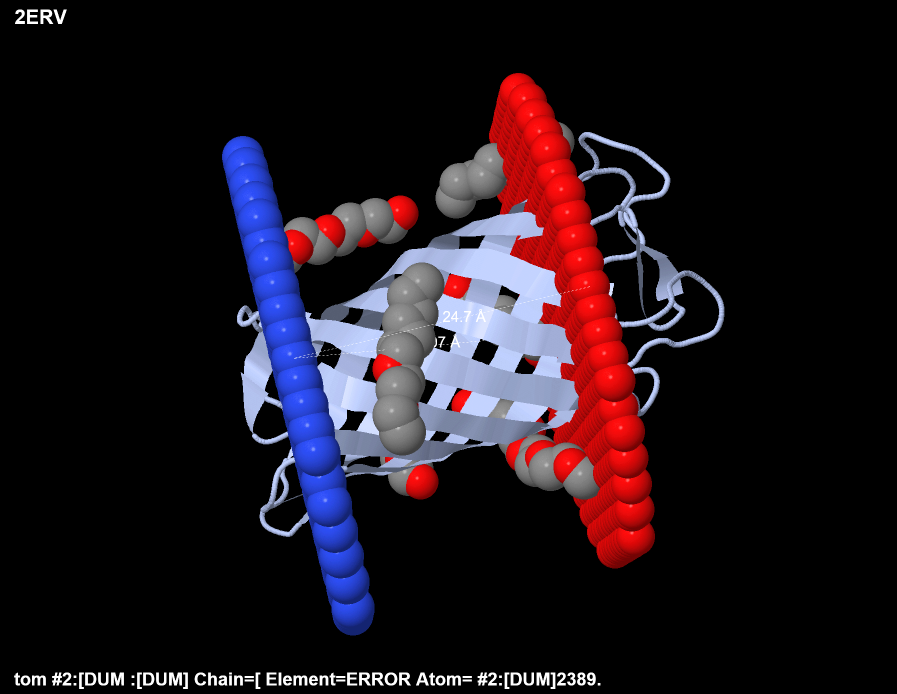
| Толщина гидрофобной части мембраны | 30,07 ангстрем | 24,7 ангстрем |
| Координаты трансмембранных спиралей | A - Tilt: 7° - Segments: 1( 6- 27), 2( 31- 47), 3( 54- 77), 4( 86- 108), 5( 116- 126), 6( 150- 172), 7( 191- 221), 8( 230- 250), 9( 254- 266), 10( 290- 315), 11( 321- 347), 12( 364- 373), 13( 392- 415) B - Tilt: 8° - Segments: 1( 6- 27), 2( 31- 47), 3( 54- 77), 4( 86- 111), 5( 116- 126), 6( 150- 172), 7( 191- 218), 8( 230- 250), 9( 254- 265), 10( 290- 315), 11( 321- 347), 12( 364- 373), 13( 390- 412) | A - Tilt: 19° - Segments: 1(2-9), 2(15-24), 3(37-48), 4(58-70), 5(76-88), 6(102-114), 7(118-127), 8(141-148) |
| Среднее количество остатков в одной трансмембранной спирали белка | 19,77 | 12,5 |
| В какой мембране находится белок | Мембрана архей | Внешняя мембрана Грамм-положительных бактерий |


Prediction of 4CZ8:B|PDBID|CHAIN|SEQUENCE ID 4CZ8:B|PDBID|CHAIN|SEQUENCE FT TOPO_DOM 1 5 NON CYTOPLASMIC. FT TRANSMEM 6 24 FT TOPO_DOM 25 30 CYTOPLASMIC. FT TRANSMEM 31 49 FT TOPO_DOM 50 60 NON CYTOPLASMIC. FT TRANSMEM 61 82 FT TOPO_DOM 83 88 CYTOPLASMIC. FT TRANSMEM 89 110 FT TOPO_DOM 111 115 NON CYTOPLASMIC. FT TRANSMEM 116 138 FT TOPO_DOM 139 158 CYTOPLASMIC. FT TRANSMEM 159 178 FT TOPO_DOM 179 197 NON CYTOPLASMIC. FT TRANSMEM 198 218 FT TOPO_DOM 219 224 CYTOPLASMIC. FT TRANSMEM 225 246 FT TOPO_DOM 247 251 NON CYTOPLASMIC. FT TRANSMEM 252 271 FT TOPO_DOM 272 291 CYTOPLASMIC. FT TRANSMEM 292 311 FT TOPO_DOM 312 316 NON CYTOPLASMIC. FT TRANSMEM 317 340 FT TOPO_DOM 341 351 CYTOPLASMIC. FT TRANSMEM 352 372 FT TOPO_DOM 373 391 NON CYTOPLASMIC. FT TRANSMEM 392 415 FT TOPO_DOM 416 422 CYTOPLASMIC. //
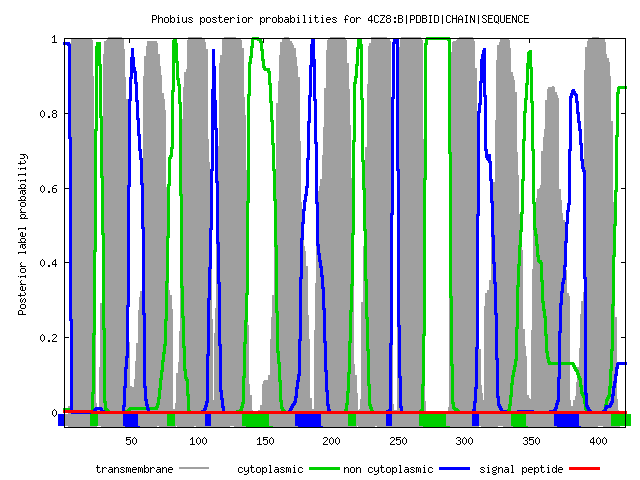
# 4CZ8:B|PDBID|CHAIN|SEQUENCE Length: 422 # 4CZ8:B|PDBID|CHAIN|SEQUENCE Number of predicted TMHs: 13 # 4CZ8:B|PDBID|CHAIN|SEQUENCE Exp number of AAs in TMHs: 270.14936 # 4CZ8:B|PDBID|CHAIN|SEQUENCE Exp number, first 60 AAs: 41.82582 # 4CZ8:B|PDBID|CHAIN|SEQUENCE Total prob of N-in: 0.02899 # 4CZ8:B|PDBID|CHAIN|SEQUENCE POSSIBLE N-term signal sequence 4CZ8:B|PDBID|CHAIN|SEQUENCE TMHMM2.0 outside 1 3 4CZ8:B|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 4 23 4CZ8:B|PDBID|CHAIN|SEQUENCE TMHMM2.0 inside 24 27 4CZ8:B|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 28 50 4CZ8:B|PDBID|CHAIN|SEQUENCE TMHMM2.0 outside 51 59 4CZ8:B|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 60 82 4CZ8:B|PDBID|CHAIN|SEQUENCE TMHMM2.0 inside 83 88 4CZ8:B|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 89 111 4CZ8:B|PDBID|CHAIN|SEQUENCE TMHMM2.0 outside 112 115 4CZ8:B|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 116 138 4CZ8:B|PDBID|CHAIN|SEQUENCE TMHMM2.0 inside 139 158 4CZ8:B|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 159 181 4CZ8:B|PDBID|CHAIN|SEQUENCE TMHMM2.0 outside 182 195 4CZ8:B|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 196 218 4CZ8:B|PDBID|CHAIN|SEQUENCE TMHMM2.0 inside 219 224 4CZ8:B|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 225 247 4CZ8:B|PDBID|CHAIN|SEQUENCE TMHMM2.0 outside 248 251 4CZ8:B|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 252 271 4CZ8:B|PDBID|CHAIN|SEQUENCE TMHMM2.0 inside 272 291 4CZ8:B|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 292 311 4CZ8:B|PDBID|CHAIN|SEQUENCE TMHMM2.0 outside 312 325 4CZ8:B|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 326 348 4CZ8:B|PDBID|CHAIN|SEQUENCE TMHMM2.0 inside 349 354 4CZ8:B|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 355 377 4CZ8:B|PDBID|CHAIN|SEQUENCE TMHMM2.0 outside 378 391 4CZ8:B|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 392 414 4CZ8:B|PDBID|CHAIN|SEQUENCE TMHMM2.0 inside 415 422

| Номер | Q9UZ55 |
| Название белка | Na+/H+ antiporter, putative |
| Длина | 443 |
| Молекулярная масса | 49188.00 |
| Виды, у которых есть этот белок | Pyrococcus abyssi (strain GE5 / Orsay) [272844] |
| Количество TMS | 13 |
| Субстрат | Na+, H+ |