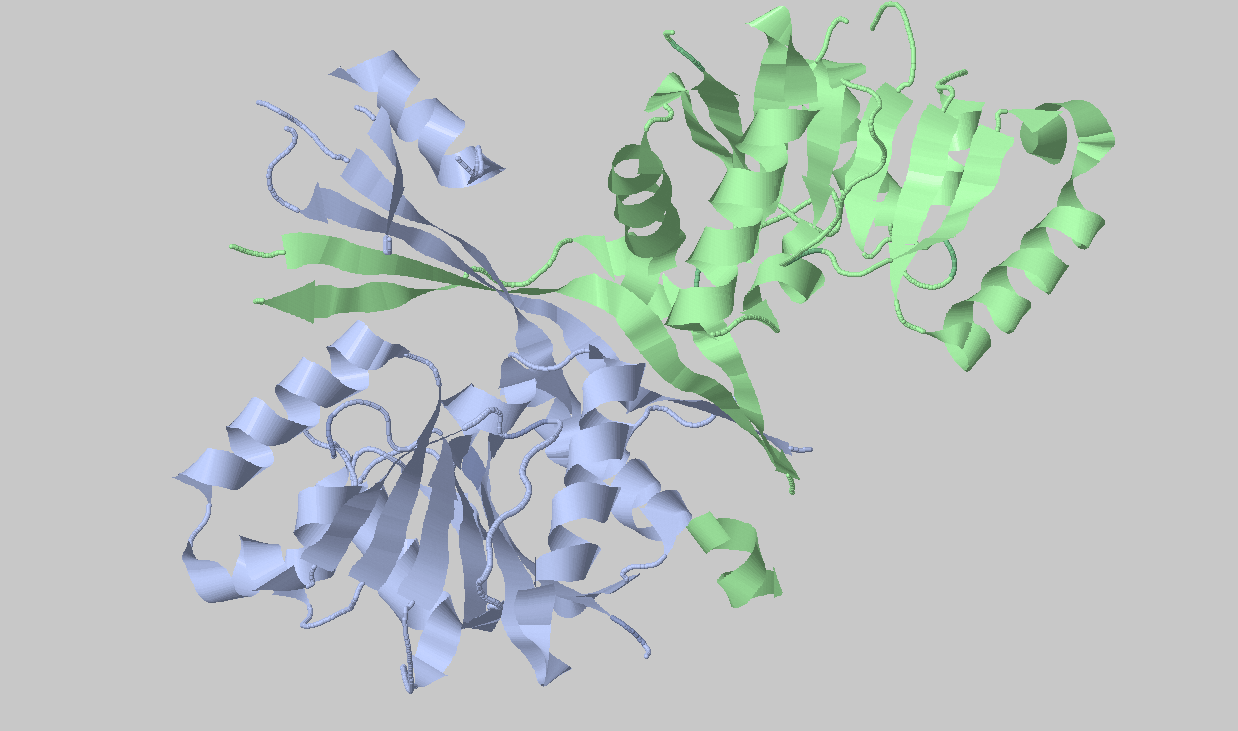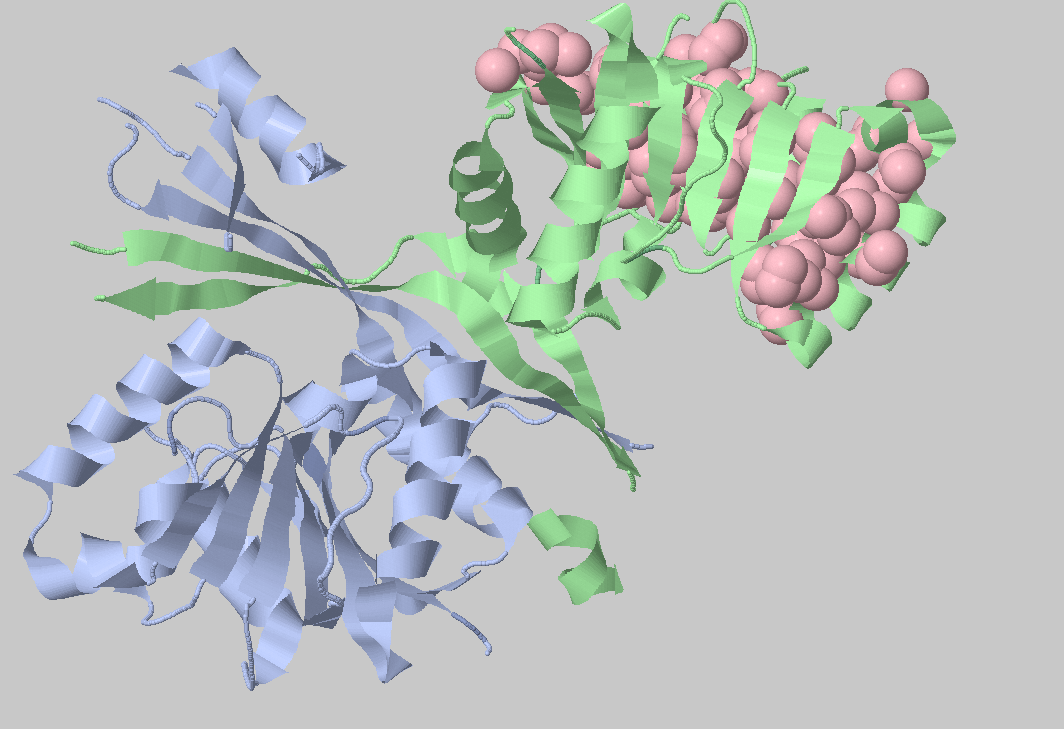Практикум 11. Гидрофобные кластеры.
На этой странице выложен отчет по практикуму 11. Гидрофобные кластеры.
1. Гидрофобные кластеры в структуре гомодимера.
Для практикума был взят белок с идентификатором 3g2m. Это гомодимер N-метилтрансферазы. Был использован сервис Clud. Для начала сервис был запущен со стандартными параметрами.
Эти параметры составляют: порог размера кластера 3 атома и порог расстояния - 5,4 ангстрем.
В результате были получены 14 кластеров, 11 из которых были небольшими по размеру, а 3 были значительными.
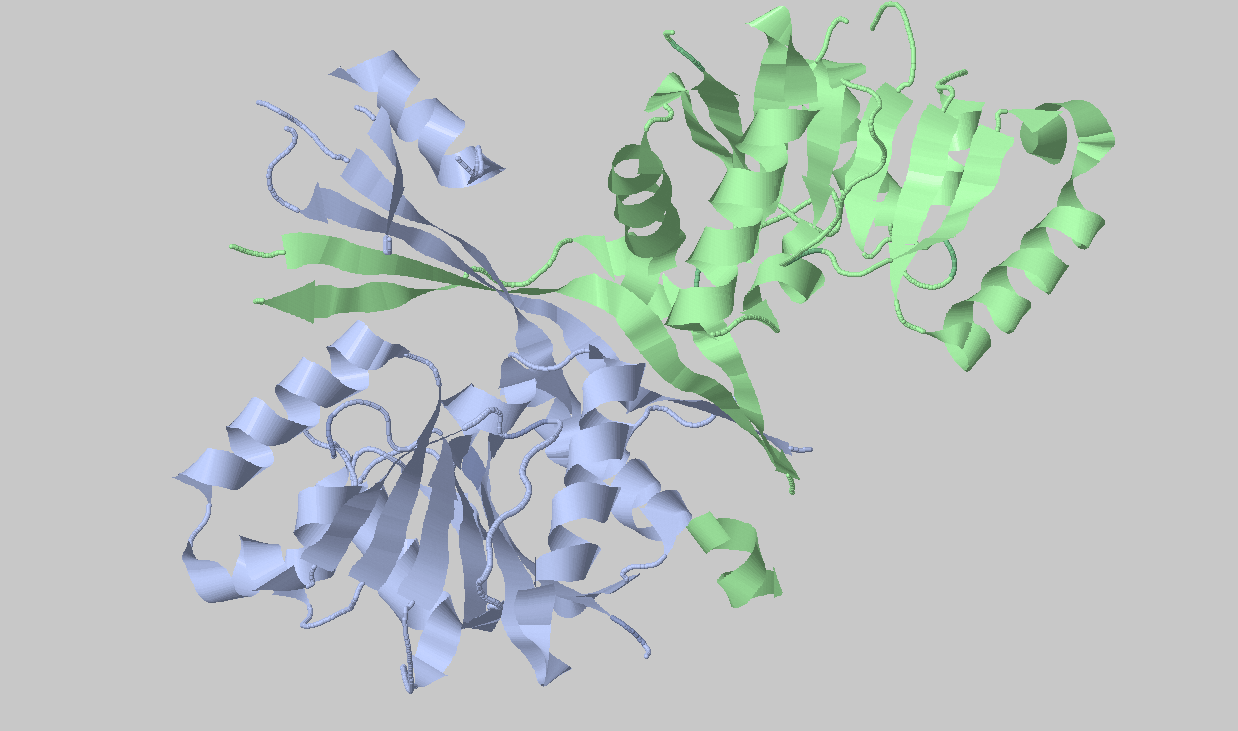
Рис. 1. Гомодимер в Jmol.

Рис. 2. Кластер 1 размером 460 атомов.

Рис. 3. Кластер 2 размером 139 атомов.
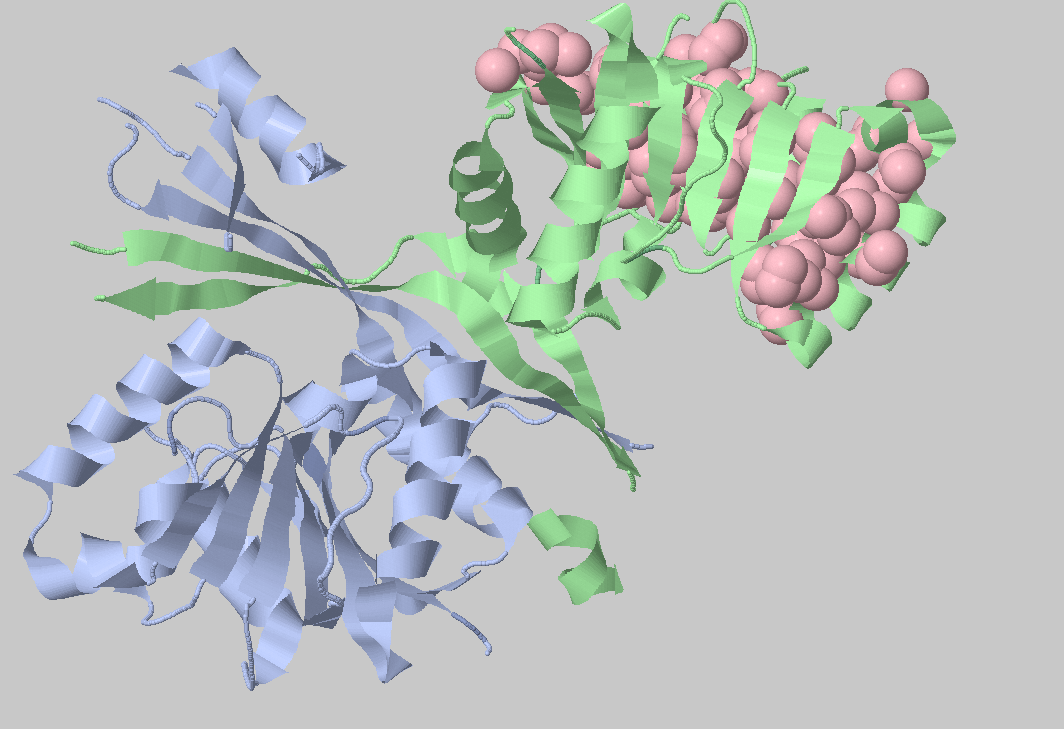
Рис. 4. Кластер 3 размером 135 атомов.
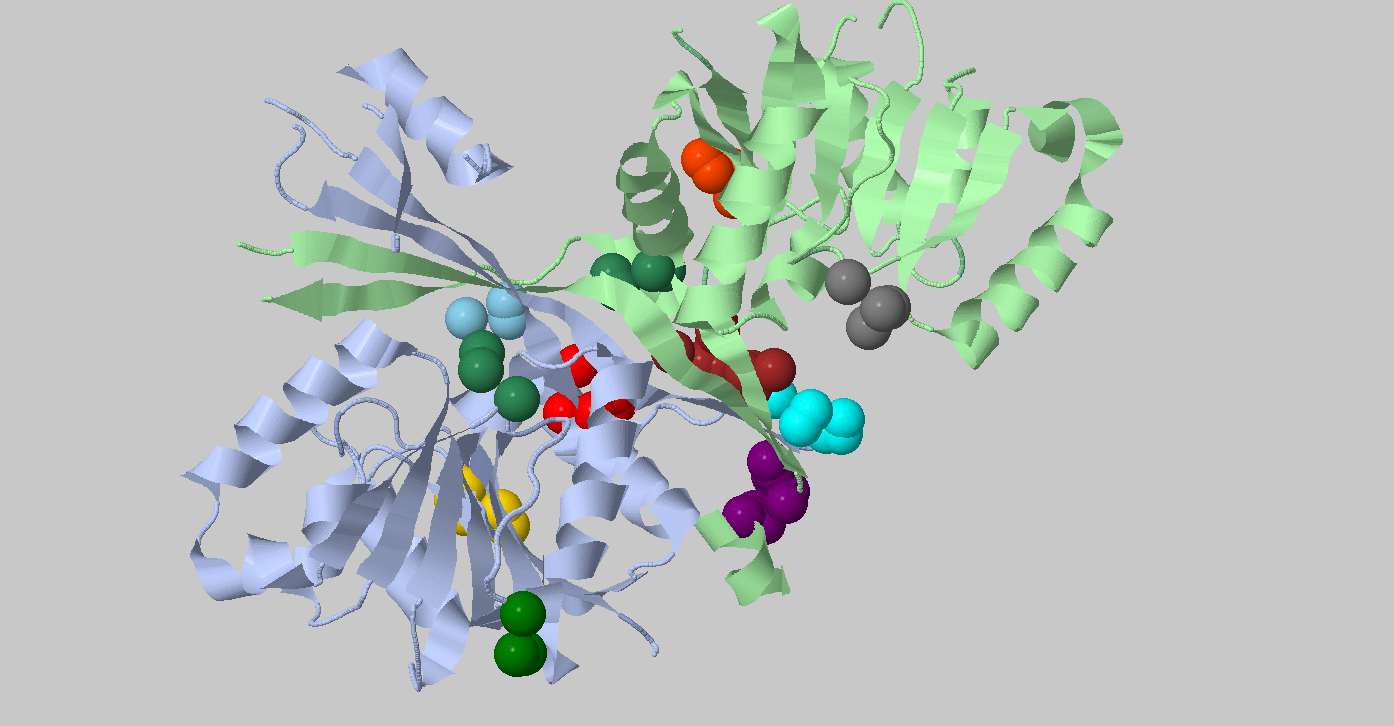
Рис. 5. Небольшие кластеры размером от 3 до 11 атомов.
Можно заметить, что наиболее крупный гидрофобный кластер охватывает димер практически полностью. При этом нужно упомянуть, что он охватывает в основном бета-листы димера.
Размер этого кластера составляет 460 атомов. Рассмотрим второй и третий по размеру кластеры.
Они соответствуют доменам в каждом мономере димера.
Если проанализировать более маленькие кластеры, то они небольшого размера по сравнению с тремя упомянтыми выше. Поэтому скорее всего можно сделать вывод, что они не имеют биологического значения.
2. Гидрофобные кластеры на интерфейсе двух цепочек в структуре гомодимера.
Для анализа контакта двух димеров был использован поиск на интерфейсе двух цепочек, то есть между цепей. Для анализа был выбран ДНК-белковый комплекс 1ynw.
Были обнаружены гидрофобные взаимодействия
между ДНК и белком. С использованием сервиса был наден один гидрофобный кластер.
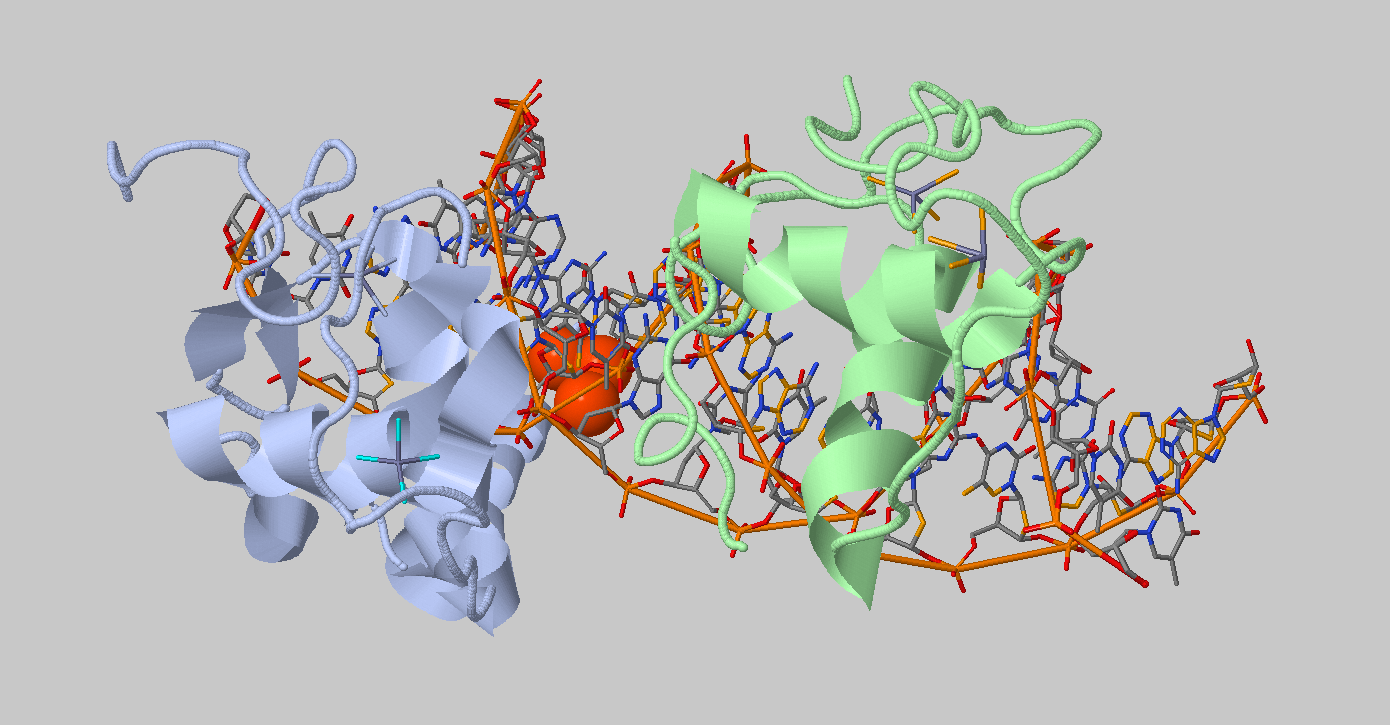
Рис. 6. Гидрофобный кластер ДНК-белкового комплекса 1ynw.
Ссылки:
[1] Hodge T. & Cope M.J.T.V. 2000. A myosin family tree. Journal of Cell Science 113:3353-3354.
[2] Briggs G.S., Smits W.K., Soultanas P. (2012). Chromosomal Replication Initiation Machinery of Low-G+C-Content Firmicutes. J Bacteriol. 194, 5162-70.
[3] Semova, I., Carten, J.D., Stombaugh, J., Mackey, L.C., Knight, R., Farber, S.A., and Rawls, J.F. (2012). Microbiota Regulate Intestinal Absorption and Metabolism of Fatty Acids in the Zebrafish. Cell Host Microbe 12, 277-288.
[4] Backhed, F., Ding, H., Wang, T., Hooper, L.V., Koh, G.Y., Nagy, A., Semenkovich, C.F., and Gordon, J.I. (2004). The gut microbiota as an environmental factor that regulates fat storage. Proc. Natl. Acad. Sci. USA 101, 15718–15723.