Практикум 1. Восстановление кристалла из PDB файла.
На этой странице выложен отчет по практикуму 2. Восстановление кристалла из PDB файла.
1. Взаимодействие белка с идентификатором 3o48 с субъединицами из соседних ячеек.
Требования к белку:
1) Модель сделана рентгеноструктурным анализом. Со страницы структуры на сайте PDBe должен быть доступен для скачивания файл с электронной плотностью.
2) Наличие файла с экспериментальными данными РСА.
3) В доступе имеется статья, которая была опубликована по итогам расшифровки.
Разрешение 1.75A.
Длина полипептидной цепи 134 а.о.
4) Имеются подходящие структуры белков, сходных одним из белков вашей модели.
Был выбран белок 3o48.
Информация о параметрах элементарной ячейки содержится в строке:
CRYST1 46.020 46.020 139.220 90.00 90.00 90.00 P 43 21 2 8
У нас есть длины направляющих векторов: 46.020 46.020 139.220 и соответствующие углы: 90.00 90.00 90.00.
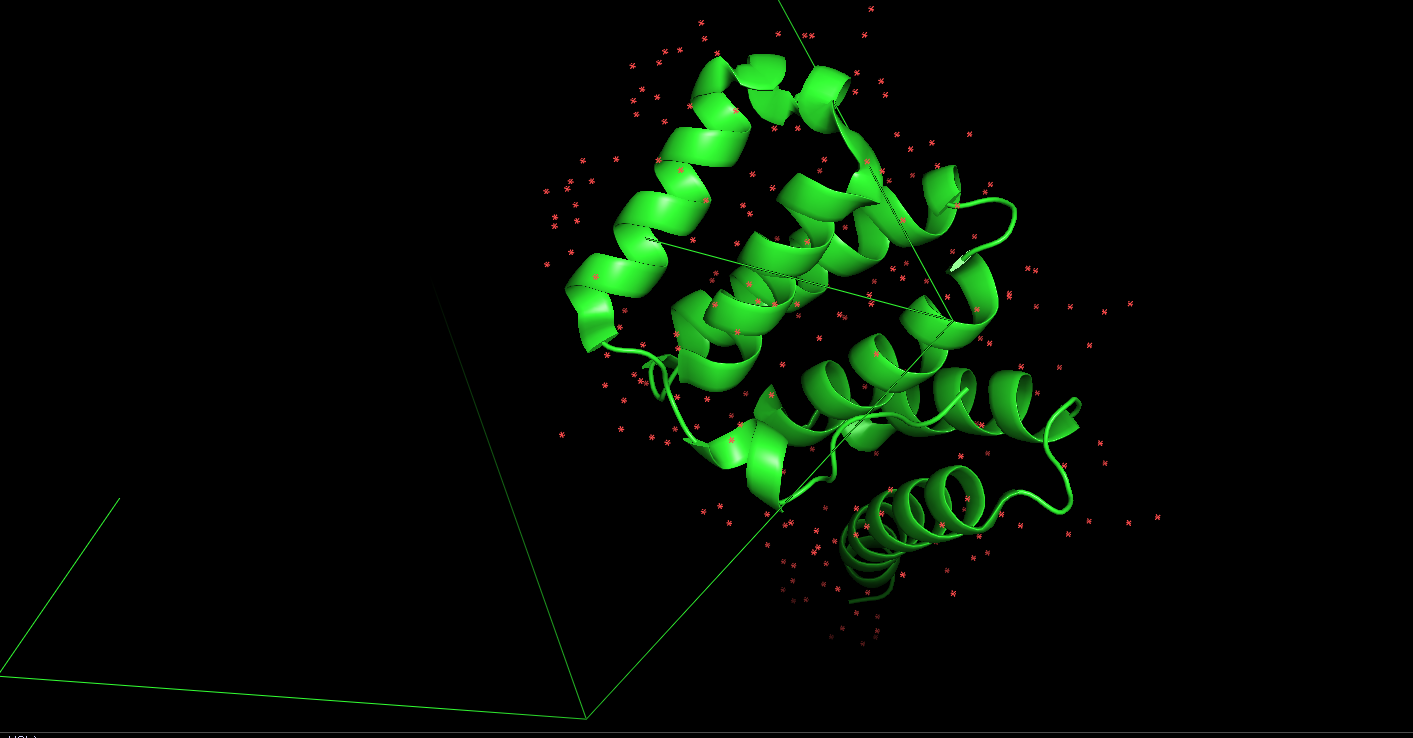
Рис. 1. Ячейка белка 3o48 в Pymol.

Рис. 2. Регулярные структуры на расстоянии не более 3 ангстрем. Изображение в программе Pymol.
Далее исследуем зоны контакта с белками из соседних ячеек. Нужно заметить, что в этом белке ни одна из молекул не входит в ячейку полностью -
каждая молекула находится не менее чем в двух ячейках. Посмотрим н контакты белка с соседними молекулами.
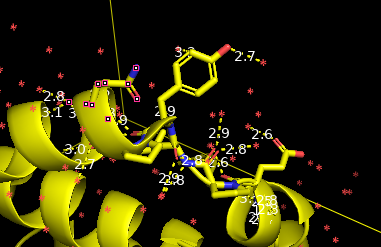
Рис. 3. Водородные связи белка 3o48 в Pymol.
Белок является мономером. Из проведенного анализ становиться ясно, что контакты являются артефактами кристаллизации и биологического значения не имеют.
2. Странное положение цепей белка.
В данном случае можно заметить, что эта структура имеет странность, которая заключается в том, что белок взаимодействует не с бороздками или сахарофосфатным остовом.
Белок взаимодействует с концом ДНК, что видно на Рис. 4.
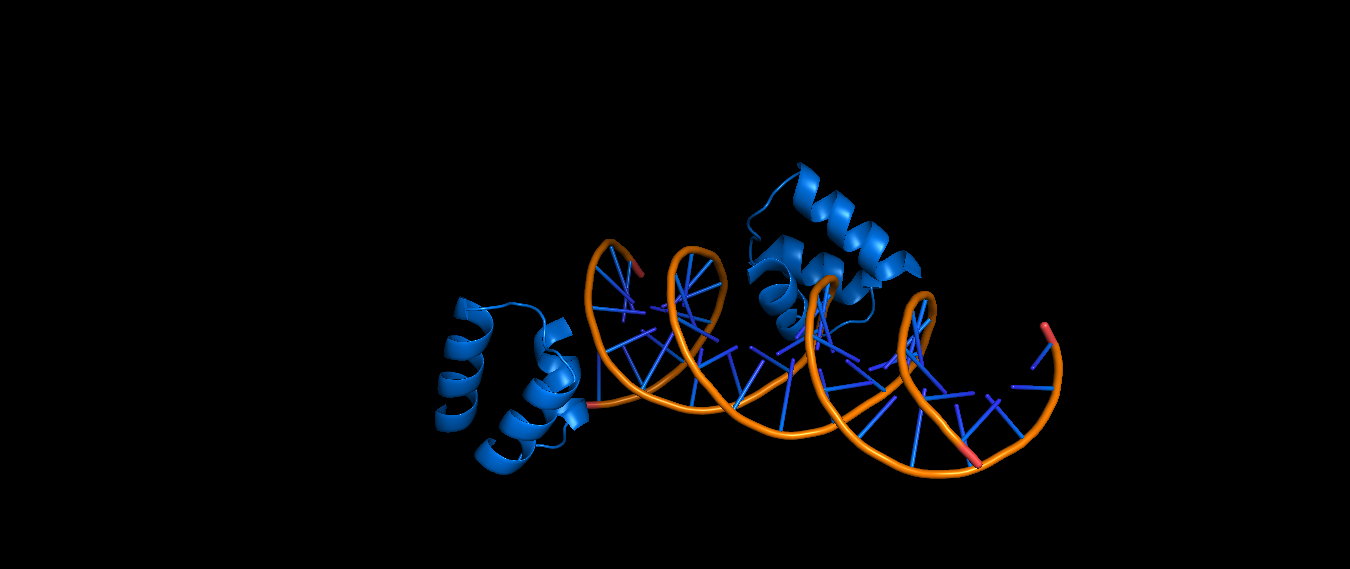
Рис. 4. Ячейка белка 3o48 в Pymol.
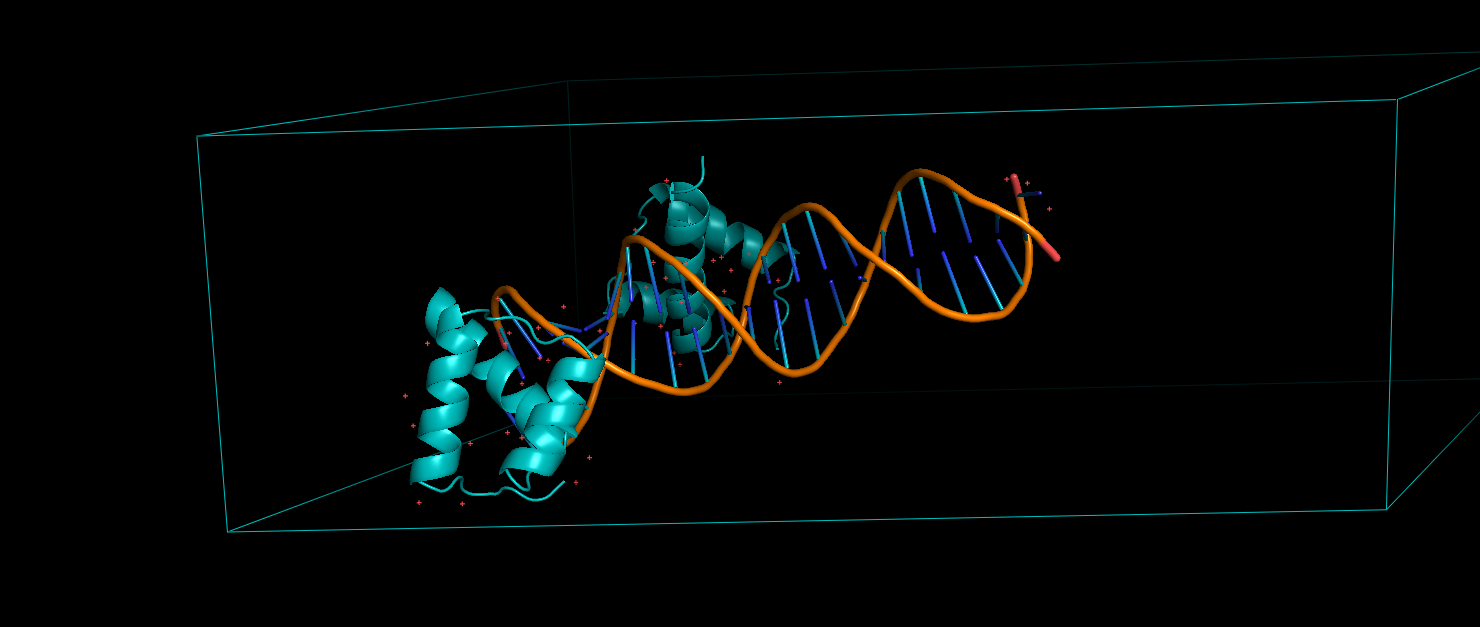
Рис. 5. Ячейка белка 3o48 в Pymol.
Ссылки:
[1] Hodge T. & Cope M.J.T.V. 2000. A myosin family tree. Journal of Cell Science 113:3353-3354.
[2] Briggs G.S., Smits W.K., Soultanas P. (2012). Chromosomal Replication Initiation Machinery of Low-G+C-Content Firmicutes. J Bacteriol. 194, 5162-70.
[3] Semova, I., Carten, J.D., Stombaugh, J., Mackey, L.C., Knight, R., Farber, S.A., and Rawls, J.F. (2012). Microbiota Regulate Intestinal Absorption and Metabolism of Fatty Acids in the Zebrafish. Cell Host Microbe 12, 277-288.
[4] Backhed, F., Ding, H., Wang, T., Hooper, L.V., Koh, G.Y., Nagy, A., Semenkovich, C.F., and Gordon, J.I. (2004). The gut microbiota as an environmental factor that regulates fat storage. Proc. Natl. Acad. Sci. USA 101, 15718–15723.




