1. Поиск структурных гомологов.
В этой работе использовался PDBeFold. Использовалась структура 4r77. Для этой структуры было найдено 214 находок.
Были выбраны две структуры разных белков с RMSD между 0,8 и 2,5 и длиной выравнивания более 50% от длины.
Были выбраны структуры 4r78:A, 2ig7:A, 2ckq:A и 2qg7:A. Совмещение структур показано на Рис. 1.
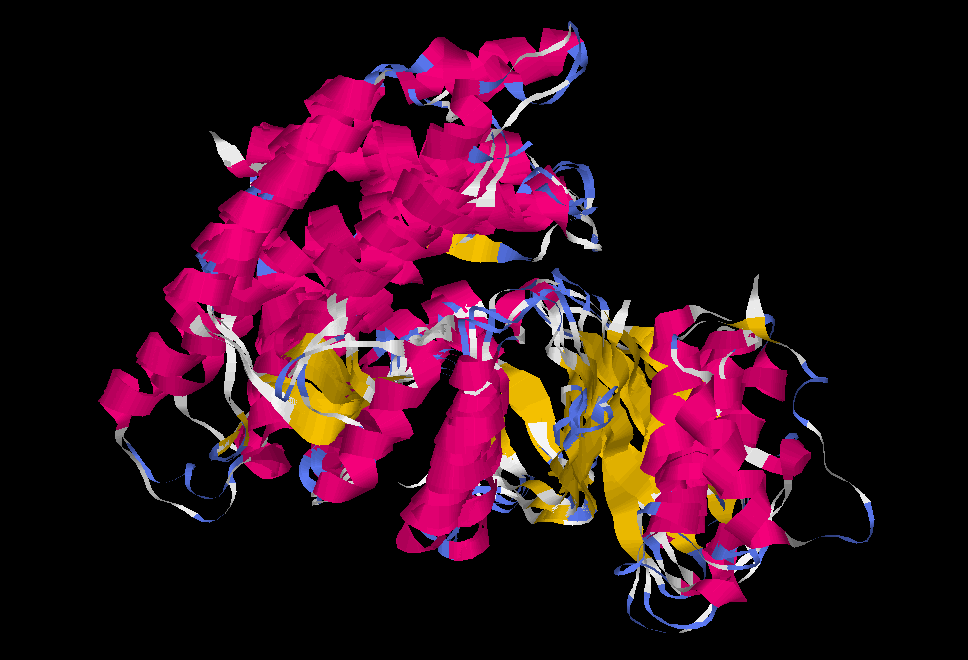
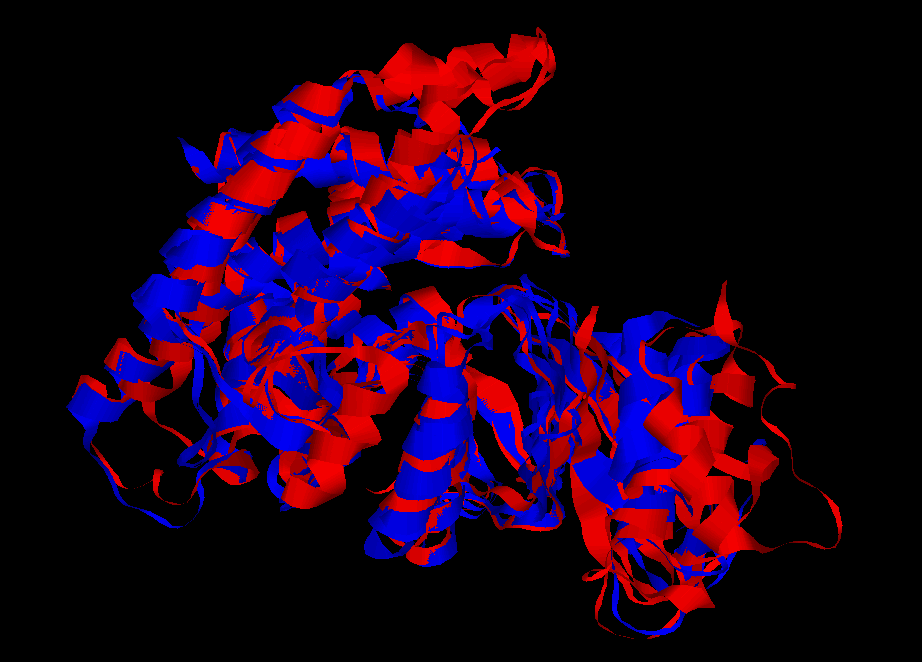
Ниже представлено множественное выравнивание.
Также был скачан файл с совмещением структур. Кроме этого множественное выравнивание скачано.
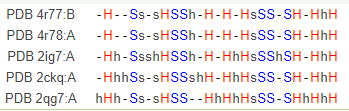
Затем было построено множественное выравнивание выбранных белков с помощью программы Jalview. Для построения выравнивания был использован алгоритм Muscle. Проект, содержащий два данных выравнивания. На Рис. 3 и Рис. 4 представлены фрагменты обоих множественных выравниваний.
Рис. 4. Структурное выравнивание.

Рис. 5. Множественное выравнивание.

Также можно привести весь проект. Нужно заметить, что эти белки являются довольно небольшими. Можно предположить, что эти белки довольно гомологичны и оба выравнивания достаточно похожи.
2. Различия в выравниваниях.
Приведем участки этих двух выравниваний, которые отличаются.
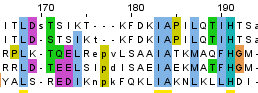
Рис. 6. Фрагмент структурного выравнивания.
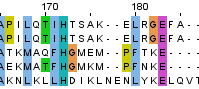
Рис. 7. Фрагмент множественного выравнивания.
Заметим, что структурное выравнивание(построенное на основании структур) помещает остаток изолейцина пятой последовательности 2qg7 в 43 позицию выравнивания с двумя лизинами и аргинином, в то время как в множественном выравнивании(построенном на основании последовательностей) на это место (к лизинам и аргининам) помещен лизин. Рассмотрим структурное выравнивание. Четыре остатка(2 лизина и два аргинина) выделены желтым, исследуемый лизин - белым.

Рис. 8. Структурное выравнивание, отображаемое cartoon.
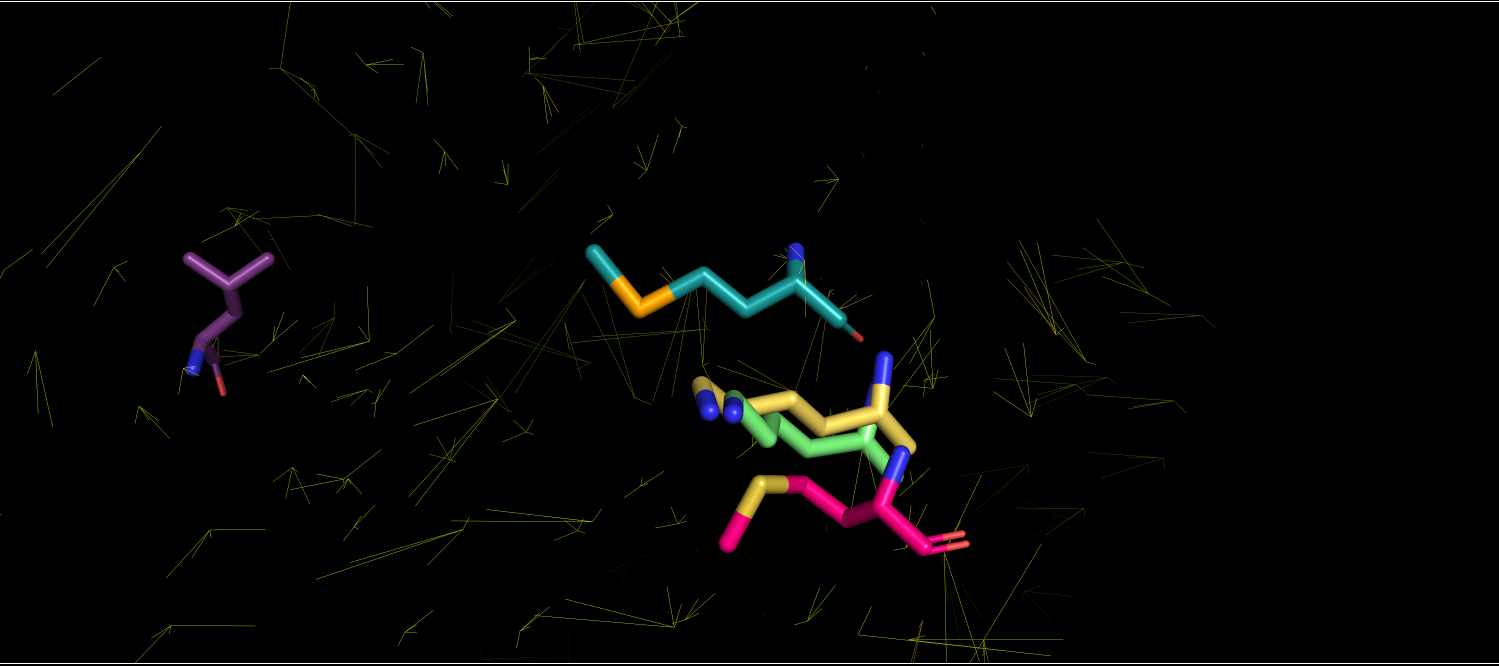
Рис. 9. Структурное выравнивание, отображаемое sticks.
Сравнивая как идет структура мы видим, что в структуре лизин пятой структуры относится совершенно к другой вторичой структуре(к бета листу), чем четыре остатка(относятся к альфа-спирали), поэтому помещать их в одну колонку выравнивания неразуино и структурное выравнивание, полученное с помощью PDBeFold скорее более правдоподобно.
Ссылки:
[1] Hodge T. & Cope M.J.T.V. 2000. A myosin family tree. Journal of Cell Science 113:3353-3354.
[2] Briggs G.S., Smits W.K., Soultanas P. (2012). Chromosomal Replication Initiation Machinery of Low-G+C-Content Firmicutes. J Bacteriol. 194, 5162-70.
[3] Semova, I., Carten, J.D., Stombaugh, J., Mackey, L.C., Knight, R., Farber, S.A., and Rawls, J.F. (2012). Microbiota Regulate Intestinal Absorption and Metabolism of Fatty Acids in the Zebrafish. Cell Host Microbe 12, 277-288.
[4] Backhed, F., Ding, H., Wang, T., Hooper, L.V., Koh, G.Y., Nagy, A., Semenkovich, C.F., and Gordon, J.I. (2004). The gut microbiota as an environmental factor that regulates fat storage. Proc. Natl. Acad. Sci. USA 101, 15718–15723.