1. Примеры атомов с коэффициентом заполнения (Occupancy), не равным 1.
был произведен поиск с помощью advanced search по структурам с разрешением (в ангстремах) менее 1 и выполненные в течение последних лет. Была найдена структура 3UC7(белок Trp-cage cyclo-TC1), в которой в части ячеек атом имеет одни координаты, в части - другие. Пример в PDB представлен ниже.
ATOM 62 CB ALEU A 7 15.332 -1.834 -10.034 0.50 13.53 C
ANISOU 62 CB ALEU A 7 1388 1717 2035 -549 146 209 C
ATOM 63 CB BLEU A 7 15.498 -1.760 -9.925 0.50 9.23 C
ANISOU 63 CB BLEU A 7 381 1369 1755 -269 -279 -112 C
ATOM 64 CG ALEU A 7 15.775 -1.369 -8.694 0.50 20.39 C
ANISOU 64 CG ALEU A 7 3144 2338 2262 -1313 -452 418 C
ATOM 65 CG BLEU A 7 14.879 -0.821 -8.946 0.50 12.24 C
ANISOU 65 CG BLEU A 7 1329 1747 1572 -330 -26 -38 C
ATOM 66 CD1ALEU A 7 15.841 -2.598 -7.799 0.50 13.30 C
ANISOU 66 CD1ALEU A 7 1265 2243 1544 -609 243 324 C
ATOM 67 CD1BLEU A 7 14.332 -1.685 -7.765 0.50 13.34 C
ANISOU 67 CD1BLEU A 7 357 2434 2275 -139 120 572 C
ATOM 68 CD2ALEU A 7 17.034 -0.470 -8.584 0.50 13.65 C
ANISOU 68 CD2ALEU A 7 683 2896 1607 -27 -242 370 C
ATOM 69 CD2BLEU A 7 15.915 0.141 -8.467 0.50 11.64 C
ANISOU 69 CD2BLEU A 7 750 1981 1690 -227 -78 140 C
Далее мы визуализируем этот остаток с помощью Pymol. Остаток выделен желтым.

Рис. 1. Остаток в Pymol.
2. Не расшифрованные аминокислотные остатки (Missing residues).
Далее был осуществлен поиск структур низкого разрешения (более 3 ангстрем). Была найдена структура 4ZJD(белок теплового шока AgsA из Salmonella typhimurium). Всего 264 не расшифрованных остатка. Вероятно эти остатки являются достаточно подвижными, возможно расположены на поверхности белковой глобулы.
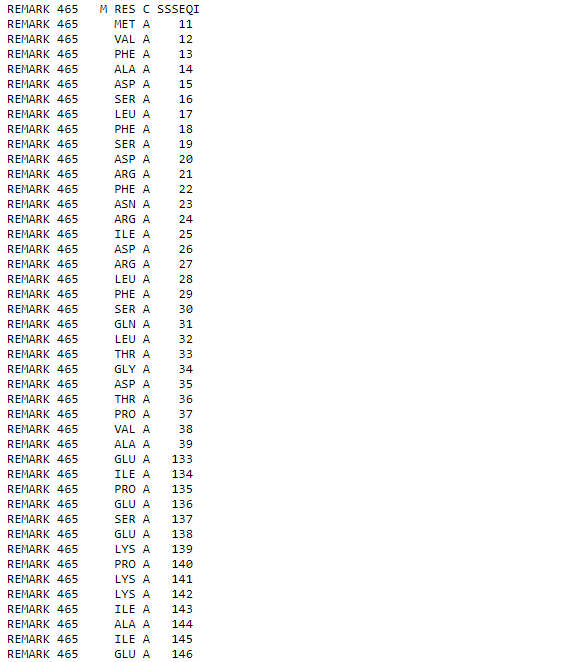
Рис. 2. Примеры не расшифрованных остатков.
3. Несовпадение последовательностей белка.
C PDB структурой белка связаны три последовательности: (1) последовательность природного белка из Uniprot; (2) последовательность белка, который кристаллизовали — он может отличаться наличием тэгов, или же быть частью природного белка, например, доменом; (3) та часть последовательности (2), которая соответствует "видимой" методом РСА (то есть одинаково расположенной во всех ячейках кристалла) части белка. С использованием advanced search был произведен поиск по разделу sequence features с помощью опции wild type protein. Был найден белок 3GJC В файле поле DBREF содержит имя белка в банке Uniprot, поле SEQADV - последовательность изменения относительно природного белка. Для этого белка имеются изменения относительно природного белка.
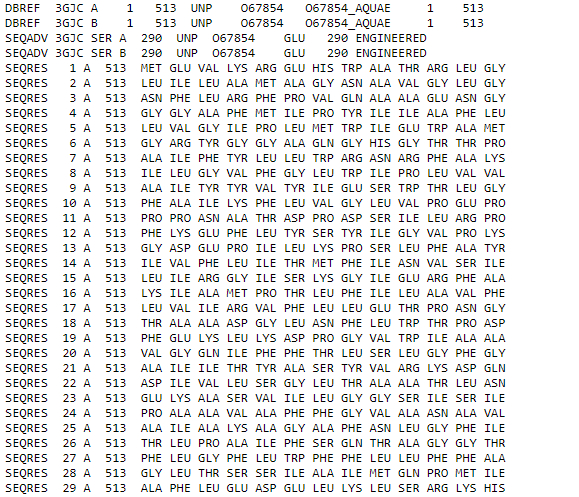
Рис. 3. Поля DBREF и SEQADV.
Также есть нерасшифрованные остатки.
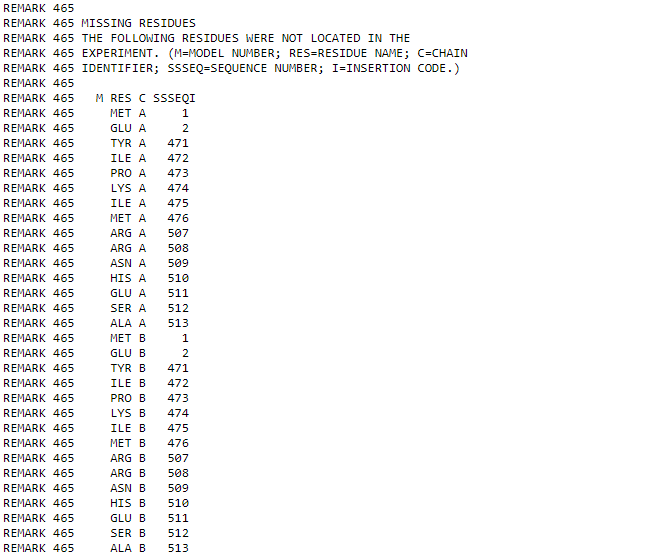
Рис. 4. Примеры не расшифрованных остатков.
4. Наибольший и наименьший B-фактор.
Рассмотрим PDB структуру 3GJC, рассмотренную в третьем пункте. B-фактор описывает насколько хорошо расссматриваемый атом вписывается в предсказанную электронную плотность. Соответственно, низкое значение B-фактора означает, что что координаты атома в структуре могут быть определены достаточно точно.
ATOM 3627 CA VAL A 465 4.081 17.682 9.168 1.00 86.96 C
ATOM 4516 CG2 THR B 77 -9.537 2.599 -39.542 1.00 14.57 C
Выше представлены примеры с наибольшим и наименьшим значениями B-фактора. Случай с большим значением говорит, что атом возможно имеет большую подвижность, что ведет к большей делокализации электронной плотности, что приводит к тому, что его координаты определены с меньшей точностью.

Рис. 5. Остаток с высоким значением B-фактора в Pymol.
Высокое значение B-фактора обусловлено тем, что остаток находится на поверхности белковой глобулы и обращен в растворитель.
Рис. 6. Остаток с низким значением B-фактора в Pymol.
Остаток с низким значением B-фактора, наоборот, находится внутри белковой глобулы и поэтому его подвижность ограничена(в том числе и стерически, поскольку остатки расположены довольно плотно внутри глобулы).
Ссылки:
[1] Hodge T. & Cope M.J.T.V. 2000. A myosin family tree. Journal of Cell Science 113:3353-3354.
[2] Briggs G.S., Smits W.K., Soultanas P. (2012). Chromosomal Replication Initiation Machinery of Low-G+C-Content Firmicutes. J Bacteriol. 194, 5162-70.
[3] Semova, I., Carten, J.D., Stombaugh, J., Mackey, L.C., Knight, R., Farber, S.A., and Rawls, J.F. (2012). Microbiota Regulate Intestinal Absorption and Metabolism of Fatty Acids in the Zebrafish. Cell Host Microbe 12, 277-288.
[4] Backhed, F., Ding, H., Wang, T., Hooper, L.V., Koh, G.Y., Nagy, A., Semenkovich, C.F., and Gordon, J.I. (2004). The gut microbiota as an environmental factor that regulates fat storage. Proc. Natl. Acad. Sci. USA 101, 15718–15723.