Нуклеотидный BLAST
Задание 1
Мой контиг – фрагмент первой хромосомы организма c 6367791 по 6371614 нуклеотид (NC_047126.1). Длинна контига – 3824по, он содержит ген транскрипционного фактора ATF4, состоящий из 4 экзонов (зеленые прямоугольные участки), 3 экзона содержат CDSs (темно-зеленые части экзонов), светло-зеленые участки экзонов представляют собой нетранслируемые области мРНК.
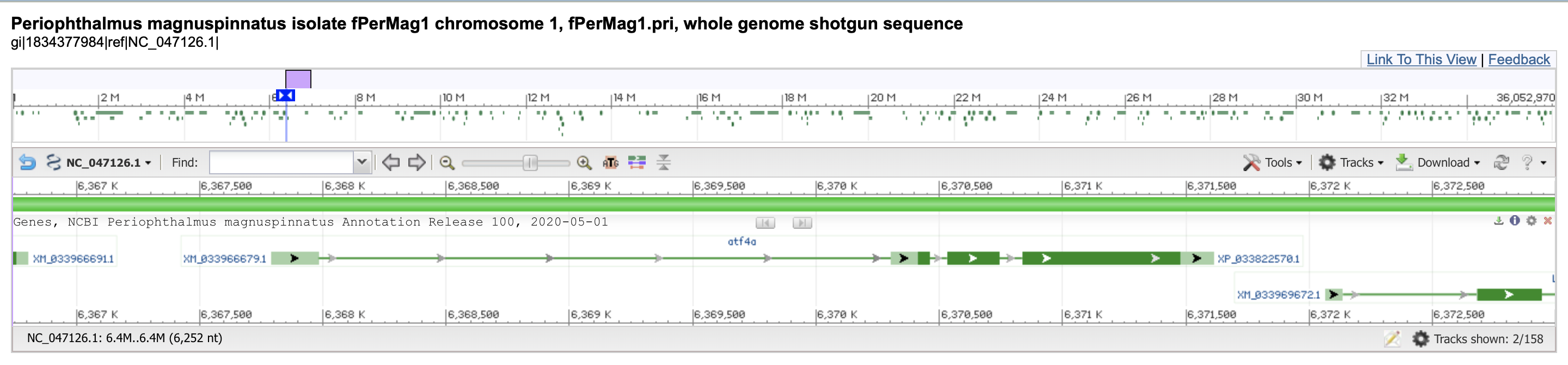
Рис 1. ген, содержащийся в контиге
1. Blastp: Исключаю таксон Хордовые - Chordata (taxid7711).В результате работы программы получаю 104 находки, большАя часть которых –"Predicted" и синтетические конструкции эукариот (Напр. Eukaryotic synthetic construct chromosome 22 или Synthetic construct Homo sapiens clone ccsbBroadEn_00117). Blastp - алгоритм, который принимает на вход нуклеотидную последовательность и ищет похожие последовательности из нуклеотидной базы данных. Его применяют когда нужно найти похожие (в перспективе - гомологичные последовательности ДНК или РНК.
2. Megablast: так как эта программа используется для поиска очень похожих последовательностей результат возможно получить только на уровне исключения отряда Gobiiformes (taxid:1489878). Получено 15 находок, из которых 11 Predicted все находки это ATF4 или ATF4-like транскрипционные факторы. Megablast - вариант blastp, осуществляющий поиск очень похожих последовательностей.
3. Blastx: Исключаю таксон Хордовые - Chordata (taxid7711). Всего получено 530 находок – большинство находок являются неохарактеризованными белками, либо ATF4, кроме того, среди находок есть транскрипционный фактор 5 этого же семейства (ATF5). В отличии от blastp, blastx транслирует полученную нуклеотидную последовательность и ищет похожие аминокислотные последовательности в белковой базе данных.
4. tblastx также транслирует полученный запрос в белок и ищет схожие в нуклеотидной базе данных, транслированной в шести рамках считывания. Для своего контига не получил ни одной находки при ограничении таксона вплоть до семейства.
Задание 2
Последовательности генома организма была проиндексированна командой:
makeblastdb -in PerMag.fna -dbtype nucl
Локальный поиск BLAST для каждой рРНК по базе данных, полученной в результате выполнения команды выше проводился с помощью команд:
blastn -task blastn -query 16s_rRNA_ecoli.fna -db PerMag.fna -out blastp_1.out -evalue 0.05 -word_size 4
blastn -task blastn -query 23s_rRNA_ecoli.fna -db PerMag.fna -out blastp_2.out -evalue 0.05 -word_size 4
16s rRNA – играет роль скэффолда для рибосомальных белков малой субъединицы, необходима ей при сканировании 5’-UTR mRNA для обнаружения старт-кодона рядом с консенсусной последовательностью Шайна-Дальгарно.
23s rRNA - локализуется в центральном протуберанце большой субъединицы рибосомы, в действительности, являясь рибозимом, катализирует пептидилтрансферазную реакцию.
В результате локальног поиска BLAST для 16s rRNA были найдены эукариотические гомологи этой рРНК у эукариот - 18s рРНК в 13 и 17 хромосомах, в 16 хромосоме была найдена альфа-7-подобная субъединица ацетилхолинового рецептора нейронов, а в 14 хромосоме - IPPE-подобный белок 3.
Для 23s рРНК почти все найденные последовательности являются либо неанотированными участками хромосом либо, псевдогенами. Из интересного, были найденны: связанный с G-белком B2-подобный рецептор в 11 хромосоме, дзета-субъединица ДНК полимеразы в 24 хромосоме, чувствительный к мРНК (управляемый протонами) ионный канал 1b, транскрипционный вариант X1 в 5 хромосоме, регуляторная субъединица 37 протеинфосфатазы 1. Кроме того была установленна гомология с 16s рРНК в митохондриальном геноме (для 16s рРНК этого установленно не было...).
Выдача для 16s rRNA Выдача для 23s rRNA