Базы данных
Для проведения дальнейшего анализа мною использовался следующий набор генов: ALB, CTSA, CTSB, CTSC, CTSD, CTSE, CTSF, CTSG, CTSH, CTSK, CTSL, CTSO, CTSS, CTSV, CTSW, CTSZ, HACD1, HACD2, LGMN, NAPSA, TPP1. По первому взгляду заметно, что есть 2 группы генов с похожими мнемониками (CTS_ и NACD_, где _ - любая буква/цифра). Логично предположить, что белки генов из одной групыы должны либо взаимодействовать друг с другом, либо быть связанны одной биологической функцией -(либо все вместе:)).
GO enrichment
Первым делом, я решил посмотреть в каких биологических процессах участвуют полученные мной белки. Для этого я использовал обогащение по биологической функции моего набора генов в базе данных GO. Согласно результатам обогащения, наиболее вероятно, что мой полученные мной белки участвуют в процессинге ангиотензина. Видимо они участвуют в протеолизе каких-то частков предшественника ангиотензина, так как в таблице есть разделы: протеолиз, процессинг белков (рис.1). Кроме того, протеазная активность изучаемого набора белков распространяется и на процессинг антигенов и презентацию их через главный комплекс гистосовместимости (рис.2).
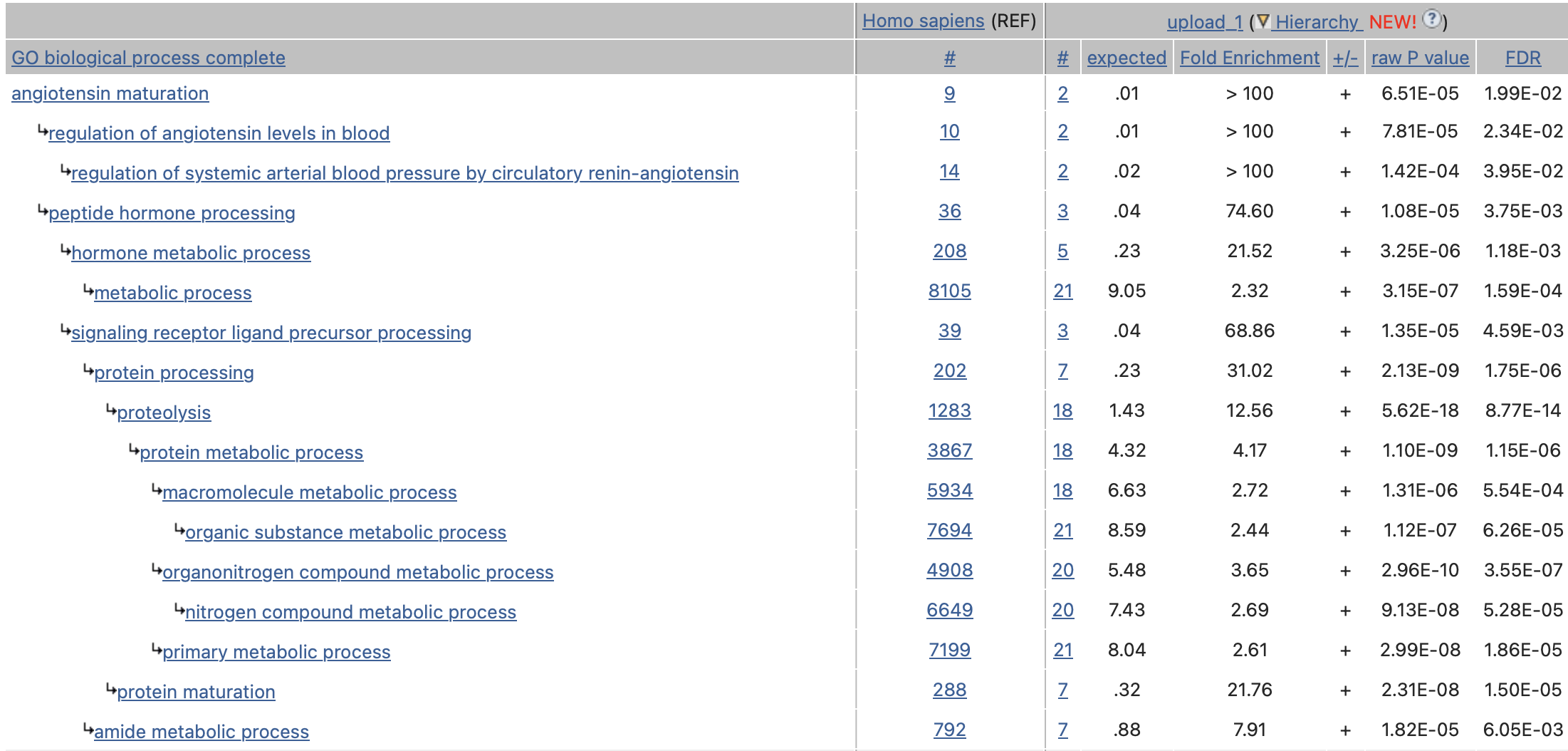
Рис.1.Функции анализируемых белков, согласно базе данных GO.
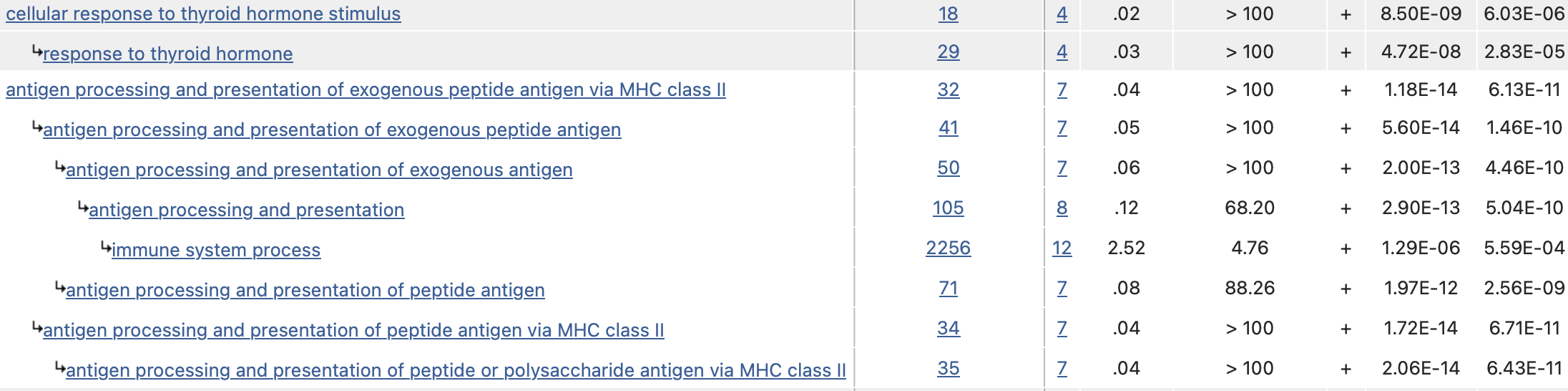
Рис.2. Другие функции исследуемых белков, согласно базе данных GO.
String analysis
Далее, я решил проверить мою гипотезу о взаимодействии моего набора белков, поэтому, весь набор был внесен для анализа в базу данных String. Перед выдачей графа, программа взяла другие мнемоники для двух белков (видимо, брала информацию о белках из отлИчной базы данных, чем той, которая использовалась при составлении задания): HACD1 = PTPLA, HACD2 = PTPLB. Результаты поиска по базе данных подтверждают мои предположения: все белки, мнемоника которых содержит 'CTS' взаимодействуют между собой, причем, этот факт определен эксперементально (рис.3). Почти все белки из этой группы взаимодействуют более чем с одним белком из этой же группы (кроме CTSO), кроме того, есть белки с другими мнемониками, которые взаимодействуют с белками этой группы: NAPSA, ALB (взаимодействия LGMN и TPP1 с белками этой группы не известны, однако есть данные об их ко-экспрессии, что может означать, что взаимодействия просто не исследованы) (рис.4). Все эти рассуждения наводят на мысль что белки группы 'CTS' могут собиратся в комплекс, причем некоторые белки присутствуют в нем почти всегда - что определяет протеазную активность комплекса, а другие - иногда (NAPSA, ALB), что придает комплексу специфичность к субстрату. Такое предположение могло бы дать объяснение факту, что эти белки участвуют в процессинге ангиотензина и антигенов.
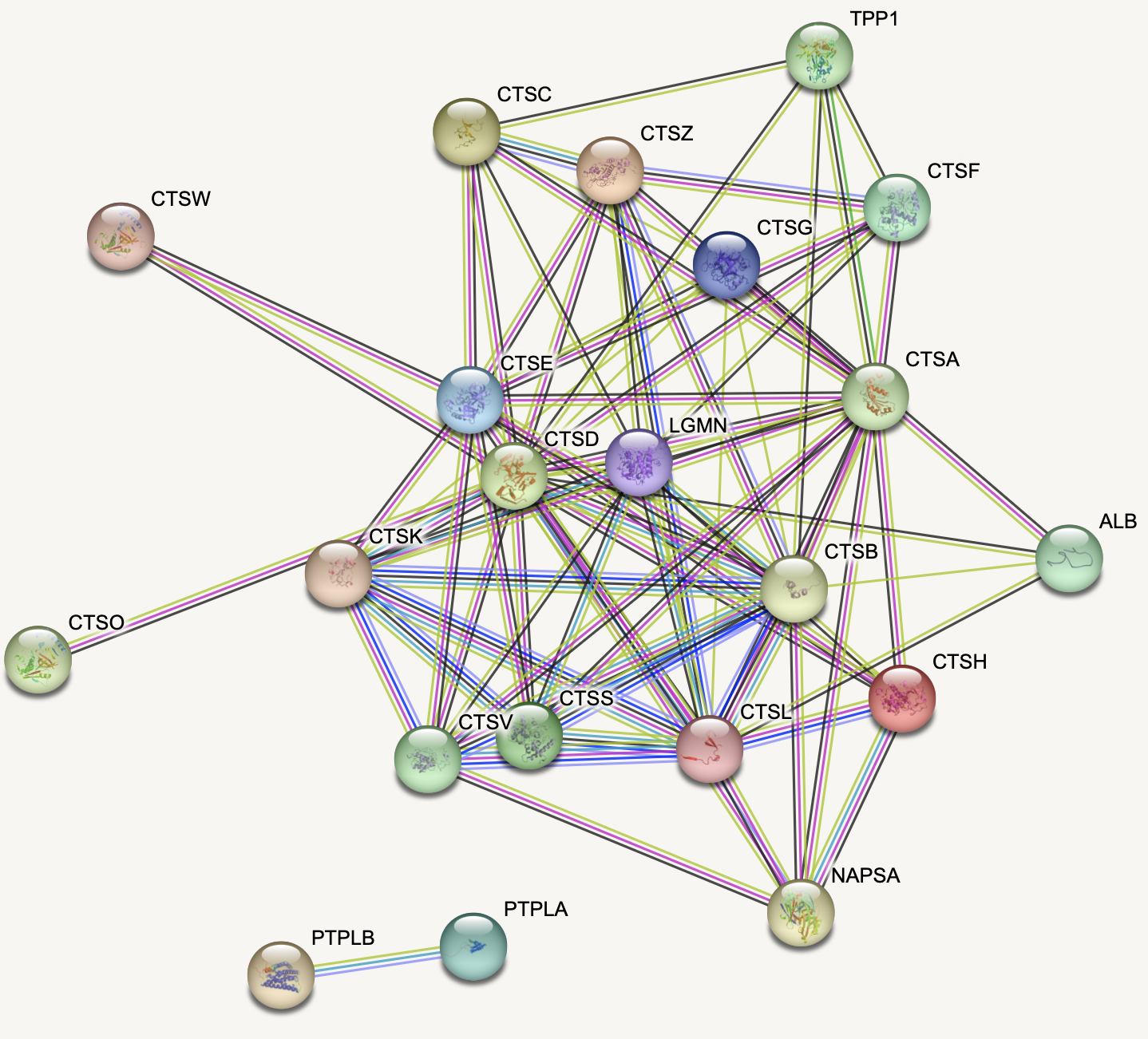
Рис.3. Граф String.
Похожесть же мнемоник другой групы белков - 'NACD', объясняется гомологией и ко-экспрессией этих белков, участвующих в элонгации длинноцепочечных жирных кислот. Помимо прочего, пространственная структура для всех белков известна, а также, база данных String более удобна для определения функций белков.
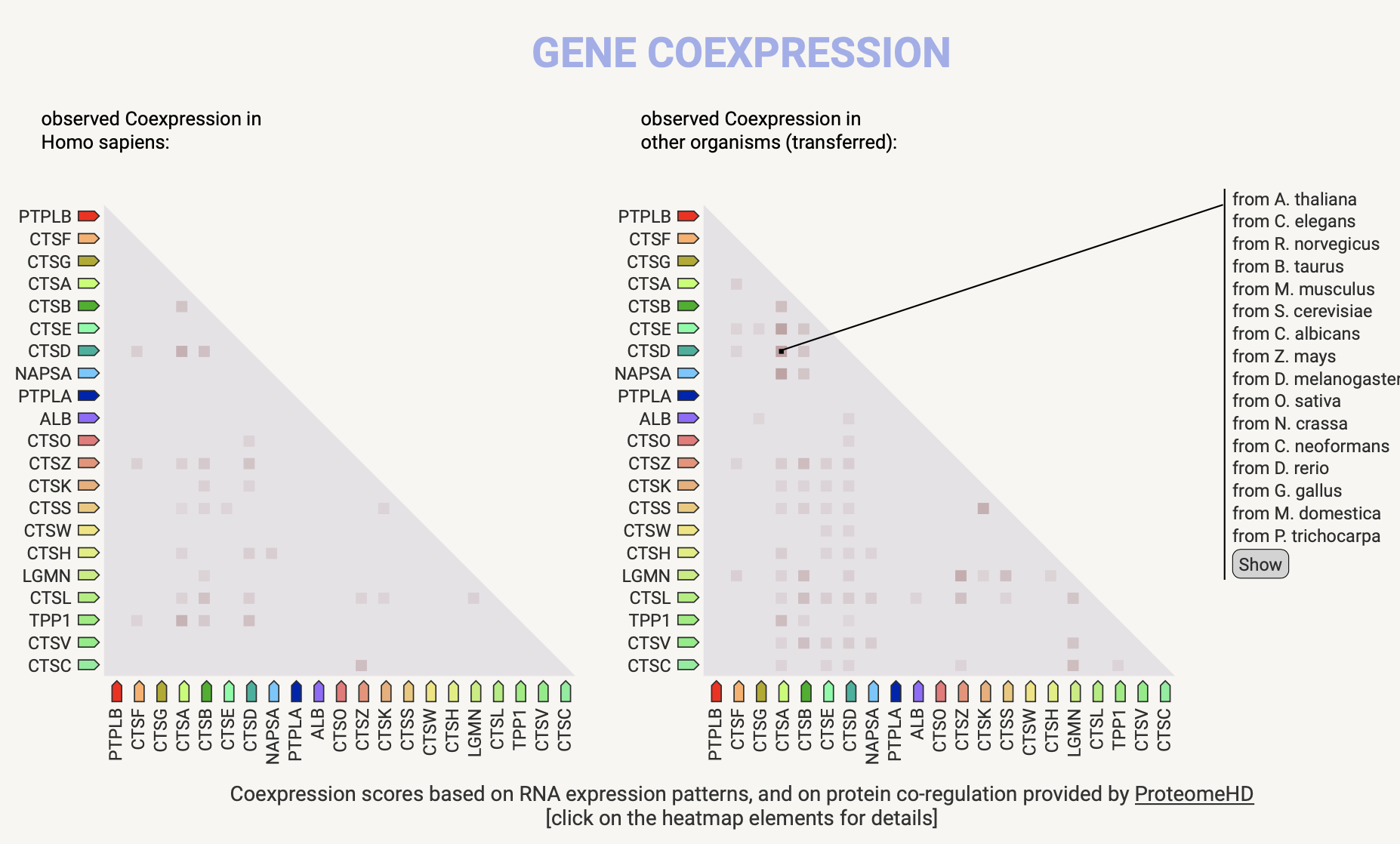
Рис.4. Схема ко-экспрессии анализируемых белков.
Reactome
Чтобы определить клеточный компартмент, в котором локализованы белки и представить бОльшее количество функций анализируемого набора белков я провел поиск по базе данных Reactome. Оказалось, что внутриклеточная локализация протеаз - лизосома (кто бы мог подумать...). Кроме того, теперь у меня есть красивая картинка (рис.5), которая показывает, что рассматриваемые белки участвуют в метаболизме белков нетолько внутриклеточных, но и внеклеточного матрикса (коллагена, например). По части иммунной системы эти белки не только участвуют в процессинге и презентации антигена, но и в процессах дегрануляции тромбоцитов и нейтрофилов.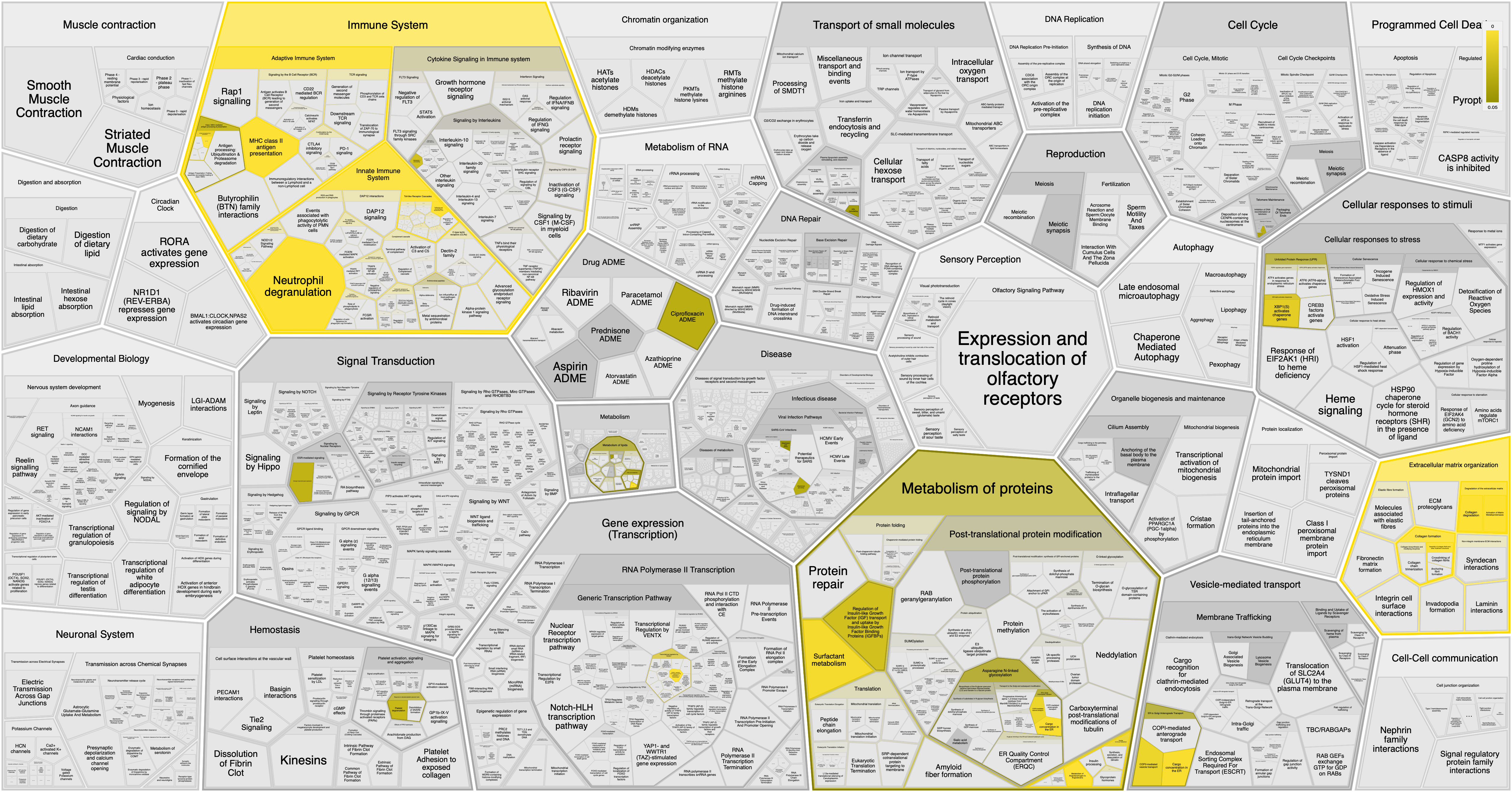
Рис.5. Функции исследуемой группы белков, согласно базе данных Reactome.