Реконструкция и визуализация паралогов и ортологов
Отобраные бактерии
| Бактерия | Мнемоника | Таксономия |
| Rubrobacter xylanophilus | RUBXD | Bacteria; Terrabacteria group; Actinomycetota; Rubrobacteria; Rubrobacterales; Rubrobacteraceae; Rubrobacter |
| Leifsonia xyli | LEIXX | Bacteria; Terrabacteria group; Actinomycetota; Actinomycetes; Micrococcales; Microbacteriaceae; Leifsonia |
| Thermobifida fusca | THEFY | Bacteria; Terrabacteria group; Actinomycetota; Actinomycetes; Streptosporangiales; Nocardiopsaceae; Thermobifida |
| Streptomyces avermitilis | STRAW | Bacteria; Terrabacteria group; Actinomycetota; Actinomycetes; Kitasatosporales; Streptomycetaceae; Streptomyces |
| Corynebacterium efficiens | COREF | Bacteria; Terrabacteria group; Actinomycetota; Actinomycetes; Mycobacteriales; Corynebacteriaceae; Corynebacterium |
| Mycobacterium leprae | MYCLE | Bacteria; Terrabacteria group; Actinomycetota; Actinomycetes; Mycobacteriales; Mycobacteriaceae; Mycobacterium |
| Rhodococcus jostii | RHOJR | Bacteria; Terrabacteria group; Actinomycetota; Actinomycetes; Mycobacteriales; Nocardiaceae; Rhodococcus |
Поиск гомологов
Поиск гомологов АТФ-связывающей субъединицы протеазы Clp E.coli производился с помощью blastp. Для этого я скачал последовательность АТФ-связывающей субъединицы протеазы Clp E.coli из записи UniProt. Далее, с помощью команды cat я объединил протеомы выбранных мной бактерий. Затем, мною был запущен поиск гомологов АТФ-связывающей субъединицы протеазы Clp E.coli программой blastp с порогом на E-value: 0,001. В результате было найдено 19 белков, большинство - АТФ-зависимые протеазы/металлпротеазы или шапероны.
Филогенетическое дерево
На основании выбранных бактерий и найденных гомологов было построенно филогенетическое дерево с помощью сервиса NGPhylogeny. Для построения множественного выравнивания был выбран алгоритм MAFT, дерево было построено алгоритмом FastME. Визуализация дерева проводилась в онлайн-сервисе iTOL(iTOL).
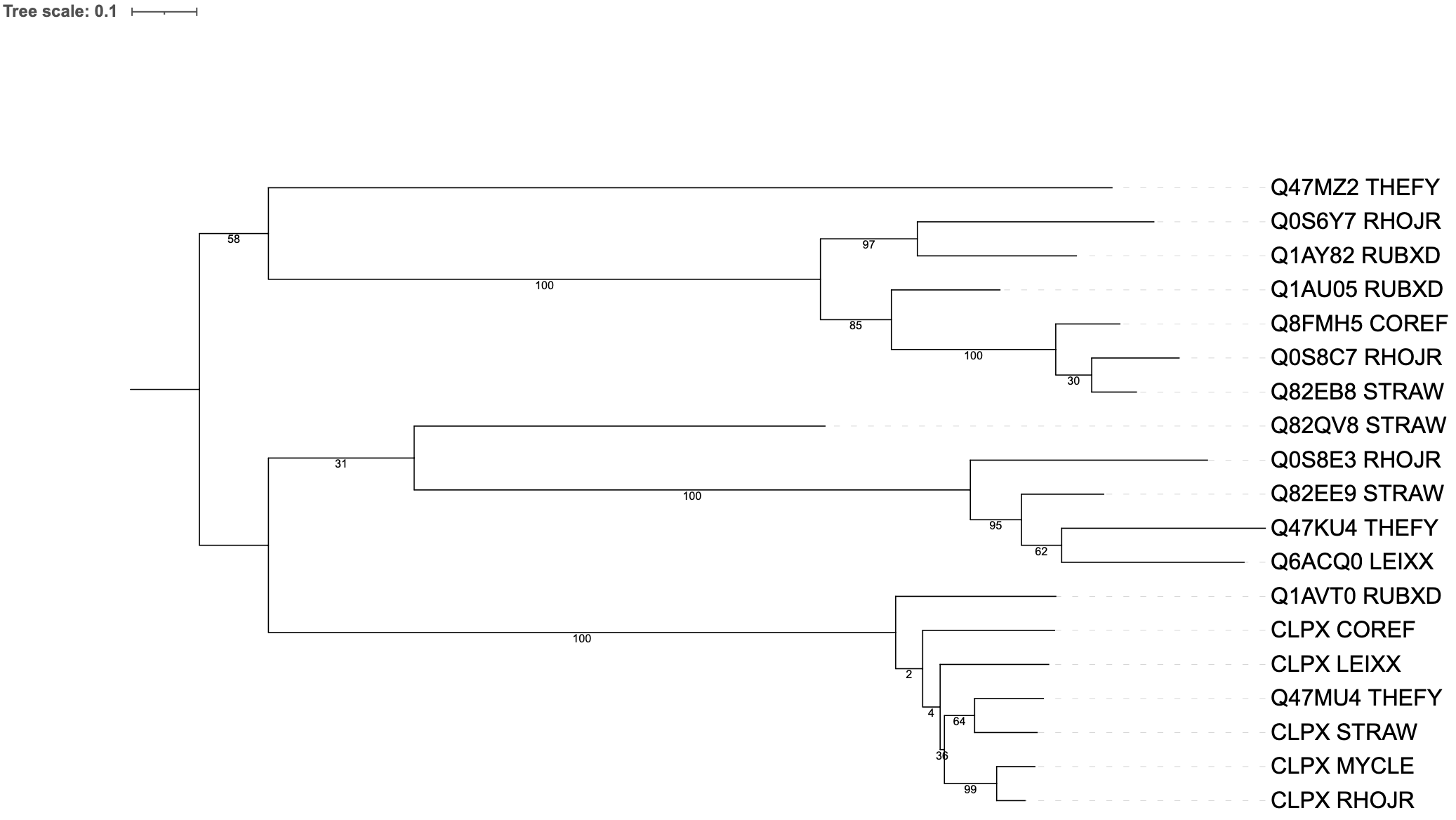
Рис.1.Филогенетическое дерево для выбранных организмов
Пары ортологов вполученном дереве: 1)CLPX_RHOJR - CLPX_MYCLE; 2)Q47MU4_THEFY - CLPX_STRAW; 3)Q82EB8_STRAW - Q0S8C7_RHOJR
Пары паралогов вполученном дереве: 1)Q1AU05_RUBXD - Q1AY82_RUBXD; 2)Q0S8C7_RHOJR - Q0S6Y7_RHOJR; 3)CLPX_COREF - Q8FMH5_COREF
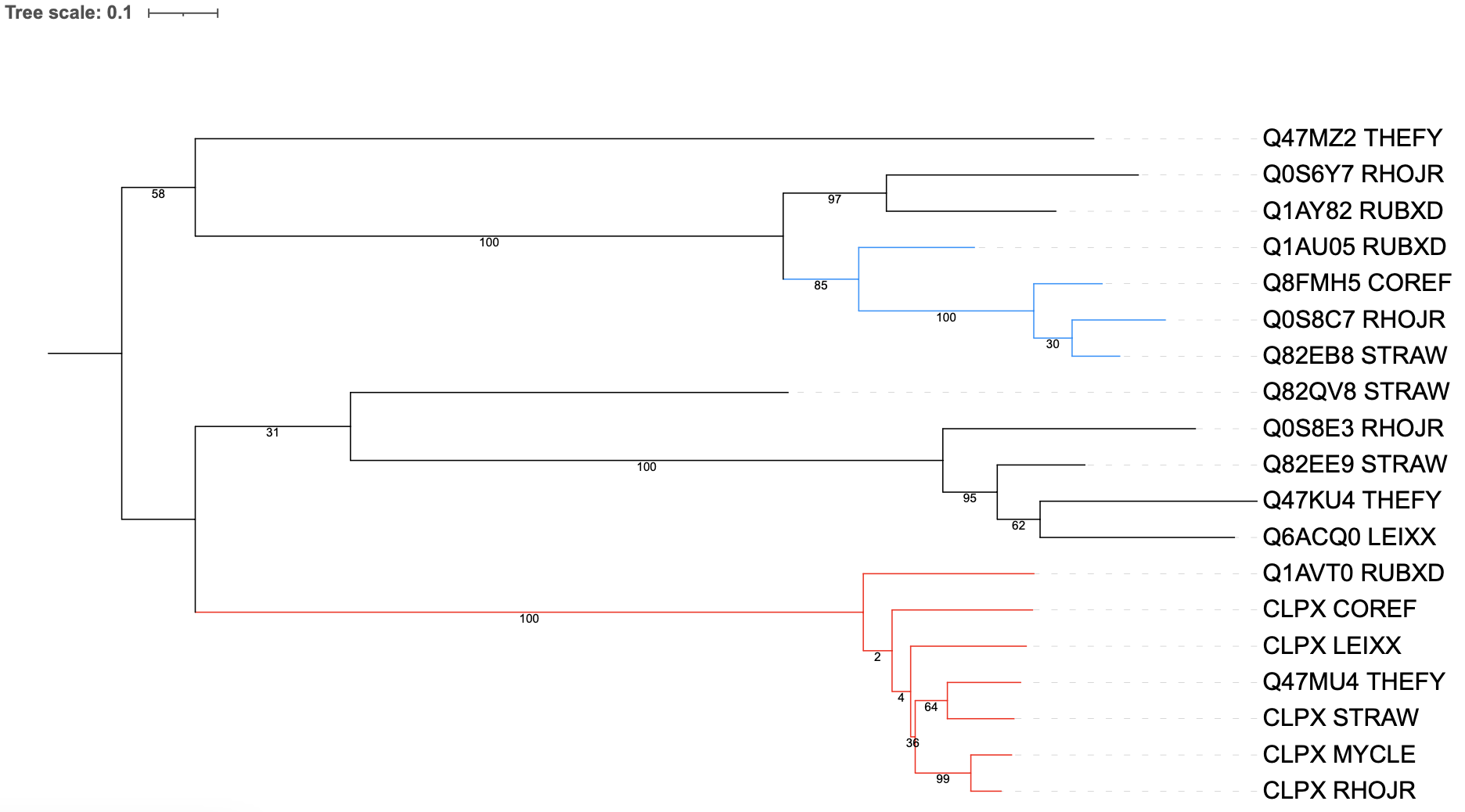
Рис.2.Филогенетическое дерево с выделенными группами ортологов. Красный - группа АТФ-связывающих доменов Clp-протеаз; синий - группа АТФ-зависимых цинковых металлопротеаз.
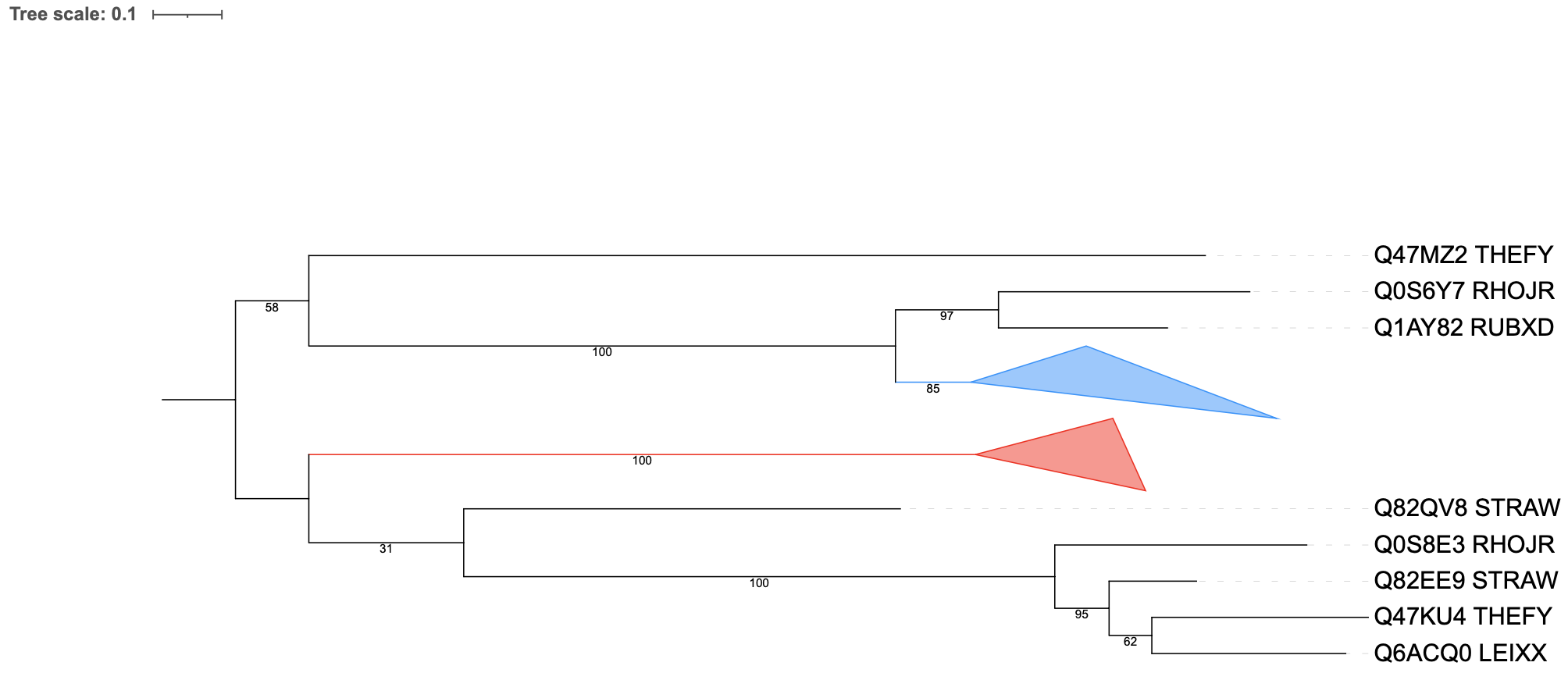
Рис.3.Филогенетическое дерево со схлопнутыми группами ортологов. Красный - группа АТФ-связывающих доменов Clp-протеаз; синий - группа АТФ-зависимых цинковых металлопротеаз FtsH.
Группа ортологов металлопротеаз слабо соответствуют филогении организмов, в то время как ортологичная группа Clp протеаз достаточно хорошо отражает дерево организмов. В группе Clp-протеаз отличаются только ветви LEIXX и COREF (в эталонном дереве поменяны местами). В синей же группе цинковых металлопротеаз выделяется ветвь {STRAW, RHOJR, COREF} против {RHOJR, RUBXD, THEFY}, которая совершенно отличается от эталонного дерева.