Сигналы и мотивы 2
Готовимся к MEME
В ходе практикума выполнялся поиск сигнала посадки σ-субъединицы РНК-полимеразы в промоторных областях генов бактерии Thermus aquatiqus. Известно, что это термофильный вид бактерий, что наталкивает на мысль о том, что у нее в геноме должен быть высокий процент GC-пар. Интересно посмотреть будут ли наблюдаться достаточно консервативный сигнал для ее генома. Для анализа была взята сборка генома Thermus aquatiqus Y51MC23. Использовав сервис Operon mapper получил координаты оперонов.
Далее, адаптировав скрипт, написанный Сергеем Бушуевым, получил последовательности обучения, тестирования и негативного контроля.
Делаем MEME
Поиск сигналов производился с помощью локального MEME командой:
meme train.fasta -dna -mod zoops -nmotifs 3 -minw 6 -maxw 50 -maxsites 50
Программа определила три мотива в обучающих последовательностях со значениями E-value: 2.2e-005; 7.4e-005 и 4.7e-001 соответственно. Дальше я буду работать со вторым мотивом, который, несмотря на меньшее E-value, привлекает меня большей длинной и большим числом консервативных А-Т позиций.
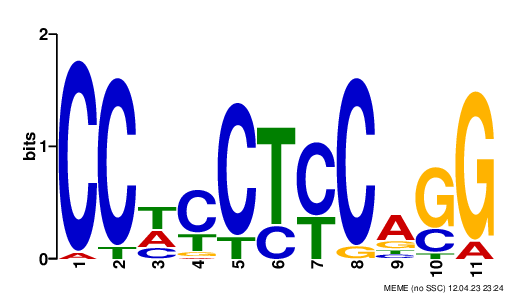
Рис.1.LOGO первого мотива
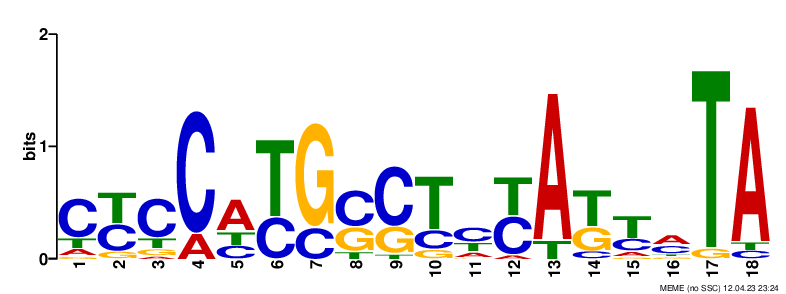
Рис.2.LOGO второго мотива
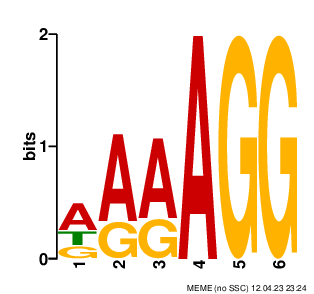
Рис.3.LOGO третьего мотива
Делаем FIMO
fimo --norc -motif CYCCAYGSCTYYAKYMTA -thresh 0.001 meme.txt test.fa
fimo --norc -motif CYCCAYGSCTYYAKYMTA -thresh 0.001 meme.txt neg_contr.fa
По результатам работы FIMO второй мотив был найден в 38 промоторах для тестового набора и в 11 последовательностях в отрицательном контроле. Различия между тестом и отрицательным контролем не слишком различаются. Могу предположить, что тут свой вклад вносит большой процент GC пар в геноме бактерии который связан не столько с функциональной ролью мотива, сколько необходим для предотвращения плавления цепей ДНК при высоких температурах, которые характерны для мест обитаний Thermus aquaticus.
Возможно именно такие участки повторяющихся GC распознаются MEME как консенсус, что не оправдывает себя в дальнейшем при сравнении тестового набора и отрицательного контроля потому как в других частях генома так же будут попадатся богатые GC участки. Это подтверждается тем, что данном отчете мной приведена лишь одна выдача FIMO, где количество находок для тестового набора превосходит отрицательный контроль. На самом же деле мной было проведено несколько итераций составления наборов последовательностей, работы MEME и работы FIMO, и во всех случаях был получен противоположный результат. Предполагаю что для более коректной работы вышеуказанных программ нужно проводить предварительную фильтрацию таких "участков малой сложности".