Сигналы и мотивы 3
Поиск консервативного домена
Для анализа был выбран домен семейства гистонов. Seed этого семейства представлен 27 последовательностями. В качестве консервативного домена был выбран участок [PY][FIV].[RK].[LVI][RK][ED][VIL] (рис.1). Этот мотив был найден в 14 из 27 последовательностях. Полученный мотив был проанализирован с помощью сервиса MyHint. В результате было найдено 309 находок. Интересно, что среди полученных белков с похожим мотивом есть белки, связывающиеся с нуклеиновыми кислотами сиквенснеспецифическим образом (рибонуклеаза Y, eIF2, YpdB - белок, регулирующий транскрипцию, элонгационные факторы G и Tu), что указывает на возможную функцианальную роль найденного мотива.
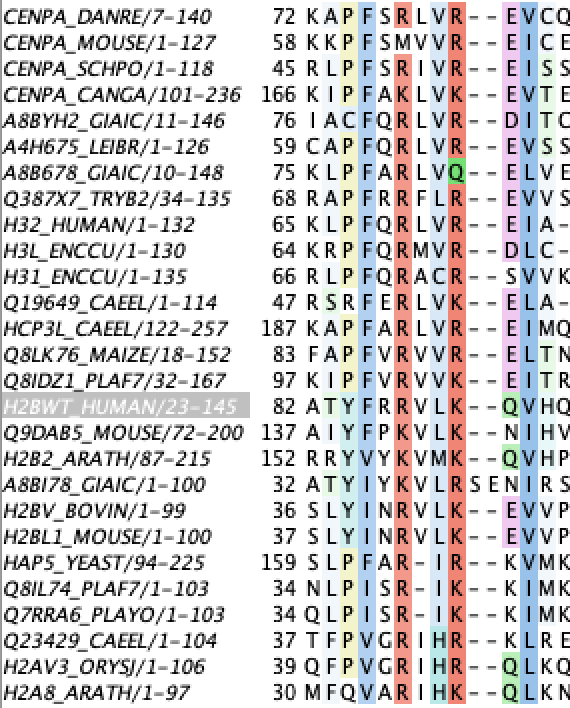
Рис.1.Выравнивание последовательностей гистонов
Найденная последовательность характерна для одной из клад
На основании полученных последовательностей было постороенно филогенетическое дерево(UPGMA, NJ). Полученный мотив есть в темно-зеленой кладе из 12 последовательностей (рис.2). Все последовательности этой группы являются гистонами Н2А и Н2В. Данный мотв есть и в других выделенных группах, однако их объем слагают не более трех последовательностей (в основном одна), что вызывает недоверие при обсуждении характерности мотива.
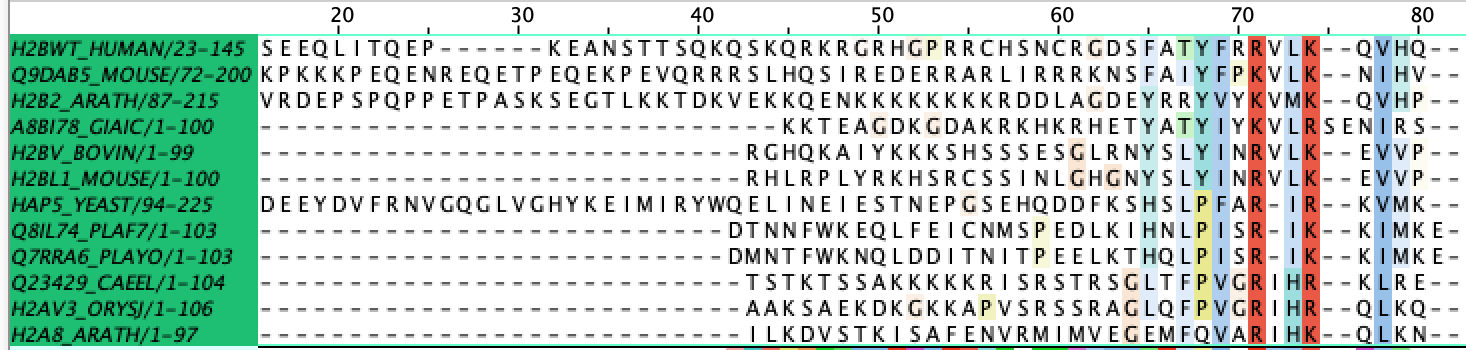
Рис.2. Филогенетическое дерево выравнивания последовательностей
PSI-BLAST
DAP5 (P78344) - гомолог эукариотического фактора инициации трансляции 4G. Известно, что он входит в состав сканирующих комплексов рибосом в процессе инициации трансляции, однако механизм его попадания туда, как и причина, остаются неизвестными.
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
| 1 | 6 | Q64350.2 | 0.028 | Q04637.4 | 2e-51 |
| 2 | 24 | P59325.1 | 0.049 | Q6K641.1 | 8e-180 |
| 3 | 0 | - | - | P41110.1 | 1e-179 |
PSI-BLAST стабилизировался на 3 итерации: не было найдено белков с E-value выше порога. Среди находок с самым высоким E-value находятся фактор инициации трансляции 4G, и его гомолог 4G-3, белок, подобный фактору инициации трансляции 5, субъединица фактора инициации трансляции 4F и другие белки, содержащие MIF4G домен.
Подсчет ТА в геноме
Для проверки гипотезы был выбран геном штамма Bacillus subtilis subsp. subtilis str. 168.Для рассчетов был взят скрипт Сафоновой Варвары. По результатам работы скрипта ожидаемое количество ТА сайтов оказалось равно 257607, а наблюдаемое - 218025. Оценка статистической значимости в скрипте проводится с помощью теста хи-квадрат. Значение p-value получилось 0.0, что говорит о наличии значимых различий между ожидаемым и наблюдаемым значением.