Поиск полиморфизмов у человека
1. Подготовка чтений
Контроль качества чтений был проведен с помощью программы FastQC, установленной на kodomo. Вызов программы выглядит следующим образом:
$ fastqc chr17.fastq
В результате ее работы был получен .html файл с отчетом. На рисунке 1 в виде боксплота представлено изменение качества чтений на каждый нуклеотид.
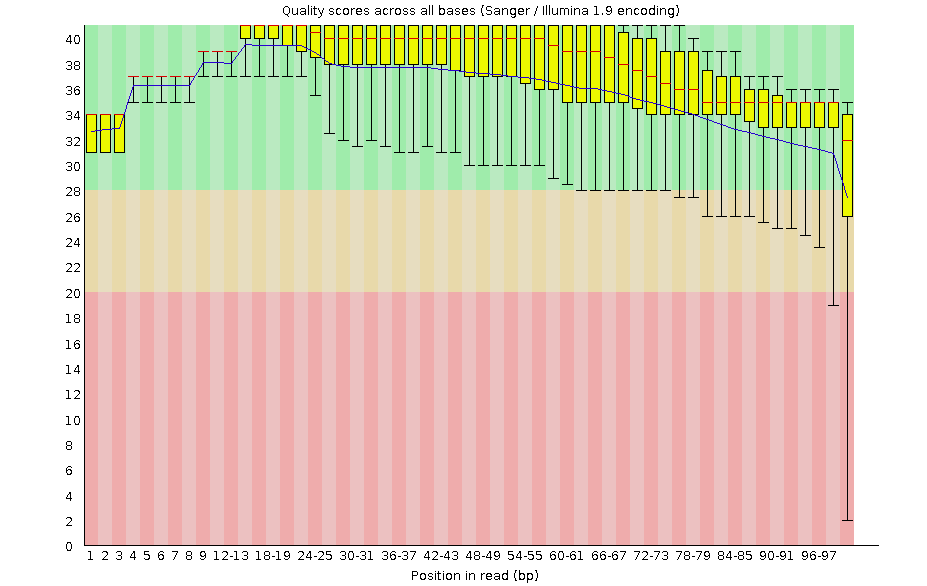
Рисунок 1. Изменение качества чтений до чистки
Далее было необходимо очистить чтения от участков плохого качества. Это можно сделать с помощью программы Trimmomatic:
$ java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 chr17.fastq chr17_trim.fastq TRAILING:20 MINLEN:50
Trimmomatic оставила чтения длиной не меньше 50 (MINLEN:50) и с конца каждого такого чтения удалила нуклеотиды с качеством ниже 20 (TRAILING:20). После чистки число чтений сократилось с 11046 до 10868 (98,39% от общего числа). Очищенные чтения подали на вход программе FastQC:
$ fastqc chr17_trim.fastq
На рисунке 2 представлена диаграмма из отчета, аналогичная той, что была получена для неочищенных чтений:

Рисунок 2. Изменение качества чтений после чистки
2. Картирование чтений
| $ hisat2-build chr17.fasta chr17 | Индексирует референсную последовательность |
| $ hisat2 -x chr17 -U chr17_trim.fastq -S chr17_align.sam --no-spliced-alignment --no-softclip | Строит выравнивание прочтений и референса в формате .sam |
| $ samtools view chr17_align.sam -bo chr17_align.bam | Переводит выравнивание чтений с референсом в бинарный формат .bam |
| $ samtools sort chr17_align.bam sorted | Сортирует выравнивание чтений с референсом по координате в референсе начала чтения |
| $ samtools index sorted.bam | Индексирует отсортированный .bam файл |
Число чтений, картированных на референсную хромосому: 10834
Число не картированных чтений: 34
3. Анализ SNP
| $ samtools mpileup -uf chr17.fasta -o snp.bcf sorted.bam | Создает бинарный файл с полиморфизмами |
| $ bcftools call -cv snp.bcf -o snp.vcf | Создает файл со списком отличий между референсом и чтениями в формате .vcf |
Описание полиморфизмов
Полученный .vcf файл содержит информацию об отличиях исследуемой хромосомы от референса. Всего было обнаружено 59 полиморфизмов, среди которых 4 являются инделями и 55 - SNP.
В таблице ниже приведены примеры встретившихся мутаций:
| # | Координата | Тип полиморфизма | Буква в референсе | Буква в чтениях | Глубина покрытия | Качество чтений |
| 1 | 44832598 | Замена | C | T | 32 | 225.009 |
| 2 | 79562977 | Делеция | AGTTGTT | AGTT | 11 | 188.468 |
| 3 | 44833088 | Делеция | ATTTTTTTTTTTTTTTTTTT | ATTTTTTTTTTTTTTTT, ATTTTTTTTTTTTTTTTT * | 91 | 5.79864 |
* Такая запись означает, что в чтениях было найдено три различных аллеля (один из них референсный). Первый аллель из чтений короче референсного на три нуклеотида, второй - на два
4. Аннотация SNP
Полученные snp были проаннотированы по 5 базам данных: refgene, dbsnp, 1000 genomes, GWAS, Clinvar. В таблице ниже приведены использованные команды.
| $ convert2annovar.pl -format vcf4 snp.vcf > snp.avinput | Файл со списком отличий между ридами и референсной последовательностью переводим в формат, с которым умеет работать annovar |
| $ annotate_variation.pl -out refgen -build hg19 snp.avinput /nfs/srv/databases/annovar/humandb.old/ | Аннотаация по refgen. Выделяет положение полиморфизма и вносимые изменения, если он находится в экзоне. |
| $ annotate_variation.pl -filter -dbtype 1000g2014oct_all -buildver hg19 -out 1000Genomes snp.avinput /nfs/srv/databases/annovar/humandb.old/ | Аннотация по 1000 genomes показывает частоты аллелей. Максимальная частота: 0.979034 для пары G/A (44827803). Минимальная: 0.000199681 для T/G (44833806). Для каждого из них также указано, относится аллель к homo или к het |
| $ annotate_variation.pl -filter -out snp_filtered -build hg19 -dbtype snp138 snp.avinput /nfs/srv/databases/annovar/humandb.old/ | Аннотация разделяет полиморфизмы по наличию в dbsnp. Согласно выдаче скрипта, 49 SNP имеют rs (аннотированны) |
| $ annotate_variation.pl -regionanno -build hg19 -out GWAS -dbtype gwasCatalog snp.avinput /nfs/srv/databases/annovar/humandb.old/ | Аннтоация по GWAS показывает ассоциированные с SNP признаки |
| $ annotate_variation.pl -filter -out clinvar -build hg19 -dbtype clinvar_20150629 snp.avinput /nfs/srv/databases/annovar/humandb.old/ | Аннотация по ClinVar предоставляет информацию о связи SNP с клиническими болезнями. В нашем случае, ClinVar не содержит информации по полиморфизмам |
RefSeq в annovar делит SNP по их локализации: intergenic(1), intronic(49), UTR3(5), exonic(3). Отсюда же можно узнать, в какие гены попали наши SNP - в CD79B (Ig-beta protein of the B-cell antigen component) и NPLOC4 (Ubiquitin recognition factor). Также присутствует 1 участок между генами NSFP1 и ARL17A.
GWAS содержит информацию о трех SNP, два из которых приходятся на наши гены и один - на интрон: CD79B - болезнь Паркинсона и рак яичников у носителей мутации в гене BRCA1, CD79B - цвет глаз и интронный SNP - рост.
В этом практикуме для работы мне досталась семнадцтая хромосома из сборки человеческого генома версии hg19 (последняя - hg38)