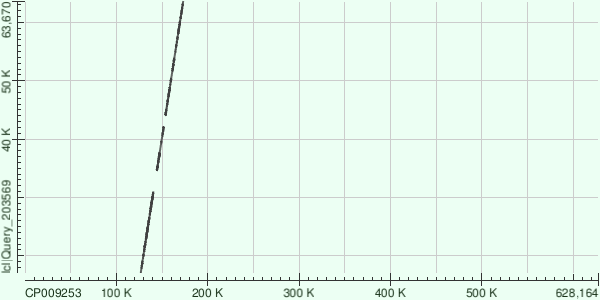
ID: 3
Lenght: 68162
Score: 5421 bits
E-value: 0
Identities: 9745/13012(75%)
Gaps: 552/13012(4%)
Coords: 18288-31028
Query cover: 6%
С помощью velveth генерируем k-меры длины 29:
$ velveth kmer 29 -fastq trimm.fastq -short
Здесь kmer - название директории, в которую после выполнения программы запишутся три файла: Log, Roadmaps и Sequences.
$ velvetg kmer
Final graph has 9594 nodes and n50 of 264, max 68162, total 1691794, using 0/11812517 reads
N50: 264
Максимальная длина: 68162
Три самых длинных контига [ID/длина/покрытие]: 3/68162/49.266600, 9/50157/54.850868, 6/47295/46.274532.
Медиана покрытий составляет около 4.5, среди всех покрытий особенно сильно выделяется покрытие контига 9481/1/64555, превышающее медиану в 14345 раз. Наименьшее покрытие при той же длине контига составило 1, например у 8250/1/1.
ID: 3
Lenght: 68162
Score: 5421 bits
E-value: 0
Identities: 9745/13012(75%)
Gaps: 552/13012(4%)
Coords: 18288-31028
Query cover: 6%

ID: 9
Lenght: 50157
Score: 5760 bits
E-value: 0
Identities: 7225/9217(78%)
Gaps: 244/9217(2%)
Coords: 16226-25315
Query cover: 5%
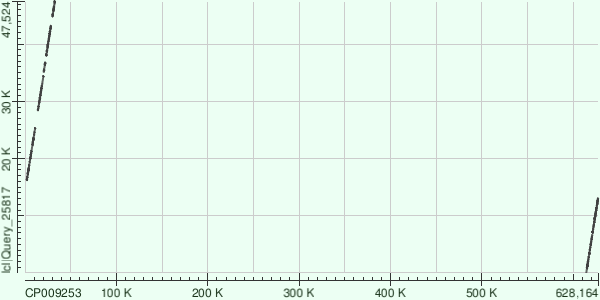
Можно видеть, что контиг разбит на две части. Первая соотвествует выравниванию с началом референсной хромосомы, вторая - с концом.
ID: 6
Lenght: 47295
Score: 4050 bits
E-value: 0
Identities: 5690/7387(77%)
Gaps: 206/7387(2%)
Coords: 20452-27763
Query cover: 4%
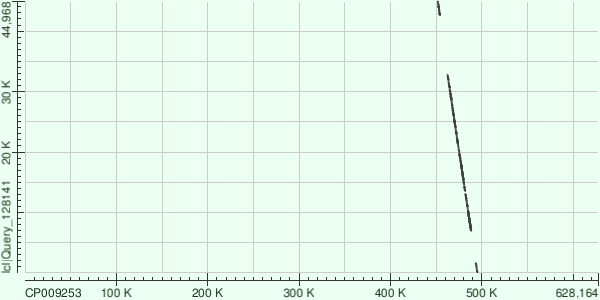
Здесь контиг инвертирован относительно референса, так как лег на обратную цепь.
Для каждого из трех контигов нашлось больше одного выравнивания с референсной хромосомой, что видно на представленных графиках - соответствия между двумя последовательностями отображены прерывистыми линиями.
Для удобства все последовательности адаптеров были объединены в один файл командой:
$ cat *.fa >> adapters.fa
Перед очисткой концов чтений удаляем возможные остатки праймеров с помощью Trimmomatic:
$ java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 SRR4240381.fastq no_adapters.fastq ILLUMINACLIP:adapters.fasta:2:7:7
Input Reads: 13710994 Surviving: 13710994 (100,00%) Dropped: 0 (0,00%)
Затем удаляем плохие буквы с концов чтений, оставив толко чтения длины не менее 30:
$ java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 no_adapters.fastq trimm.fastq LEADING:20 TRAILING:20 MINLEN:30
Input Reads: 13710994 Surviving: 11812517 (86,15%) Dropped: 1898477 (13,85%)
Исходный файл весил 1.4ГБ, полученный после очистки - 1.2ГБ (на 0.2ГБ меньше).