1. Структуры ДНК

Рисунок 1. Структуры A-, B- и Z-форм ДНК, визуализированные в программе PyMol

Рисунок 1. Структуры A-, B- и Z-форм ДНК, визуализированные в программе PyMol
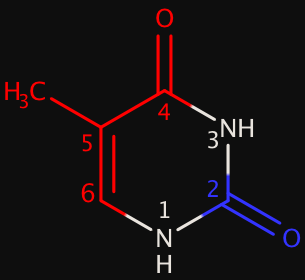
Из записи 1bna B-формы ДНК был выбран тимин-19, для которого в дальнейшем было определено расположение атомов относительно большой и малой бороздок. На рисунке справа представлена визуализация тимина в MarvinSketch. Красным цветом помечены атомы, явно смотрящие в сторону большой бороздки, синим - в сторону малой, белым выделены атомы, обращенные к сахаро-фосфатному остову.
Так, в сторону большой бороздки обращены атомы С2 и O2
В сторону малой - C4, O4, C5, C6, C7
В таблице 1 приведены некоторые характеристики трех различных форм ДНК.
| A-форма | B-форма | Z-форма | |
| Тип спирали (правая/левая) | Правая | Правая | Левая |
| Шаг спирали (Å) | 28 | 33.7 | 43.5 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки | 7.9 (B/DG'29/P - A/DA'6/P) | 17.2 (A/DG'9/P - B/DG'29/P) | 18.3 (A/DC'8/P - B/DC'30/P) |
| Ширина малой бороздки | 16.8 (A/DA'6/P - B/DG'37/P) | 11.7 (A/DC'8/P - B/DG'29/P) | 8.2 (A/DG'6/P - B/8MG'10/P) |
Таблица 1. Результаты сравнения A-, B- и Z-форм ДНК
С помощью программ find_pair и analyze пакета 3DNA были получены все значения, необходимые для анализа и сравнения торсионных углов, структуры водородных связей и стекинг-взаимодействий в молекулах нуклеиновых кислот.
Для перевода pdb-файлов с рассматриваемыми структурами в старый, понятный 3DNA формат, была использована установленная на kodomo программа remediator.
Ниже представлена часть содержимого файла XXXX.out со значениями торсионных углов в заданной структуре тРНК.
Strand I base alpha beta gamma delta epsilon zeta chi 1 G --- --- 163.4 88.6 -140.9 -65.6 178.3 2 G -70.4 177.6 54.4 77.2 -156.2 -75.1 -168.7 3 G -37.3 164.5 45.3 80.1 -159.0 -60.1 -160.4 4 G 153.6 -176.7 -179.2 82.4 -144.1 -73.9 -171.0 5 U -61.1 172.2 52.3 81.6 176.5 -79.2 -157.4 6 A 179.3 -134.9 170.0 104.3 -81.4 -49.9 -167.2 7 C 28.8 -148.1 46.8 87.0 -154.4 -66.4 -166.7 8 G -58.4 179.5 50.5 82.8 -155.1 -76.1 -159.9 9 A -57.0 173.0 38.9 80.3 -159.8 -85.3 -152.4 10 G 146.7 -156.2 178.3 82.1 -132.3 -76.8 -178.0 11 G -35.8 159.8 46.6 80.9 -159.3 -64.4 -172.0 12 U -85.5 -157.9 50.5 86.4 -160.0 -59.6 -160.7 13 U -77.6 -177.2 54.6 88.1 -122.8 -101.3 -162.5 14 A -117.4 -166.8 59.6 80.4 -144.4 -62.1 -175.6 15 U -59.7 164.3 58.1 83.8 -137.9 -63.5 -167.6 16 U -62.0 175.8 49.7 85.3 -158.1 -59.0 -156.6 17 C -61.8 179.1 45.0 85.4 -124.9 -134.8 -156.3 18 C 106.4 -110.9 -178.8 83.9 -105.5 -94.8 180.0 19 G -49.4 145.5 52.4 76.0 -151.2 -69.1 -170.1 20 G -60.9 -179.4 49.5 77.4 -148.6 -71.7 -159.9 21 C -53.1 170.9 47.6 79.4 -104.1 -128.2 -141.4 22 G -148.7 158.2 41.7 87.6 -150.6 -67.6 -176.9 23 C -75.6 -167.1 52.2 83.9 -162.1 -74.2 -159.8 24 C 136.3 -165.8 -171.6 83.3 -126.8 -63.1 -172.3 25 A -45.3 165.1 50.8 79.0 -177.7 -73.6 -156.7 26 A 174.8 170.5 172.2 90.5 -119.7 -62.8 -170.0 27 G -47.6 152.6 55.4 83.3 173.8 24.9 -144.4 28 G -105.5 124.5 77.6 89.5 -170.2 -156.1 -133.5
Strand II base alpha beta gamma delta epsilon zeta chi 1 C -61.2 178.2 50.2 84.3 -137.0 -88.2 -148.2 2 C -66.4 176.0 52.5 80.6 -150.8 -76.3 -160.3 3 C -67.7 179.0 55.6 82.2 -148.1 -72.9 -172.4 4 C -63.5 166.5 54.5 80.8 -161.1 -71.0 -152.9 5 A -53.4 171.5 46.8 83.0 -149.4 -78.3 -154.4 6 U -56.8 171.9 44.3 80.7 -153.5 -75.6 -161.9 7 G 144.8 -145.8 -178.3 84.9 -134.7 -78.7 177.9 8 C -47.8 154.9 46.4 83.8 -152.3 -87.4 -152.8 9 U -73.3 -165.3 46.2 80.2 -152.4 -87.6 -152.5 10 C -67.2 -178.7 53.4 85.1 -165.8 -56.5 -170.5 11 C -90.2 -161.0 50.9 85.4 -152.9 -76.6 -169.0 12 A -73.2 -118.1 67.2 103.4 -176.6 -101.5 -91.5 13 G 40.5 -173.5 37.3 92.6 -27.5 147.2 -118.0 14 U -59.0 162.2 50.8 83.9 -109.1 -119.7 -154.0 15 U 132.7 -136.4 -177.3 84.7 -108.5 -76.7 179.0 16 A -56.9 -166.3 45.1 83.6 -154.6 -94.0 -153.3 17 G -63.0 163.8 49.3 78.2 -166.2 -67.0 -170.8 18 G 151.0 176.2 -179.5 87.7 -126.6 -73.9 -175.4 19 C -68.4 -165.7 45.4 85.8 -176.8 -61.9 -139.6 20 C -57.1 174.4 49.4 79.2 -152.1 -69.4 -154.3 21 A -72.1 -178.9 53.7 80.6 -155.0 -59.4 -159.2 22 C -58.7 170.1 50.7 77.1 -147.5 -53.9 -166.5 23 G -62.2 173.9 56.0 80.6 -149.9 -69.2 -167.2 24 G -54.5 166.2 48.5 80.9 -154.3 -75.9 -170.9 25 A -36.2 -175.4 57.8 85.6 169.2 20.2 -113.3 26 U -66.7 -138.4 57.6 82.0 -152.1 -128.2 -161.4 27 C -5.9 130.7 49.6 97.5 177.0 152.8 -153.0 28 C 151.5 -152.4 49.3 84.6 -139.7 -75.4 -165.0
Сравнение значений торсионных углов, полученных для тРНК и трех форм ДНК, указывает на сходство тяжей тРНК с A-формой ДНК. Также в пользу сходства с А-ДНК свидетельствет часть выдачи программы, в которой сравниваются пары динуклеотидов тРНК с таковыми в правозакрученных формах других нуклеиновых кислот (A-like; B-like; TA-like; intermediate of A and B, or other cases). Таким образом, все рассмотренные динуклеотиды оказались схожи с динуклеотидами именно A-формы ДНК.
Помимо значений торсионных углов, в файле XXXX.out можно найти информацию о всех водородных связях между основаниями в заданной структуре тРНК, ниже приведен соответствующий фрагмент записи.
Strand I Strand II Helix
1 (0.014) ....>B:.902_:[..G]G-----C[..C]:.971_:B<.... (0.007) |
2 (0.007) ....>B:.903_:[..G]G-----C[..C]:.970_:B<.... (0.007) |
3 (0.005) ....>B:.904_:[..G]G-----C[..C]:.969_:B<.... (0.005) |
4 (0.009) ....>B:.905_:[..G]G-----C[..C]:.968_:B<.... (0.007) |
5 (0.012) ....>B:.906_:[..U]U-----A[..A]:.967_:B<.... (0.010) |
6 (0.012) ....>B:.907_:[..A]A-----U[..U]:.966_:B<.... (0.009) |
7 (0.005) ....>B:.949_:[..C]C-----G[..G]:.965_:B<.... (0.009) |
8 (0.004) ....>B:.950_:[..G]G-----C[..C]:.964_:B<.... (0.003) |
9 (0.003) ....>B:.951_:[..A]A-----U[..U]:.963_:B<.... (0.002) |
10 (0.007) ....>B:.952_:[..G]G-----C[..C]:.962_:B<.... (0.005) |
11 (0.005) ....>B:.953_:[..G]G-----C[..C]:.961_:B<.... (0.006) |
12 (0.002) ....>B:.954_:[..U]U-**--A[..A]:.958_:B<.... (0.010) |
13 (0.003) ....>B:.955_:[..U]U-**+-G[..G]:.918_:B<.... (0.005) x
14 (0.009) ....>B:.937_:[..A]A-**--U[..U]:.933_:B<.... (0.006) |
15 (0.004) ....>B:.938_:[..U]U-**--U[..U]:.932_:B<.... (0.005) |
16 (0.004) ....>B:.939_:[..U]U-----A[..A]:.931_:B<.... (0.003) |
17 (0.003) ....>B:.940_:[..C]C-*---G[..G]:.930_:B<.... (0.009) |
18 (0.005) ....>B:.941_:[..C]C-----G[..G]:.929_:B<.... (0.010) |
19 (0.006) ....>B:.942_:[..G]G-----C[..C]:.928_:B<.... (0.005) |
20 (0.009) ....>B:.943_:[..G]G-----C[..C]:.927_:B<.... (0.008) |
21 (0.005) ....>B:.944_:[..C]C-**--A[..A]:.926_:B<.... (0.009) |
22 (0.011) ....>B:.910_:[..G]G-----C[..C]:.925_:B<.... (0.005) |
23 (0.002) ....>B:.911_:[..C]C-----G[..G]:.924_:B<.... (0.009) |
24 (0.002) ....>B:.912_:[..C]C-----G[..G]:.923_:B<.... (0.011) |
25 (0.004) ....>B:.913_:[..A]A-**+-A[..A]:.945_:B<.... (0.005) |
26 (0.005) ....>B:.914_:[..A]A-**--U[..U]:.908_:B<.... (0.009) |
27 (0.007) ....>B:.915_:[..G]G-**+-C[..C]:.948_:B<.... (0.019) x
28 (0.026) ....>B:.919_:[..G]G-----C[..C]:.956_:B<.... (0.003) +
"+" обозначены изолированные пары, участвующие в стабилизации третичной структуры тРНК.
"Х" означает смену цепи.
В состав стеблей (stems) могут входить только канонические (WC) или неоднозначные пар оснований (Wobble base pairs). Учитывая это, можем сделать вывод, что исследуемая тРНК состоит из четырех стеблей, составленных из пар 1-6, 7-11, 18-20 и 22-24, соответственно.
Также в структуре содержится 9(8) неканонических пар оснований. Все они, вместе с канонической двадцать восьмой G-C парой, вносят вклад в стабилизацию структуры тРНК.
Из файла ХХХХ.out была выбрана пара с наибольшей площадью перекрывания - двадцатая.
Затем, с помощью программы stack2img было получено схематичное изображение их стекинг-взаимодействия.
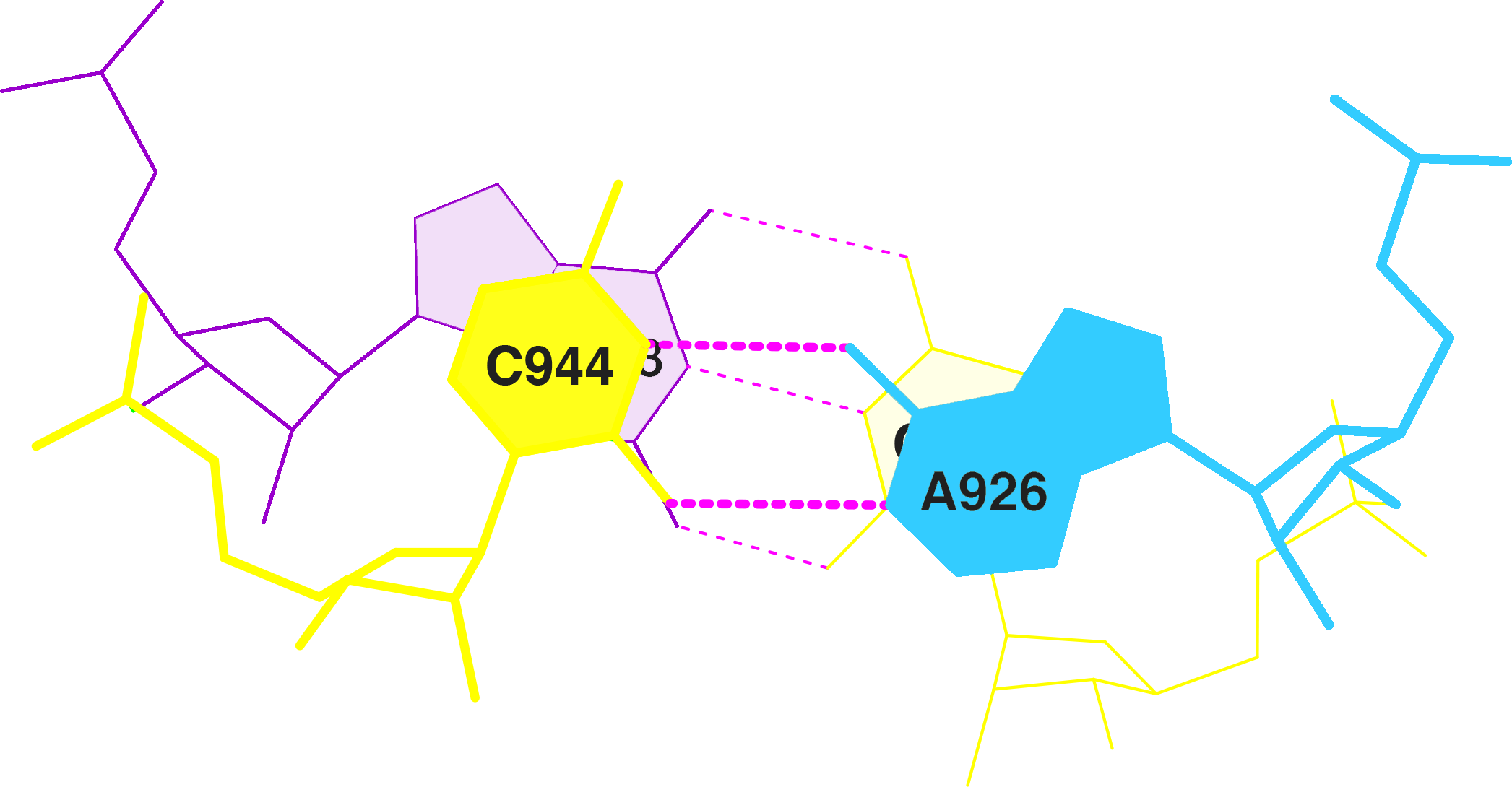
Рисунок 2. Стекинг-взаимодействие двадцатой пары
С помощью программы fiber пакета 3DNA были получены 3 сруктуры A-, B- и Z-форм ДНК (на рисунке 1 модели представлены в том же порядке), коротокие цепи которых составлены из пяти повторов последовательности "GATC". Для Z-формы это десятикратный повтор последовательности "GC".
Скачать полученные структуры можно здесь.