1. Предсказание вторичной структуры тРНК
С помощью инвертированных повторов
Einverted позволяет найти инвертированные участки в молекуле рассматриваемой нуклеиновой кислоты. Запуск программы со стандартными входными параметрами не дал никаких результатов, поэтому было решено уменьшить Minimum match score с 50 до 25, значения других параметров остались по умолчанию. Таким образом, был найден один инвертированный повтор, принадлежащий акцепторному стеблю:
SEQUENCE: Score 21: 7/7 (100%) matches, 0 gaps
1 tggggta 7
|||||||
71 accccat 65
По алгоритму Зукера
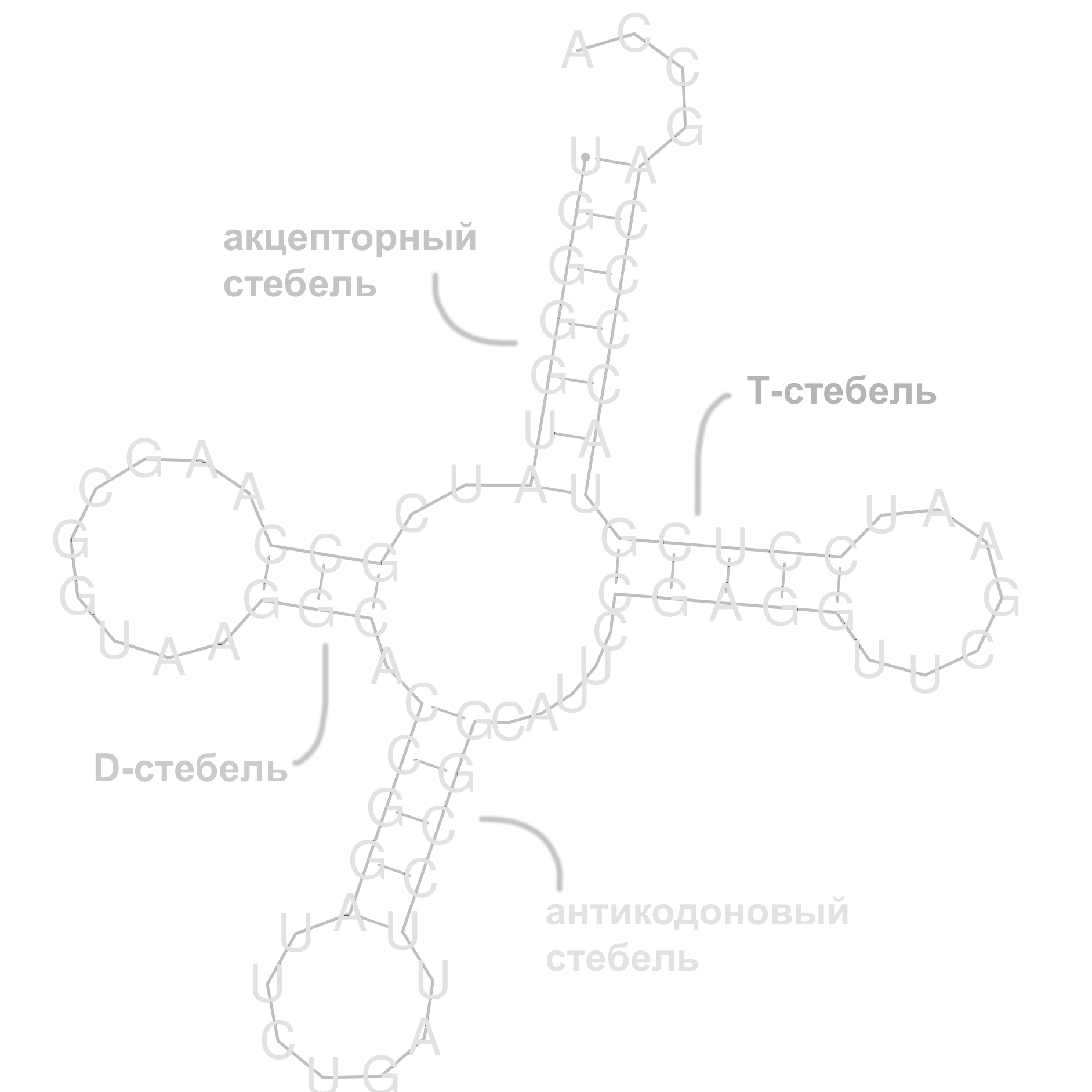
На рисунке 1 представлена вторичная структура тРНК из комплекса 1o0b, предсказанная с помощью алгоритма Зукера. Алгоритм реализуется уже упомянутой выше программой RNAfold. Такое предсказание было получено при первом запуске программы со стандартными параметрами.
В таблице 1 приведены результаты сравнения трех описаний, полученных с помощью программ find_pair | analize, einverted и RNAfold.
Рисунок 1. (справа) Вторичная структура 1o0b, сгенерированная RNAfold
| Участок структуры | find_pair | einverted | RNAfold |
| Акцепторный стебель |
5'-902-907-3' 5'-966-971-3' 6 пар |
7 пар из 7 | 7 пар |
| D-стебель |
5'-910-912-3' 5'-923-925-3' 3 пары |
3 пары | |
| T-стебель |
5'-949-953-3' 5'-961-965-3' 5 пар |
5 пар | |
| Антикодоновый стебель |
5'-941-943-3' 5'-927-929-3' 3 пары |
3 пары | |
| Общее число канонических пар нуклеотидов | 16 | 7 | 17 |
Таблица 1. Реальная и предсказанная вторичная структура тРНК из файла 1P47.pdb
2. Поиск ДНК-белковых контактов
Для поиска ДНК-белковых контактов в заданной структуре 1p47 сначала были определены следующие множества атомов:
1. Множество атомов кислорода 2'-дезоксирибозы (set1)
2. Множество атомов кислорода в остатке фосфорной кислоты (set2)
3. Множество атомов азота в азотистых основаниях (set3)
По следующим ссылкам можно скачать скрипт c определением множеств и c их последовательным отображением в программе Jmol.
Для описания полярных и неполярных ДНК-белковых контактов с помощью скрипта были найдены все пары атомов ДНК и белка на расстоянии 3.5Å и 4.5Å соотвественно.
Результаты представлены в таблице 2:
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 1 | 11 | 12 |
| остатками фосфорной кислоты | 9 | 11 | 20 |
| остатками азотистых оснований со стороны большой бороздки | 14 | 17 | 31 |
| остатками азотистых оснований со стороны малой бороздки | 0 | 0 | 0 |
Таблица 2. Контакты разного типа в комплексе 1P47.pdb
Можно заметить, что большая часть полярных и неполярных контактов приходится на контакты белка с сахарофосфатным остовом. Самыми распространенными среди полярных контактов оказались контакты между белком и кислородами фосфатной группы, среди неполярных - взаимодействия между белком и неполярными атомами оснований, направленных в сторону большой бороздки. Небольшую долю контактов занимают контакты белка с остатками 2'-дезоксирибозы - всего 6. Для оснований, обращенных к малой бороздке, ни полярных, ни неполярных контактов найдено не было.
Nucplot
Ниже представлена схема ДНК-белковых контактов - результат работы программы nucplot, установленной на kodomo:
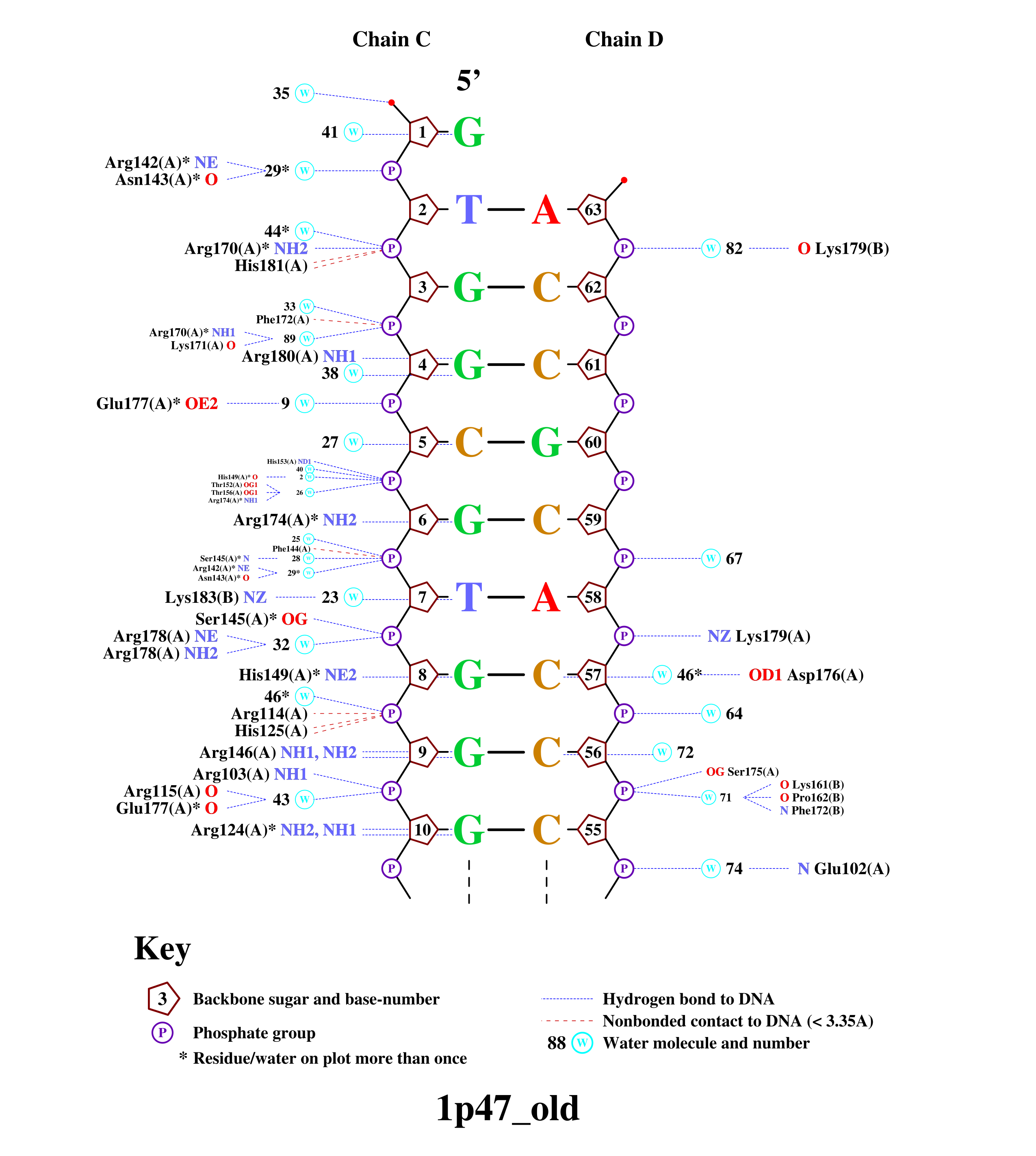
Согласно схеме, составленной nucplot, наибольшее количество контактов c ДНК имеет остаток Arg170(A) - два контакта, один непосредственно с молекулой ДНК, другой - при участии молекулы воды. Этот же остаток я считаю наиболее важным для распознования последовательности ДНК, так как на него приходится наибольшее число контактов с разными основаниями. Визуализация упомянутых контактов представлена на рисунке 2.
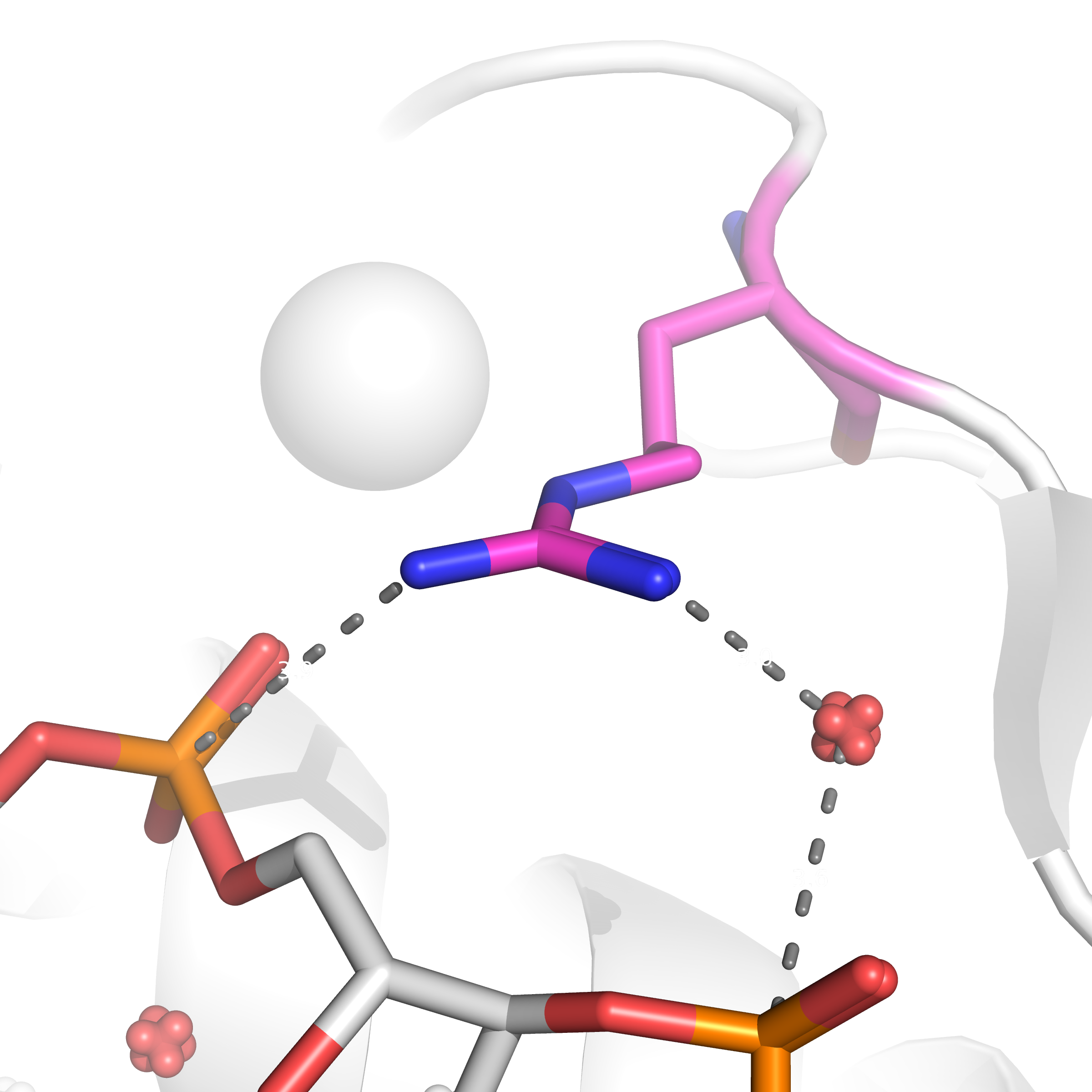
Рисунок 2. Визуализация контактов Arg170(A) с ДНК
В прошлом практикуме с помощью программ find_pair и analyze были получены сведения о реальной вторичной структуре заданной тРНК. В этом задании мы сравним их с выдачей программ einverted из пакета EMBOSS и RNAfold из пакета Viena Rna Package.