Скобочная формула дерева: (((А:50,B:50):40,(C:70,D:70):30):15,(E:60,F:60):4);
Полученное дерево:
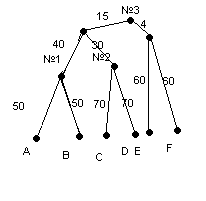
Скобочная формула дерева: (((А:50,B:50):40,(C:70,D:70):30):15,(E:60,F:60):4);
Полученное дерево:

| A | B | C | D | E | F | |
| №1 | - | - | + | + | + | + |
| №2 | + | + | - | - | + | + |
| №3 | + | + | + | + | - | - |
Длина гена - 1362 п.о.
Формула подсчета: Число мутаций= (1362/100) *(длина соответствующей ветви), так как длина ветви(эволюционное расстояние)
дано с рассчетом на 100 нуклеотидов.
Текст скрипта:
msbar ecoli_gene.fasta ABCD.fasta -point 4 -count 204 -auto msbar ecoli_gene.fasta EF.fasta -point 4 -count 54 -auto msbar ABCD.fasta AB.fasta -point 4 -count 545 -auto msbar ABCD.fasta CD.fasta -point 4 -count 409 -auto msbar AB.fasta A.fasta -point 4 -count 681 -auto msbar AB.fasta B.fasta -point 4 -count 681 -auto msbar CD.fasta C.fasta -point 4 -count 953 -auto msbar CD.fasta D.fasta -point 4 -count 953 -auto msbar EF.fasta E.fasta -point 4 -count 817 -auto msbar EF.fasta F.fasta -point 4 -count 817 -auto
Мутантные последовательности были помещены в один файл mut.fasta в виде "выравнивания". Эта программа требует на вход только само выравнивание, не учитывает попарные расстояния между мутантами.
Результатом действия данного метода является филограмма (бескорневая!!) следущего вида:
Чтобы реконструировать дерево алгоритмами UPGMA или Neighbor-joining, необходимо посчитать попарные расстояния между последовательностями
программой fdnadist:
Алгоритм UPGMA дал в результате кладограмму следущего вида:
Как можно заметить, это дерево имеет корень.
Алгоритм Neighbor-joining дал в результате бескорневую филограмму следущего вида:
Представим внутренние ветви всех деревьев как разбиения листьев и сравним их:
Как видно из сравнения, все деревья одинаковы и совпадают с исходным по разбиениям.
fdnaml mut.fasta -ttratio 1 -auto
+------------B
|
| +--------------F
| +---4
| | +----------------E
1----------3
| | +----------------D
| +------2
| +---------------C
|
+---------A
fdnadist mut.fasta -ttratio 1 -auto
Полученный файл mut .fdnadist был подан на вход программе fneighbor:
fneighbor mut.fdnadist -auto
для реконструкции алгоритмом Neighbor-joining и
fneighbor mut.fdnadist -treetype u -auto
для реконструкции алгоритмом UPGMA.
+---------------------A
+--------------------1
! +---------------------B
+--4
! ! +------------------------------E
! +-----------2
--5 +------------------------------F
!
! +---------------------------------C
+-----------3
+---------------------------------D
+------------B
!
! +---------------C
! +-------2
! ! +-----------------D
1-----------3
! ! +----------------E
! +---4
! +-------------F
!
+---------A
A B C D E F
Исходное дерево
Алгоритм максимального правдоподобия
Алгоритм UPGMA
Алгоритм Neighbor-joining
. . * * * *
+ + + + * * . . * *
+ + + + * * * * . .
+ + + +
На главную страницу >>>
© Гурьянова Наталья Николаевна