Задания по предсказанию генов эукариот
1. Описание контига и одного из генов на нем, для которого предсказан альтернативный сплайсинг
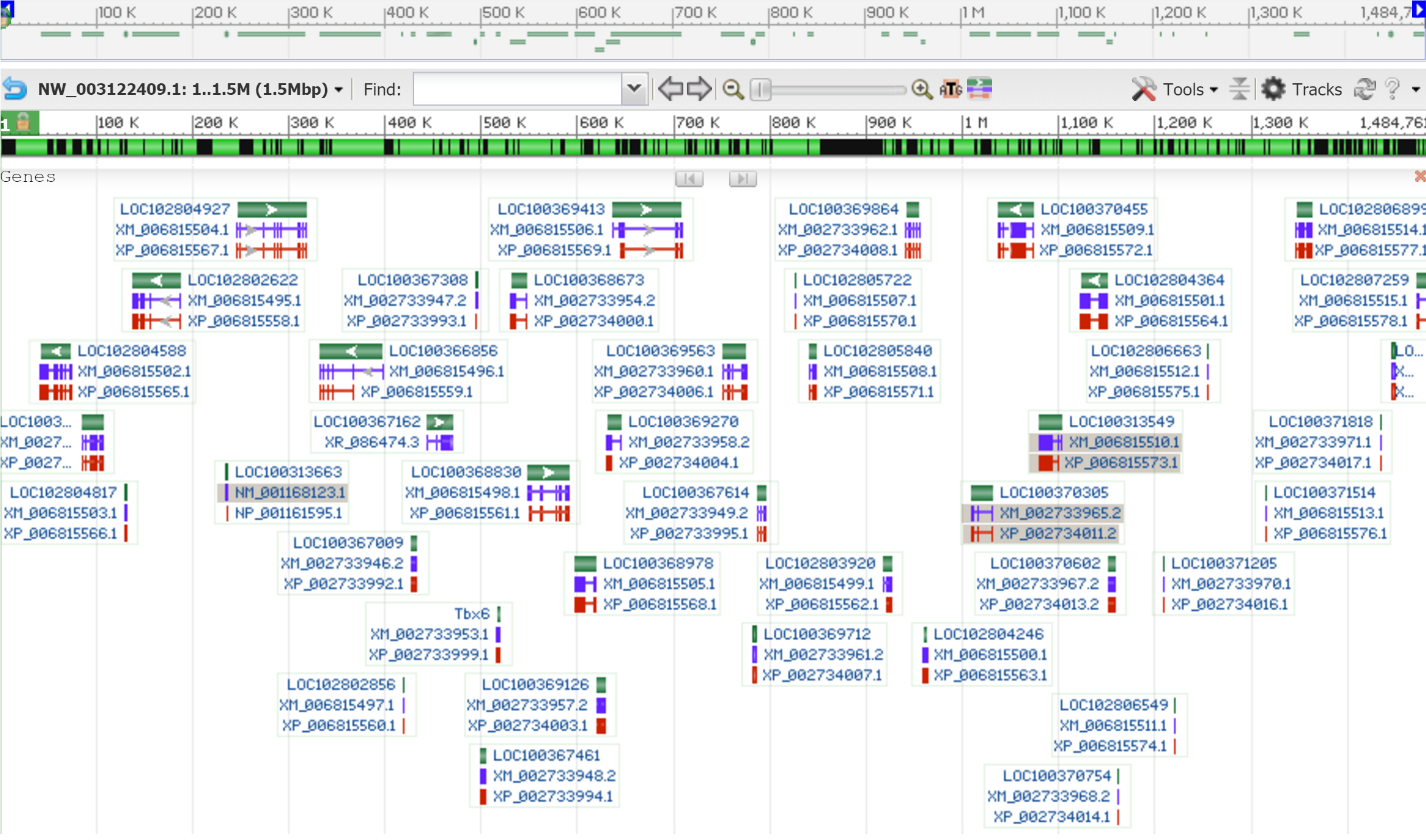
В выданном мне контиге и идентификатором NW_003122409 альтернативного сплайсинга не предсказано (рис. 1). Поэтому я взяла один из резервных контигов: с идентефикатором NW_016683393.
Он принадлежит Xenopus tropicalis штамма Nigerian- когтистой шпорцевой лягушке (рис. 2) - важному модельному организму. Таксономия объекта представлена в таблице №1.
|
|
| Рис. 1 Изображение контига с идентификатором NW_003122409 |
|
|
||||||||||||||||||||||||||||||||||||||||||||||||
| Рис. 2 Xenopus tropicalis | ||||||||||||||||||||||||||||||||||||||||||||||||
| Таблица №1 Таксономия |
|
| Царство | Animalia |
| Тип | Chordata |
| Класс | Amphibia |
| Порядок | Anura |
| Семейство | Pipidae |
| Род | Xenopus |
| Вид | X. tropicalis |
В геномном браузере NCBI был найден нужный скаффолд
(ссылка на последовательность в формате fasta).
Длина скаффолда составляет 118935 пар оснований. Всего в нем содержится 27 генов, 26 из которых кодируют белки. Для шести генов известен альтернативный сплайсинг.
Вручную был найден один из таких генов: slc4a4. Всего с этом гене 28 экзонов. Транскрибируется 3 мРНК,
с которых транслируются 3 различные изоформы белка slc4a4 (electrogenic sodium bicarbonate cotransporter).
| мРНК | Изоформа и идентификатор |
| XM_004919133.3 | X1, XP_004919190.2 |
| XM_004919135.3 | X2, XP_002940622.3 |
| XM_002940576.4 | X3, XP_004919192.2 |
Электрогенный со-транспортер бикарбоната натрия - трансмембранный белок, участвующий в проведении ионов натрия и бикарбона через плазматическую мембану клеток, играет важную роль в поддержании кнутриклеточного pH.
Это электрогенный процесс, требующий точных стехиометрических соотношений транспортируемых ионов. Белок состоит из трех консервативных доменов: ae, HCO3_cotransp и Band 3 cytoplasmic domain.
Краткая аннотация доменов:
AE - ионообменный протеин соответствующего семейства - катализирует антипорт, то есть согласованный перенос двух растворов через мембрану в противоположных направлениях, в особенности HCO3-:Cl-. Найден только у животных.
Также является со-транспортером Na+:HCO3-, то есть переправляет одно по, а другое против градиента концентрации, катализирует реабсорбцию HCO3- в проксимальных канальцах почек.
HCO3_cotransp - семейство со-транспортеров HCO3-.
Band 3 cytoplasmic domain - играет роль якоря в мембранных белках.
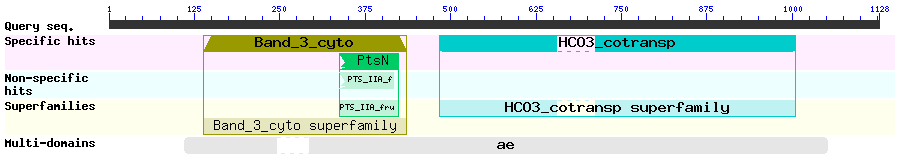
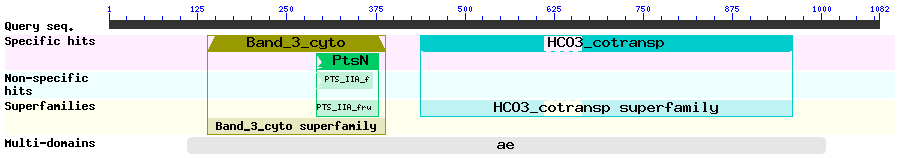
На рисунках 4, 5 и 6 представлены расположение доменов в изоформах белка slc4a4. Видно, что неизменным по длине остается только домен HCO3_cotransp.
|
|
|
Рис. 4 Домены изоформы X1 белка slc4a4 |
|
|
|
Рис. 5 Домены изоформы X2 белка slc4a4 |
|
|
|
Рис. 6 Домены изоформы X3 белка slc4a4 |
2. Предсказание генов и белок-кодирующие области в контиге
Для предсказания генов был использован сервер AUGUSTUS в режиме prediction. Входные параметры представлены на рисунке 7.
|
|
|
Рис. 7 Входные параметры для AUGUSTUS |
В качестве модели был выбран вид Danio rerio, как самый близкий из предложенных. Параметры модели необходимы для того,
чтобы получить требуемое предсказание.
Предсказание UTR (предсказание нетранслируемых участков) для выбранной модели недоступно, поэтому выбрано значение параметра false.
Посколько в предыдущем задании было выяснено наличие альтернативно сплайсируемых генов, в соответствующем поле был выбран параметр medium, их учитывающий.
Остальные параметры взяты по умолчанию.
В результате работы был получен архив predictions.tar.gz с шестью файлами (таблица №2).
| Таблица №2 Описание полученных файлов |
|
| Расширение | Содержание |
| augustus.gtf | Предсказание генов в формате gtf (второй версии gff) |
| augustus.gff | Предсказание генов в обычном формате gff (General Feature Format) |
| augustus.gbrowse | Файл с координатами для Gbrowse |
| augustus.codingseq | Предсказание кодирующих регионов ДНК в формате fasta |
| augustus.cdsexons | Предсказание экзонов в формате fasta |
| augustus.aa | Последовательности белков в формате fasta, полученные после трансляции предсказанных генов |
Из GenBank была скачана таблица с параметрами контига в формате gff3 (xen_features.gff3).
С помощью команды
grep 'CDS' xen_features.gff3 | awk '{print $4,$5,$7}' >> info_xen_gb.txt
из файла с особенностями был получен файл info_xen_gb.txt, удобный для дальнейшей работы. Аналогичный файл aug.txt для предсказаний AUGUSTUS
был получен командой
grep 'CDS' augustus.gff | awk '{print $4,$5,$7}' >> aug.txt.
Далее скриптом kek.py из практикума 11 было проведено сравнение, в результате которого было обнаружено, что AUGUSTUS ни одного гена не предсказал правильно.
Причем даже по отдельности правильно предсказанных N- или C-концов не оказалось.
Для гена slc4a4 предсказание, очевидно, также не совпало, причем данный ген находится в районе 1400000, а предсказание AUGUSTUS заканчивается в районе 1300000 нуклеотиов.