A- и В- формы ДНК. Структура РНК
Задание 1. Построение модели структур A-, B- и Z-формы ДНК с помощью инструментов пакета 3DNA
С помощью инструментов пакета 3DNA построены модели структур A-, B- и Z-формы ДНК. Программой fiber созданы А-, B- и Z-формы дуплексов ДНК,
последовательность одной из нитей которых представляет собой 5 раз повторенную последовательность "gatc"
(за исключение Z-формы ДНК, нить которой состоит из повторенной 10 раз последовательности GC).
Структурf дуплекса в А-форме сохранена в файле gatc-a.pdb, структура дуплекса в В-форме - в файле gatc-b.pdb,
в Z-форме - в файле gatc-z.pdb.
Задание 2. Сравнение структур 3-х форм ДНК с помощью средств JMol
Упр. 1
Цель - научиться находить большие и малые бороздки.
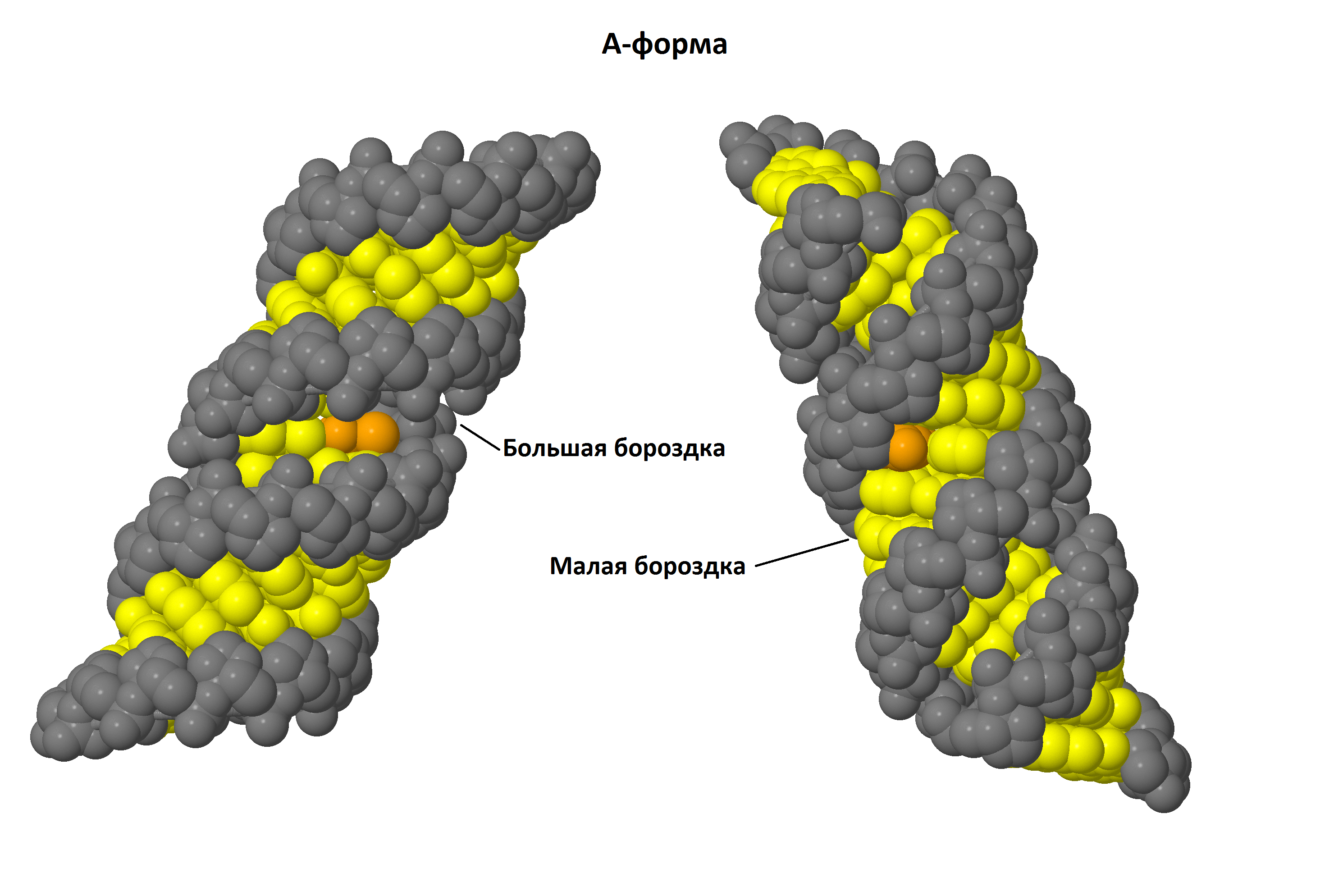
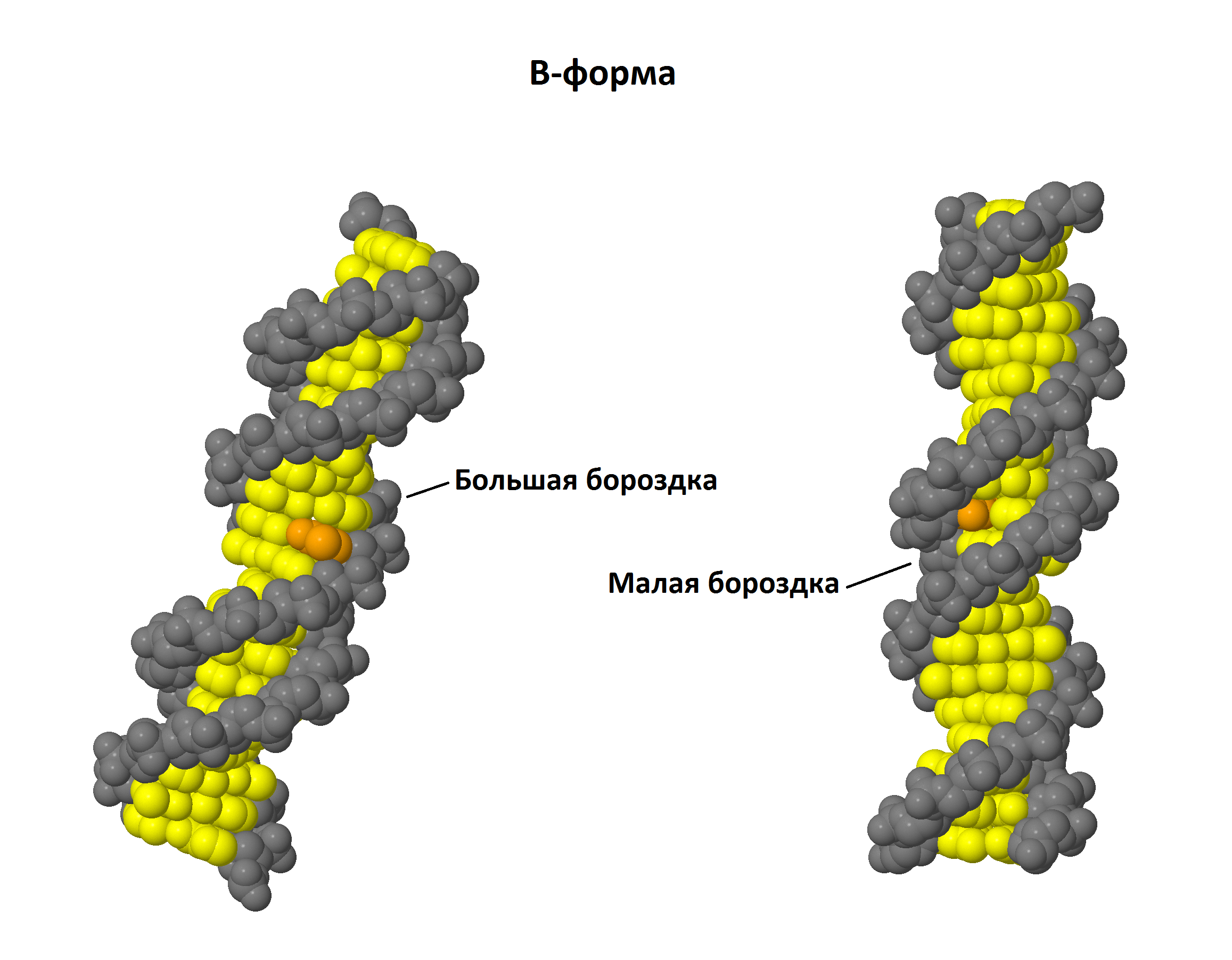
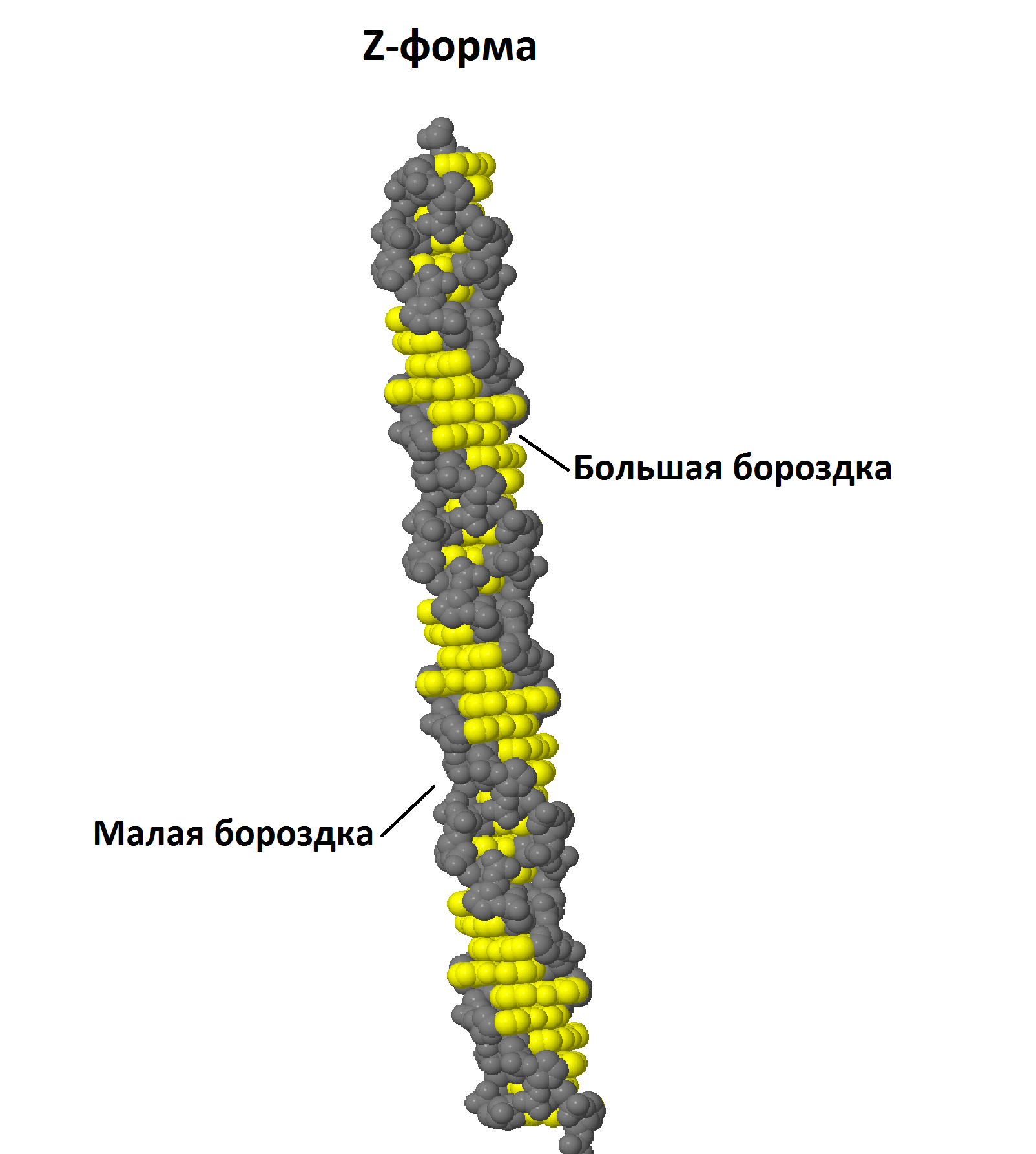
С помощью JMol были получены изображения А- и В-форм ДНК. Рис. 1, 2 и 3 демонстрируют большую и малую бороздки в двухцепочечных моделях ДНК.
Остов обозначен серым, основания - желтым, один из остатков тимина - оранжевым.
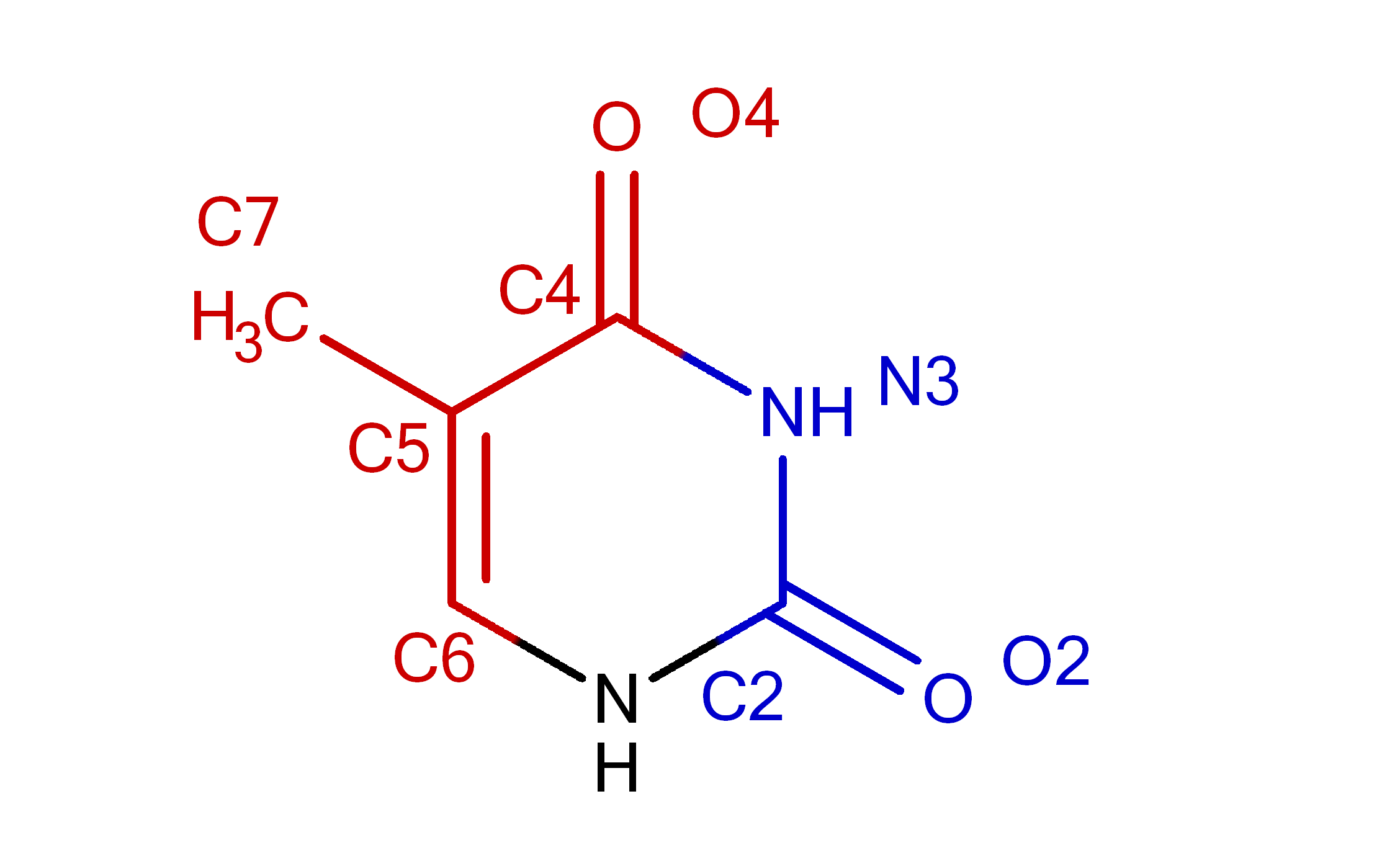
На рис. 4 представлено полученное в MarvinSketch основание,
где выделины красным цветом атомы, смотрящие в сторону большой бороздки, синим - в сторону малой.
В таблице №1 преставлено, куда обращены атомы тимина в А- и В-формах ДНК. В Z-структуре тимина нет.
|
|
| Рис. 1 А-форма |
|
|
| Рис. 2 B-форма |
|
|
| Рис. 3 Z-форма |
|
|
| Рис. 4 Тимин |
|
Таблица №1 Расположение атомов тимина |
||
| B-форма | A-форма | |
| Малая бороздка, имена атомов | O2, C2, N3 | O2, C2, N3 |
| Большая бороздка, имена атомов | O4, C7, C4, C6, C5 | O4, C7, C4, C6, C5 |
| Остальные атомы | N1 | N1 |
Упр. 2 Цель - сравнить основные спиральные параметры разных форм ДНК. На 3D модели JMol были изучены структуры A-, B- и Z-форм. Результаты представлены в таблице №2.
|
Таблица №2 Сравнение основных спиральных параметров |
|||
| A-форма | B-форма | Z-форма | |
| Тип спирали | правая | правая | левая |
| Шаг спирали (Å) | 28,3 | 33,75 | 43,5 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки | 18,5 ([DC]12:A - [DG]33:B) | 20,58 ([DA]26:B - [DG]9:A) | 23,51([DC]56:B - [DC]20:A) |
| Ширина малой бороздки | 9,63 ([DT]27:B - [DT]7:A) | 13,2 ([DT]27:B - [DT]19:A) | 9,87 ([DC]32:A - [DG]53:B) |
Задание 3. Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA
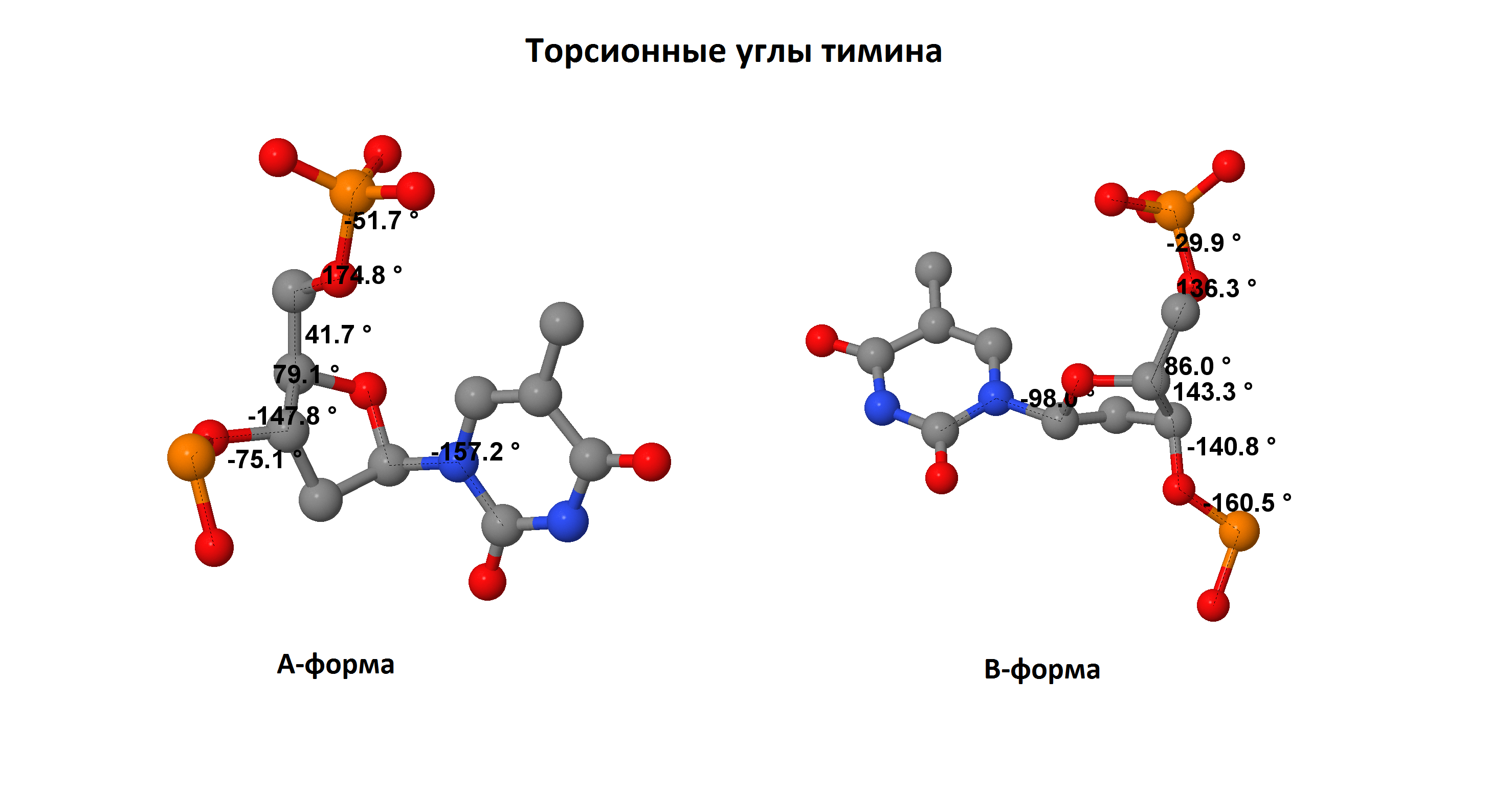
Упр. 1 Цель - сравнить торсионные углы в структурах ДНК.
Были сравнены значения торсионных углов в структурах А-, В- и Z-форм ДНК. Для форм А- и В- результаты были получены с помощью JMol (рис. 5).
При помощи команд find_pair и analyze были получены значения торсионных углов для Z-формы ДНК (файл gatc-z.out).
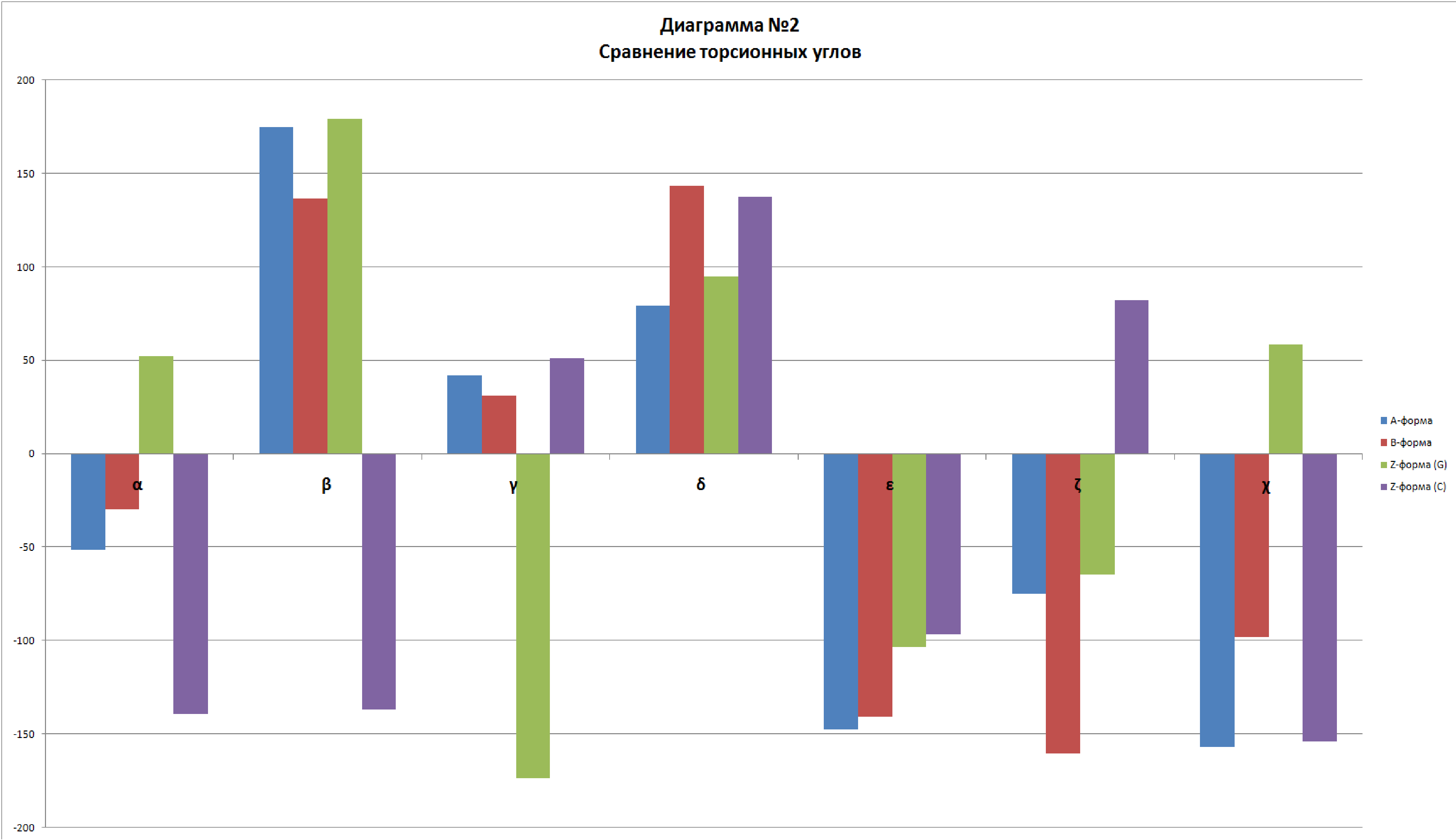
Видно, что торсионные углы в A- и B-форм ДНК не зависят от нуклеотида. Z-форма имеет два различных значения каждого угла, в зависимости от нуклеотида (таблица №3, диаг. №1).
Торсионные углы одинаковы двя обеих цепей ДНК во всех трех структурах (в отличие от РНК).
Больше всего отличаются углы δ A- и B-форм (почти в 2 раза), что связано с конформациями дезоксирибозы: 3'-эндо у A-ДНК и 3'-экзо у В-ДНК
(атом C3' "торчит" из плоскости сахара в сторону атома C5'- эндоконформация, в противоположную - экзо), и углы ζ (больше чем в 2 раза).
Из таблицы и диаграммы №1 видно, что торсионные углы А- и В-форм совпадают по знаку.
Торсионные углы Z-формы ДНК отличаются сильно от двух других форм, в том числе по знаку (логично, учитывая, что Z-ДНК - левозакрученная).
|
|
| Рис. 5 Торсионные углы |
|
Таблица №3 Сравнение торсионных углов |
|||
| А-форма | В-форма | Z-форма (для G и C соответственно) | |
| α (O3'-P-O5'-C5') | -51,7 | -29,9 | 52,0 и -139,5 |
| β (P-O5'-C5'-C4') | 174,8 | 136,3 | 179,0 и -136,7 |
| γ (O5'-C5'-C4'-C3') | 41,7 | 31,1 | -173,8 и 50.9 |
| δ (C5'-C4'-C3'-O3') | 79,1 | 143,3 | 94.9 и 137.6 |
| ε (C4'-C3'-O3'-P) | -147,8 | -140,8 | -103.6 и -96.5 |
| ζ (C3'-O3'-P-O5') | -75,1 | -160,5 | -64.8 и 81.9 |
| χ (O4'-C1'-N1-C2) | -157,2 | -97,9 | 58.7 и -154.3 |
|
|
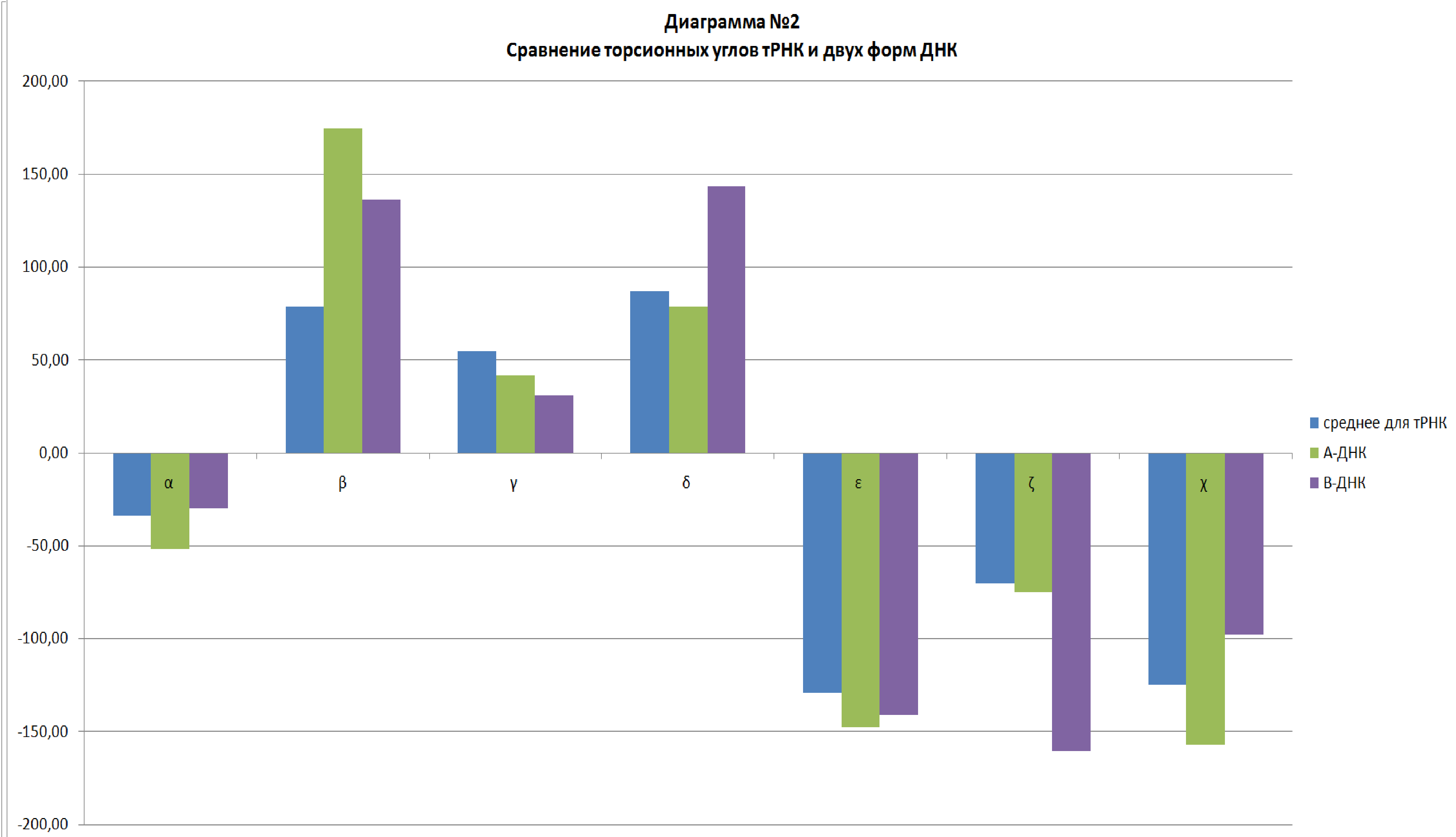
Далее были определены значения торсионных углов в структуре тРНК (1gtr). С помощью команд find_pair и analyze эти данные были получены
(файл rna.out). На их основе составлена таблица №5 и диаграмма №2,
где представлены средние значения всех торсионных углов, характерных для данной тРНК, а также для 2-х форм ДНК.
Анализируя таблицу и диаграмму, можно сделать вывод, что торсионные углы тРНК в большей степени схожи с углами в А-ДНК.
Для всех нуклеотидов (и сравнивая с ДНК) достаточно консервативен только угол дельта.
Конформации рибозы различные: C3'-endo, C3'-exo, C4'-exo, C2'-exo, C2'-endo. Максимум - 72,74% - составляет C3'-endo.
Такой же вывод можно сделать и анализирую диаграмму №2: по углу дельта больше всего совпадений с А-формой ДНК, где, как известно, сахар представлен 3'-эндоконформацией.
Средние значения торсионных углов были вычислены при помощи программы Excel, без учета крайних нуклеотидов.
Самый "деформированный" нуклеотид - 7-ой гуанин второй цепи, отклонение значений его торсионных углов от среднего оказалось наибольшим.
|
Таблица №4 Сравнение торсионных углов РНК и ДНК |
|||||||
| α | β | γ | δ | ε | ζ | χ | |
| Среднее для тРНК | -33,63 | 78,75 | 54,76 | 86,78 | -129,31 | -70,15 | -125,06 |
| А-ДНК | -51,7 | 174,8 | 41,7 | 79,1 | -147,8 | -75,1 | -157,2 |
| B-ДНК | -29,9 | 136,3 | 31,1 | 143,3 | -140,8 | -160,5 | -97,9 |
|
|
Программами find_pair и analyze были найдены углы в структуре ДНК 1rh6. С помощью Excel были определены средние значение каждого из торсионных углов, не учитывая краевые нуклеотиды (таблица №5). В заданной структуре ДНК самый "деформированный " - 12-й нуклеотид (T) первой цепи.
Упр. 2 Цель - научиться определять структуру водородных связей.
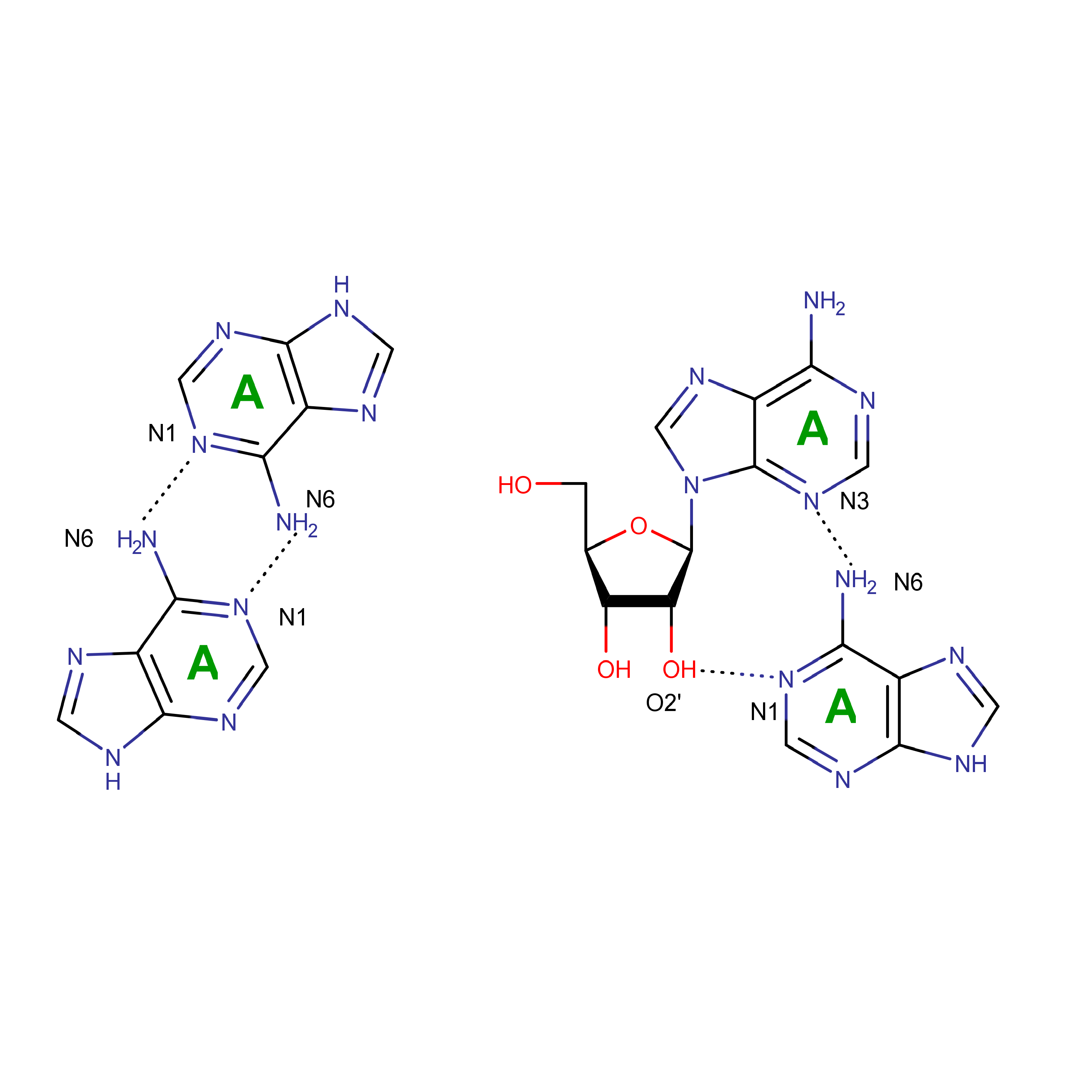
В файле rna.out найдена информация о водородных связях внутри молекулы тРНК (таблица №6). В таблице "-" обозначены каноничные водородные взаимодействия, "*" - неканоничные.
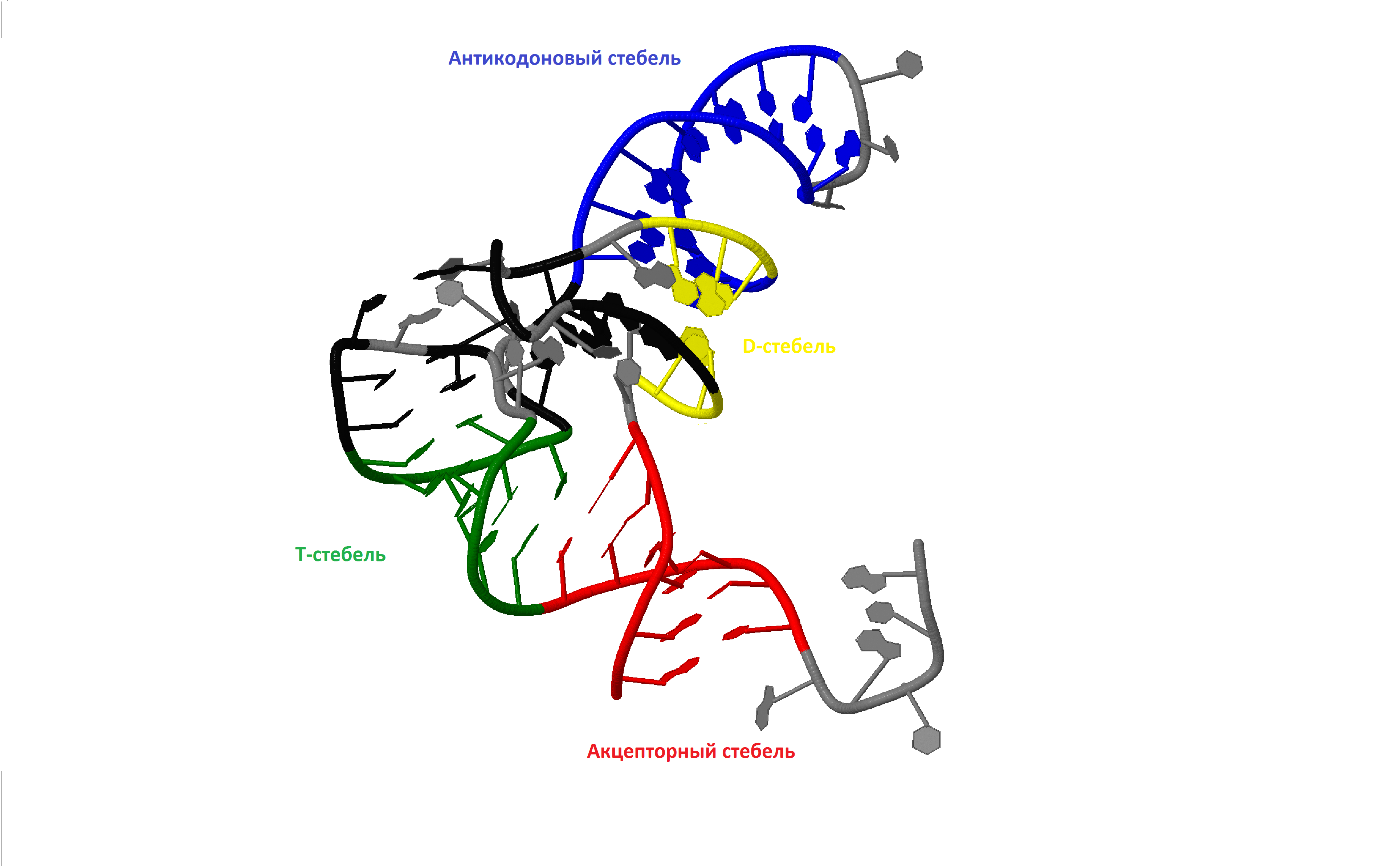
Чтобы определить номера нуклеотидов, образующих стебли во вторичной структуре тРНК, необходимо разобраться в принципах строения тРНК.
Структура характеризуется наличием четырех стеблей и трех петель, которая получила название "клеверного листа".
Акцепторный стебель образован 7 парами нуклеотидов, причем 3' конец длиннее. К оставшемуся "хвосту" присоединяется транспортируемая аминокислота.
T-стебель содержит последовательность TΨC , где Ψ - псевдоуридин. T-пеля содержит нуклеозид тимидина (T).
D-стебель предшествует петле, содержащей дигидроуридин (D). Ближе к 5' концу.
Стебель с петлей формируют ветвь. Антикодоновая ветвь содержит антикодоновый триплет в составе своей петли. Слева и справа от нее расположены D- и T-ветви,
соответственно названные так из-за присутствия в их петлях необычных консервативных нуклеозидов дигидроуридина (D) и тимидина (T).
Антикодонный стебель оканчивается одноименный петлей, состоящей из 7 нуклеотидов, включая модифицированный пурин и варьирующее основание с одной стороны и два пиримидиновых – с другой стороны.
В дополнение к трем петлям клеверного листа в структуре тРНК выделяют также дополнительную V-петлю - между T- и антикодоновыми стеблями. Ее размеры различаются у разных тРНК.
На рис. 6 и в таблице №5 акцепторный стебель обозначен красным, Т-стебель - зеленым, антикодоновый - синим, D-стебель - желтым.
Дополнительные водородные связи в тРНК, стабилизирующие ее третичную структуру - водородные связи, не имеющие отношения к стеблям в таблице и на рисунке обозначены черным.
|
Таблица №5 Водородные связи |
|||||
| № остатка | Остаток | Взаимодействие | Остаток | № остатка | Структура |
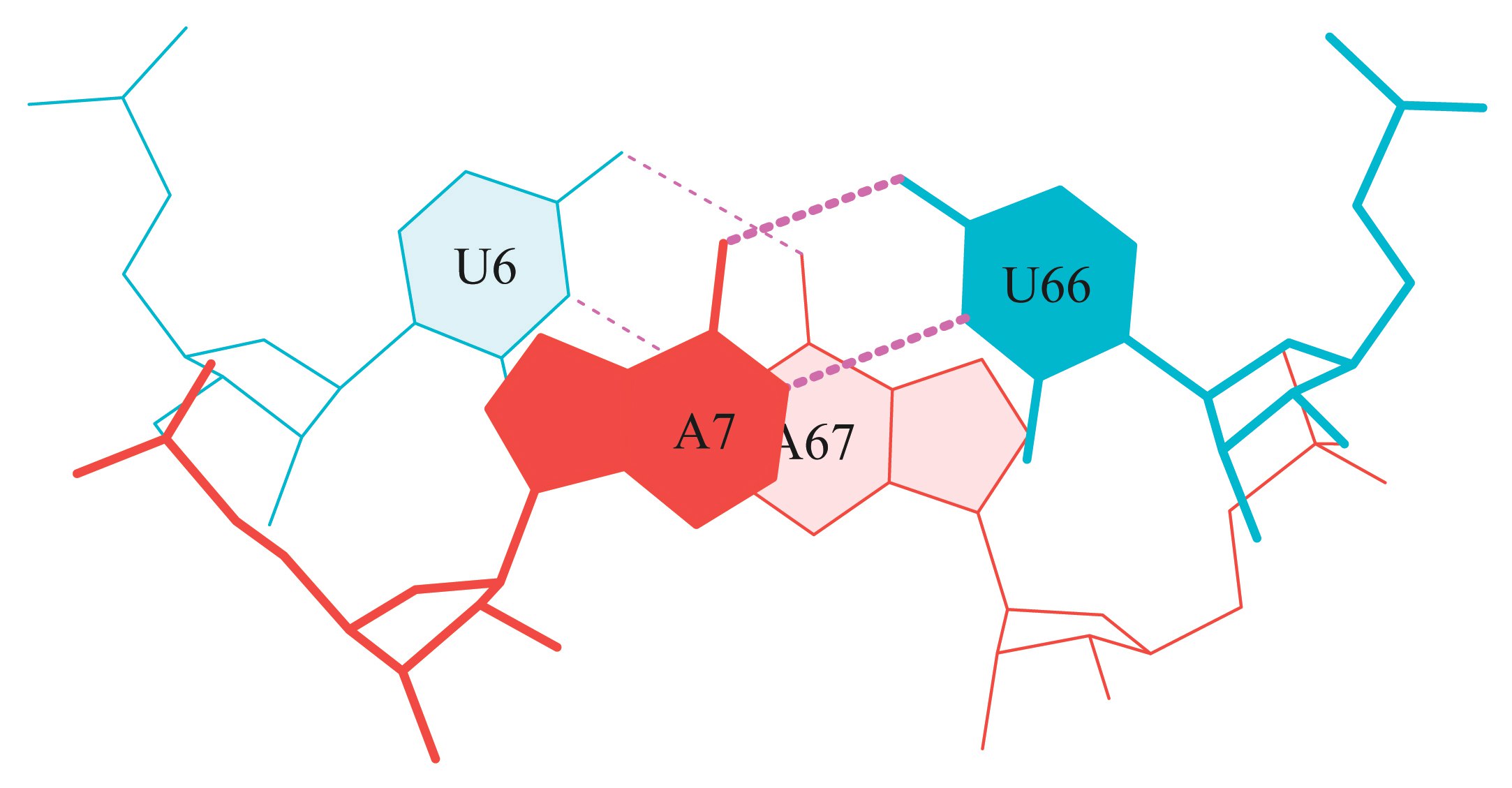
| 2 | G | - | C | 71 | Акцепторный стебель |
| 3 | G | - | C | 70 | |
| 4 | G | - | C | 69 | |
| 5 | G | - | C | 68 | |
| 6 | U | - | A | 67 | |
| 7 | A | - | U | 66 | |
| 49 | C | - | G | 65 | T-стебель |
| 50 | G | - | C | 64 | |
| 51 | A | - | U | 63 | |
| 52 | G | - | C | 62 | |
| 53 | G | - | C | 61 | |
| 54 | U | * | A | 58 | |
| 55 | U | * | G | 18 | |
| 37 | A | * | U | 33 | Антикодоновый стебель |
| 38 | U | * | U | 32 | |
| 39 | U | - | A | 31 | |
| 40 | C | - | G | 30 | |
| 41 | C | - | G | 29 | |
| 42 | G | - | C | 28 | |
| 43 | G | - | C | 27 | |
| 44 | C | * | A | 26 | |
| 10 | G | - | C | 25 | D-стебель |
| 11 | C | - | G | 24 | |
| 12 | C | - | G | 23 | |
| 13 | A | * | A | 45 | |
| 14 | A | * | A | 21 | |
| 15 | G | * | C | 48 | |
| 19 | G | - | C | 56 |
|
|
| Рис. 6 Структура тРНК |
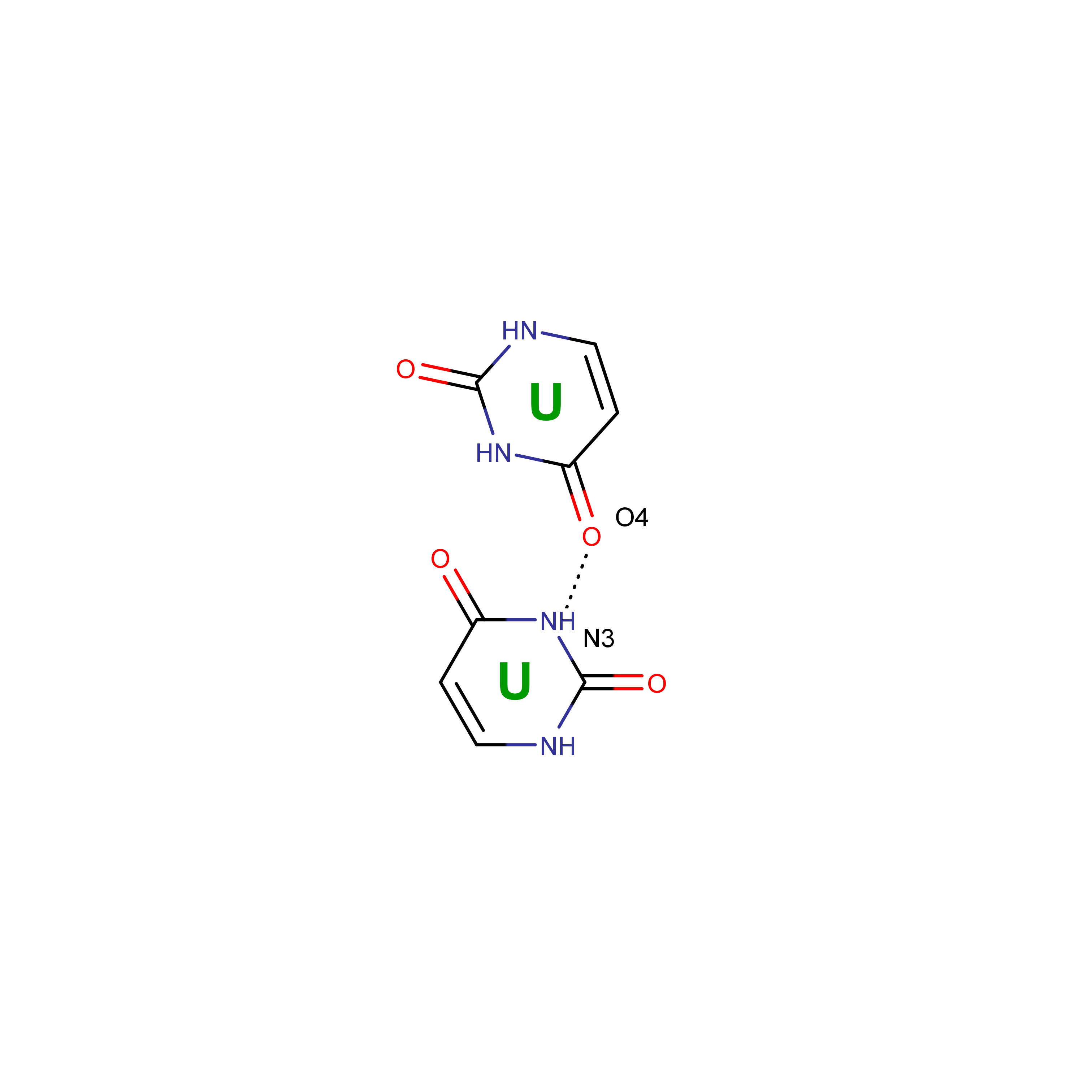
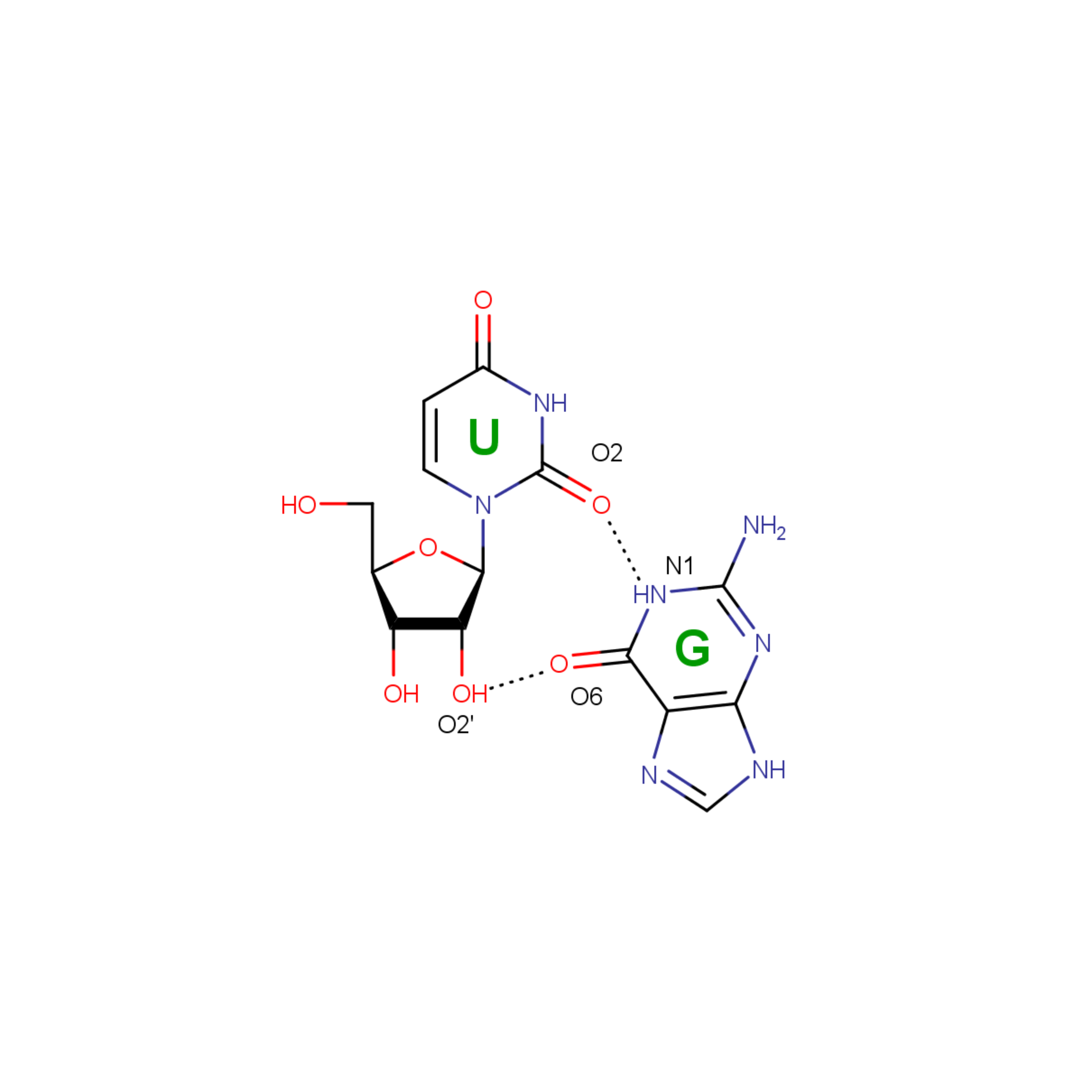
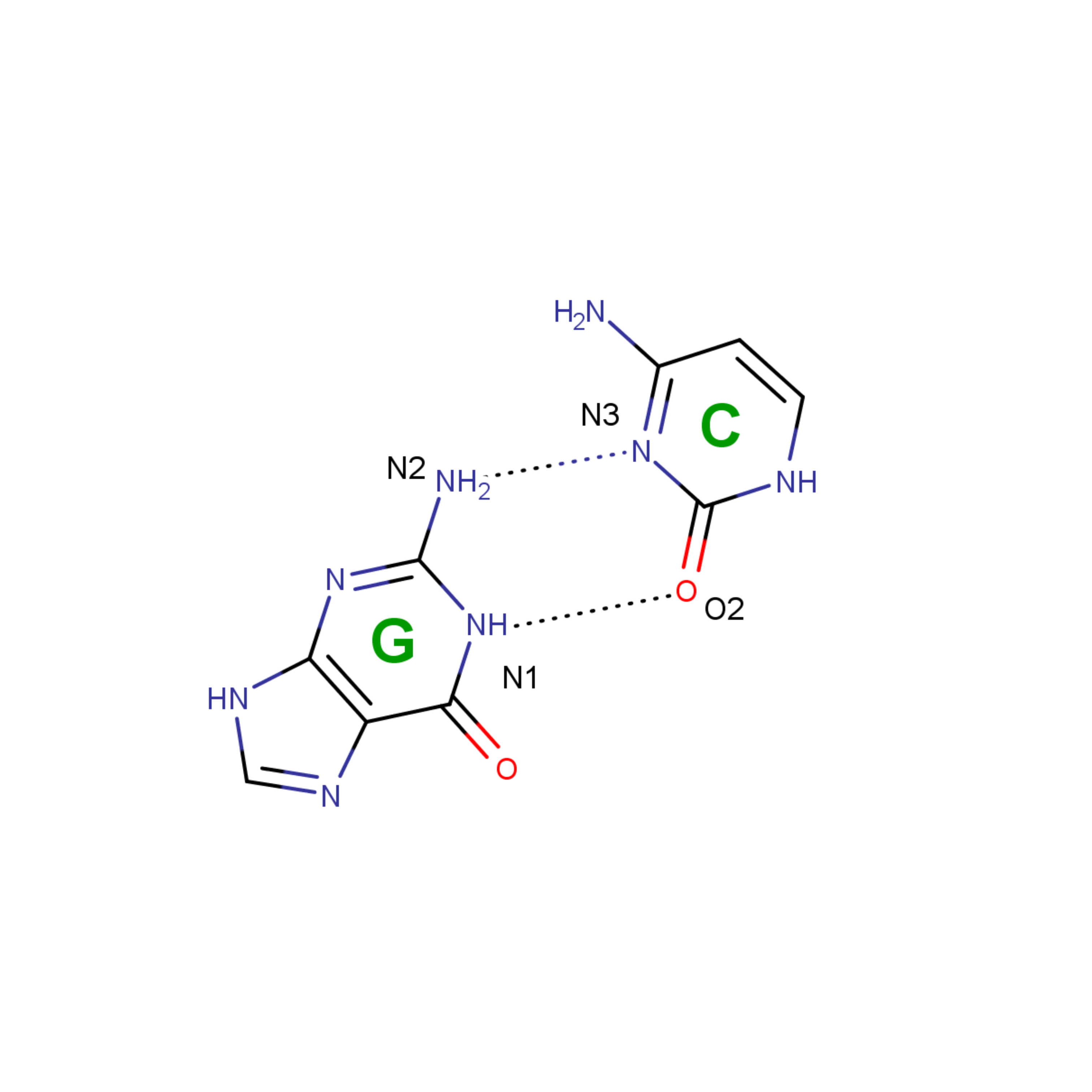
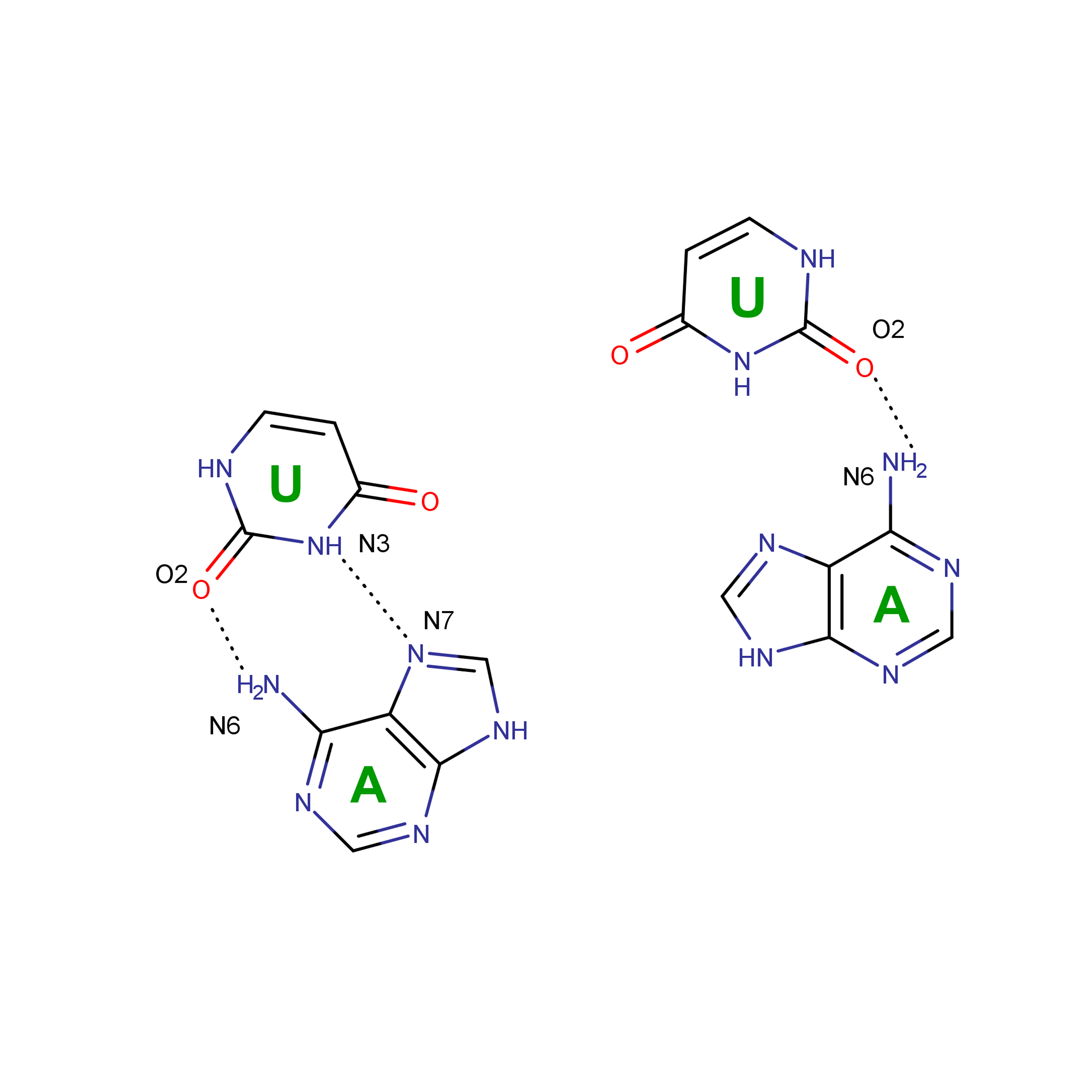
На рисунках 7-11 представлены неканонические взаимодействия, присутствующие в данной тРНК. Надо заметить, что присутствуют как некомплементарные пары, так и комплементарные, но с неканоническими водородными связями.
|
|
|
| Рис. 7 Взаимодействие U-U | Рис. 8 Взаимодействие G-U |
|
|
|
| Рис. 9 Взаимодействие G-C | Рис. 10 Взаимодействие A-U |
|
|
| Рис. 11 Взаимодействие A-A |
Упр. 3 Цель - Научиться находить возможные стекинг-взаимодействи.
В файле stacking.pdb, полученном после действия программы analyse, записаны преобразованные координаты динуклеотидных пар, между которыми осуществляется стэкинг-взаимодействие.
В файле rna.out найдена информации о площади перекрытия между двямя последовательными парами оснований.
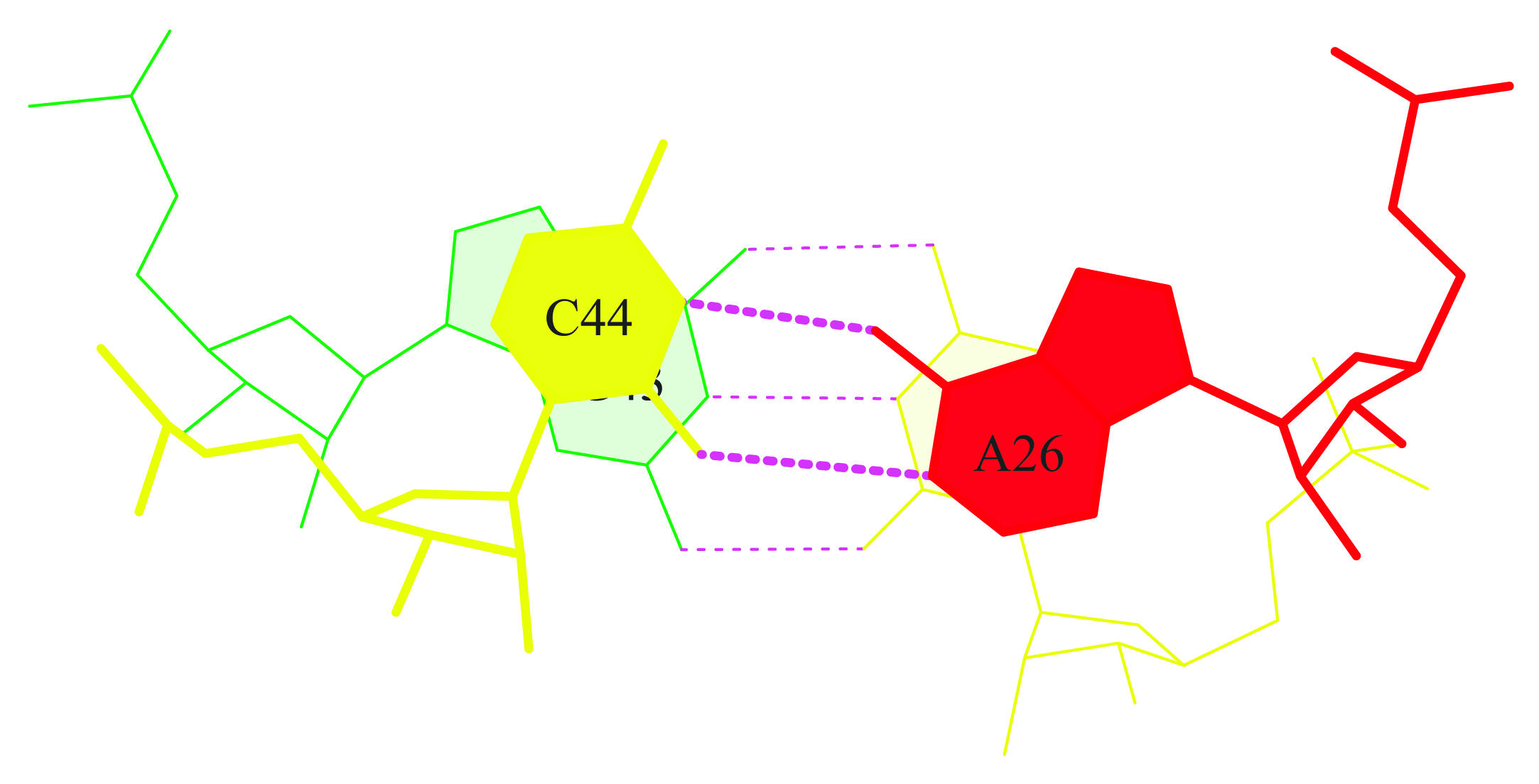
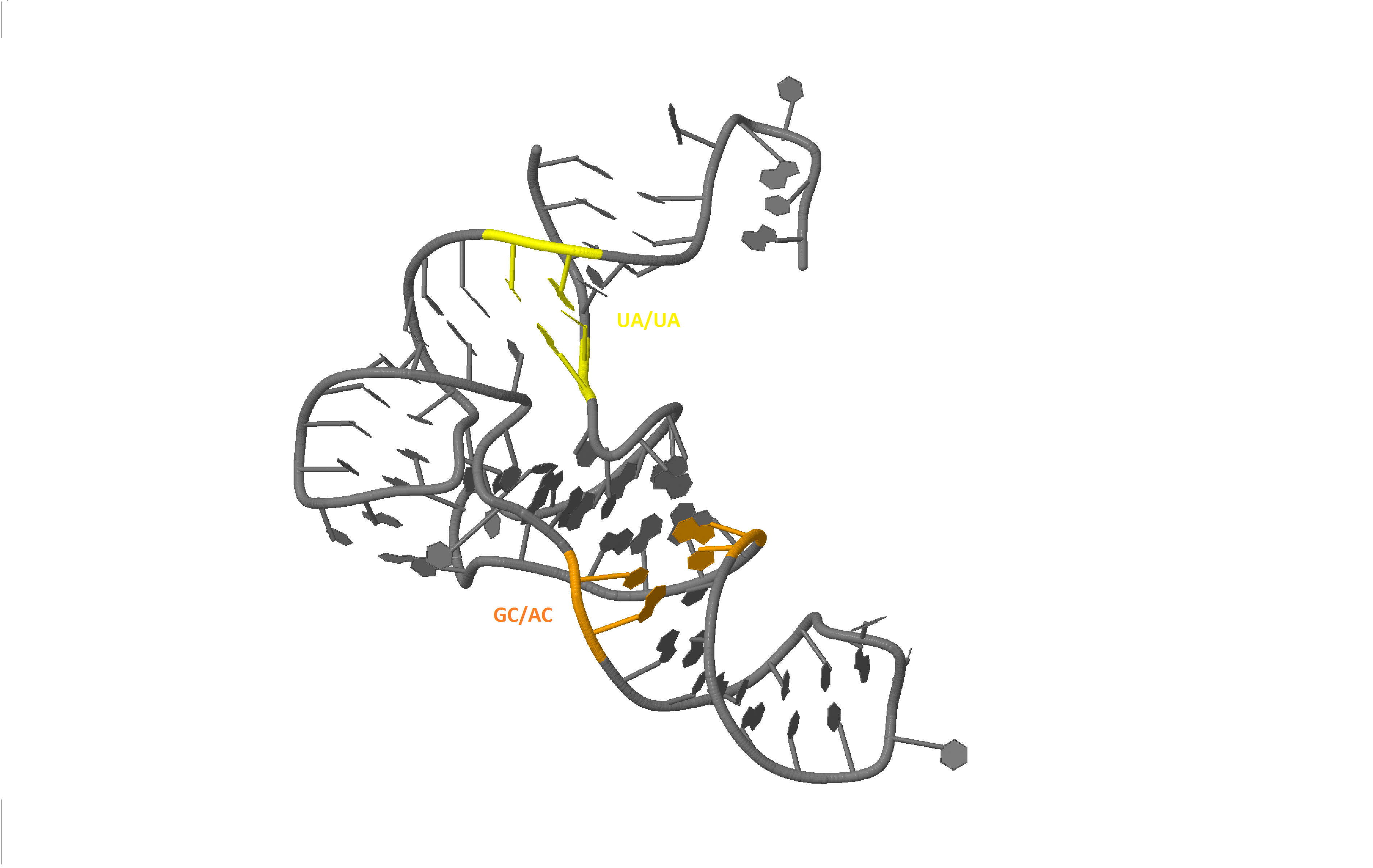
Видно, что максимальная площадь перекрытия у 20-й пары (GC/AC), минимальная - у 5-й (UA/UA). Структуры вырезаны в отдельный файл с помощью команды ex_str.
С помощью программы stack2img созданы стандартное изображение стекинг-взаимодействия этих двух пар (рис. 12 и 13).
|
|
|
| Рис. 12 Наибольшая площадь перекрывания | Рис. 13 Наименьшая площадь перекрывания |
|
|
| Рис. 14 Ориентация GC/AC и UA/UA пар в тРНК |