Комплексы ДНК-белок
Задание 1. Предсказание вторичной структуры заданной тРНК
Упр.1. Предсказание вторичной структуры тРНК путем поиска инвертированных повторов
Программа einverted из пакета EMBOSS позволяет найти инвертированные участки в нуклеотидных последовательностях.
Einverted получает на вход нуклеотидную последовательность и выдает sequence_pr13.fasta и sequence.inv файлы, содержащие информацию об
обнаруженных комплементарных участках последовательности и предполагаемых водородных связях на этих участках соответственно.
Запуск программы с парамерами по умолчанию оказался неудачным: файлs оказались пустыми. Посему пришлось подбирать оптимальные параметры.
Параметры, при которых результаты максимально отражали реальность, представлены на рисунке 1. Тем не менее, программе удалось предсказать только 6 пар, причем неканонические взаимодействия она не опеределяет.
В общем, find_pair справилась с задачей гораздо лучше.
|
|
| Рис. 1 Оптимальные параметры для einverted |
Упр.2. Предсказание вторичной структуры тРНК по алгоритму Зукера
На этом этапе использовалась программа RNAfold из пакета Viena Rna Package, основанная на алгоритме Зукера. Она принимает на вход последовательность РНК
и расчитывает ее вторичную структуру с минимальной свободной энергией.
Командой cat 1gtr.fasta | RNAfold --MEA >> rna_fold.fasta был получен файл rna_fold.fasta,
где первая строчка - последовательность РНК, а последующие - предсказанная структура.
Полученные результаты трактовать заметно сложнее, чем в случае использования предыдущей программы.
Используются обозначения: точки - нуклеотиды, не образующие водородные связи, круглые скобки - спаренные нуклеотиды, квадратные и фигурные скобки - взаимодействия, образующие псевдоузлы.
Из также выданного файла 1GTR:B|PDBID|CHAIN|SEQUENCE_ss.ps
были получены номера спаренных нуклеотидов:
[1 69],
[2 68],
[3 67],
[5 65],
[6 64],
[9 23],
[10 22],
[11 21],
[25 41],
[26 40],
[27 39],
[28 38],
[29 37],
[47 63],
[48 62],
[49 61],
[50 60],
[51 59].
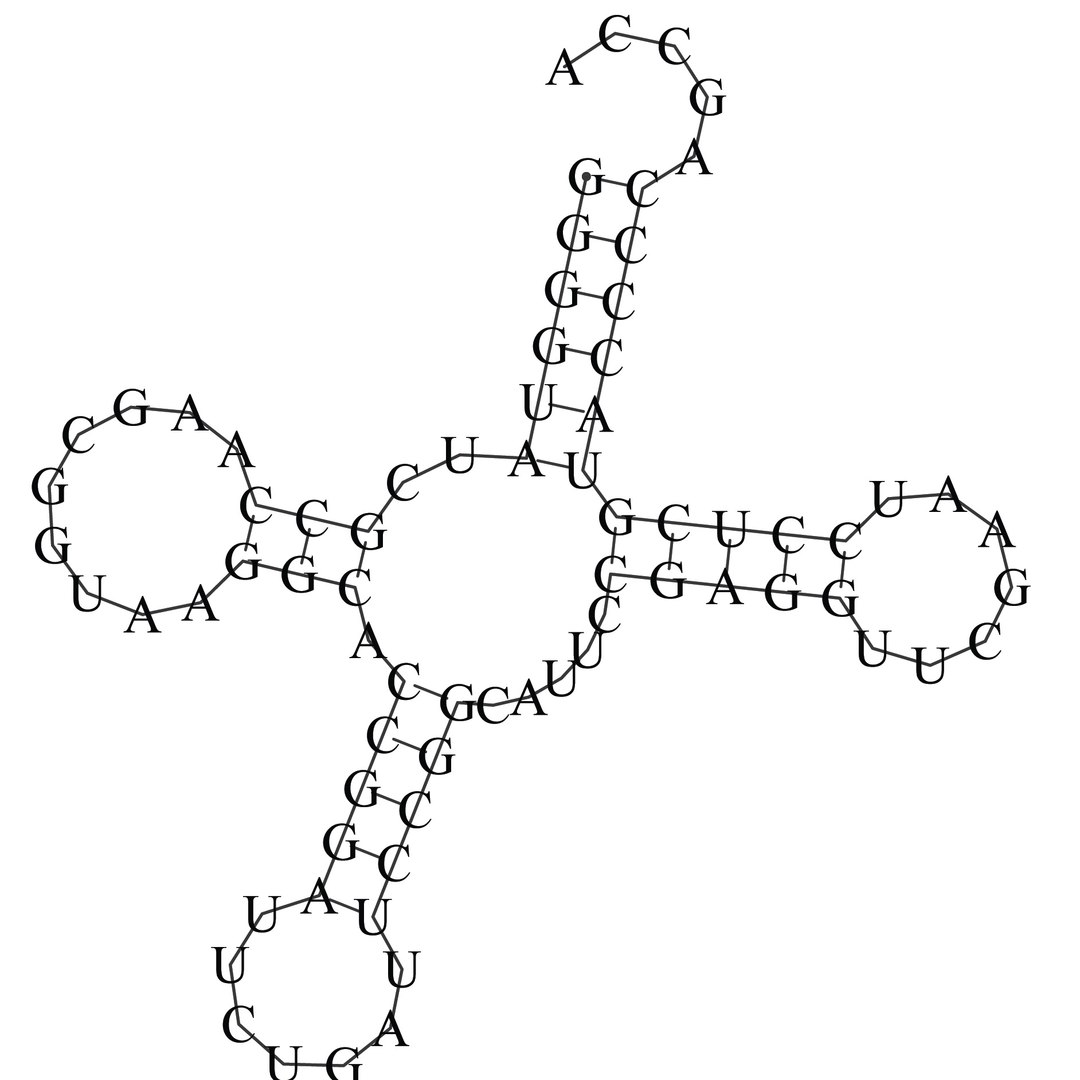
На рисунке 2 представлена предсказанная вторичная структура, полученная из этого же файла, на рисунке 3 - матрица для предсказанной вторичной структуры.
|
|
|
| Рис. 2 Предсказанная структуры тРНК с помощью RnaFold | Рис. 3 Матрица для предсказанной вторичной структуры |
В общем, программа RNAfold неплохо справляется с поставленной задачей. В таблице №2 представлено сравнение работы 3-х программ.
| Таблица №2 Сравнение реальной и предсказанной вторичной стуктуры тРНК |
|||
| Позиции в стуктуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера | |
| Акцепторный стебель | 2-7, 66-71 (всего 6) | 1-6, 64-69 (всего 6; абсолютно соответсвует, но почему-то сбита нумерация) | 1-6, 64-69 (всего 6) |
| Т-стебель | 49-53, 61-65 (всего 5) | 0 | 47-51, 59-63 (всего 5) |
| Антикодоновый стебель | 37-44, 26-33 (всего 8) | 0 | 25-29, 37-41 (всего 5) |
| D-стебель | 10-12, 23-25 (всего 3) | 0 | 9-11, 21-22 (всего 3) |
| Всего | 22 | 6 | 19 |
Из таблицы видно, что программа einverted плоха для определения вторичной структуры РНК, в то время как RNAfold обнаруживает все четыре стебля и почти верно определяет число взаимодействующих нуклеотидов для каждого из них.
Задание 2. Поиск ДНК-белковых контактов в заданной структуре
Упр. 1
Скрипт определяет множество атомов кислорода 2'-дезоксирибозы (set1),
множество атомов кислорода в остатке фосфорной кислоты (set2),
множество атомов азота в азотистых основаниях (set3) и дает последовательное изображение всей структуры,
только ДНК в проволочной модели, той же модели, но с выделенными шариками множеством атомов set1, затем set2 и set3.
Упр. 2
Далее требуется описать ДНК-белковые контакты в структуре с PDB ID 1rh6 и сравнить количество контактов разной природы.
Будем считать полярными атомы кислорода и азота, а неполярными атомы углерода, фосфора и серы.
Назовем полярным контактом ситуацию, в которой расстояние между полярным атомом белка и полярным атомом ДНК меньше 3.5 Å.
Аналогично, неполярным контактом будем считать пару неполярных атомов на расстоянии меньше 4.5 Å.
Результаты представлены в таблице №3.
| Таблица №3 Контакты разного типа в комплексе 1rh6.pdb |
|||
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 4 | 22 | 26 |
| остатками фосфорной кислоты | 11 | 17 | 28 |
| остатками азотистых оснований со стороны большой бороздки | 3 | 5 | 8 |
| остатками азотистых оснований со стороны малой бороздки | 1 | 0 | 1 |
Видно, что большинство контактов образуются с участием фосфорной кислоты. На втором месте - с остатками 2'-дезоксирибозы. Также можно отметить, что взаимодействий с остатками оснований большой бороздки контактов значительно больше, чем с остатками оснований со стороны малой. Это обуславливается большей пространственной доступностью атомов большой бороздки.
Упр.3
Здесь требуется получить популярную схему ДНК-белковых контактов с помощью программы nucplot.
Чтобы перевести файл в старый формат bdp, необходимый для работы програмыы, была использована команда remediator --old 1rh6.pdb >> 1rh6_old.pdb.
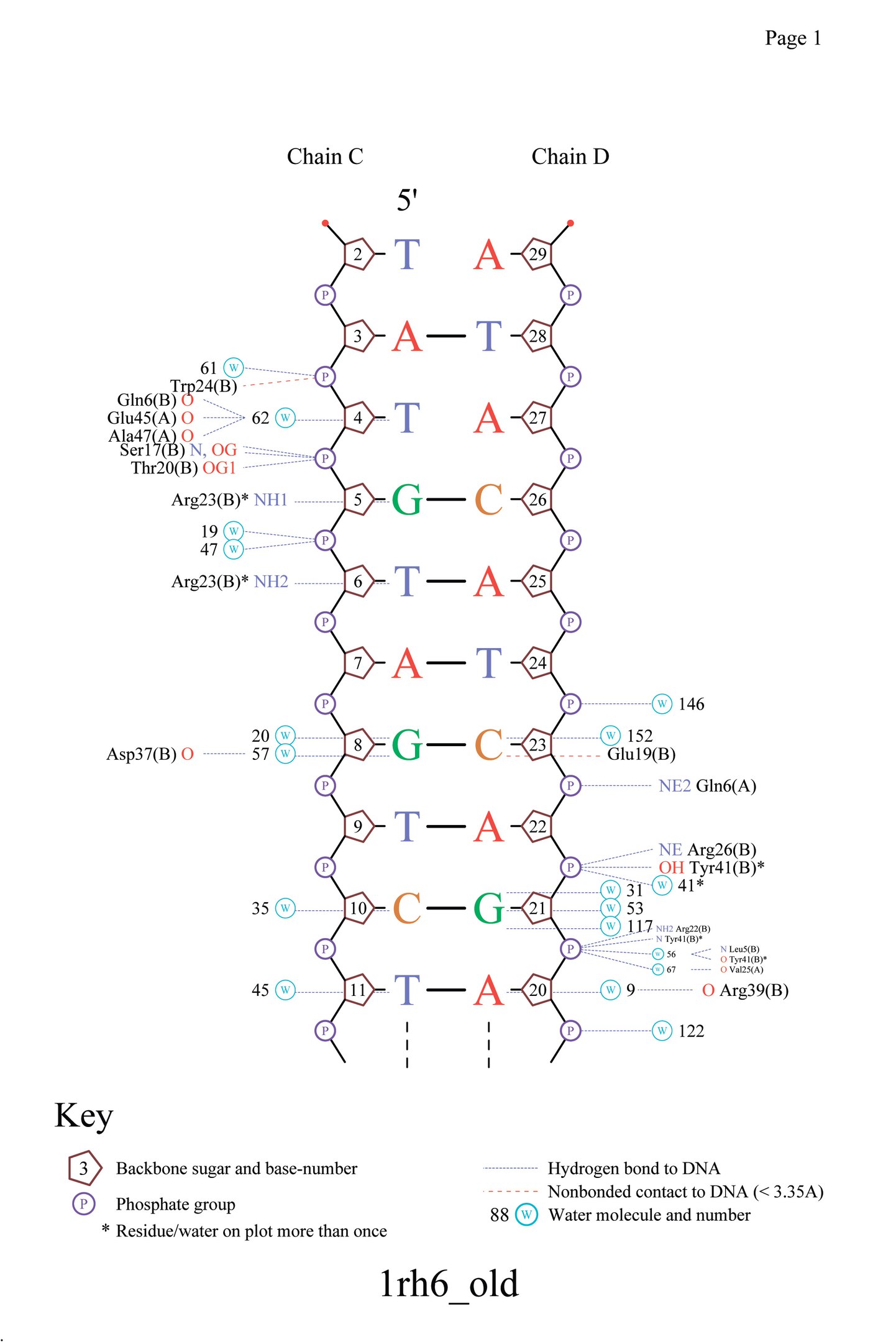
Затем, командой nucplot 1rh6_old.pdb был получен рис. 4 (из файла nucplot.ps).
Программа нашла не только определенные нами контакты, но и различные другие.
|
|
| Рис. 4 Популярная схема ДНК-белковых контактов |
Данный белок образует не очень много контактов с ДНК. Большинство контактов образует Arg23, а именно 2 контакта, с цепью C. Этот же остаток аминокислоты будет наиболее важным
для распознавания последовательности ДНК, поскольку именно у него самое большое количество связей с азотистыми основаниями.
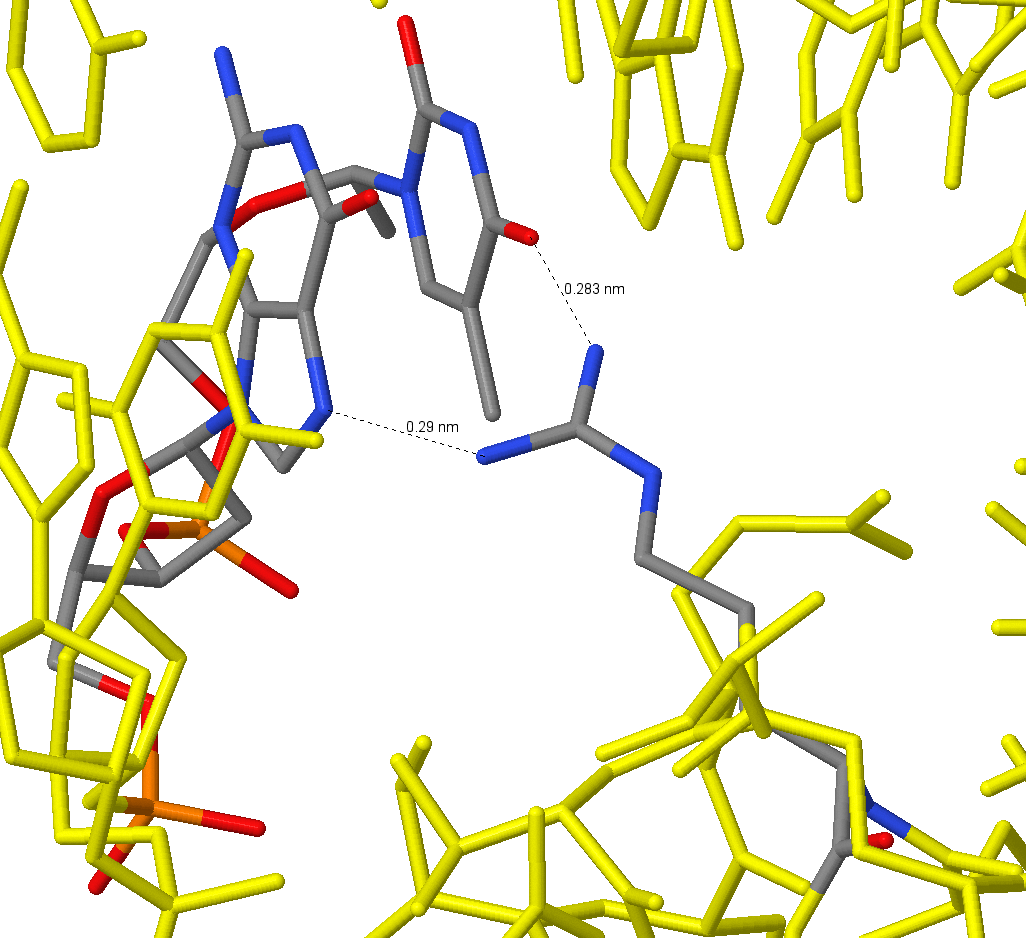
С помощью JMol было создано изображение взаимодействий Arg23 с ДНК (рис. 5).
|
|
| Рис. 5 Контакт Arg23 с G5 и T6 |