Прочтение последовательностей по Сэнгеру
Цель практикума - прочитать последовательность ДНК на основании данных, полученных из капиллярного секвенатора по Сэнгеру. Составить отчёт о проблемах при чтении хроматограмм.
Характеристика хроматограммы
1. Для прямой цепи начальный нечитаемый участок равен примерно 19(1-19) нуклеотидам, конечный - примерно 127(567-693) нуклеотидов.
Для цепи, обратной комплиментарной, конечный равен 20 (1-20, для не перевернутой), начальный - 45 (653-697, для не перевернутой).
Легко заметить, что качество сигнала максимально в средний части хроматограммы, а в начале и в конце качество сигнала плохое, не отличимое от шума.
2. Отношение сигнала и шума примерно 7.
3. Исходные файлы
прочтение прямой цепочки
прочтение обратной цепочки
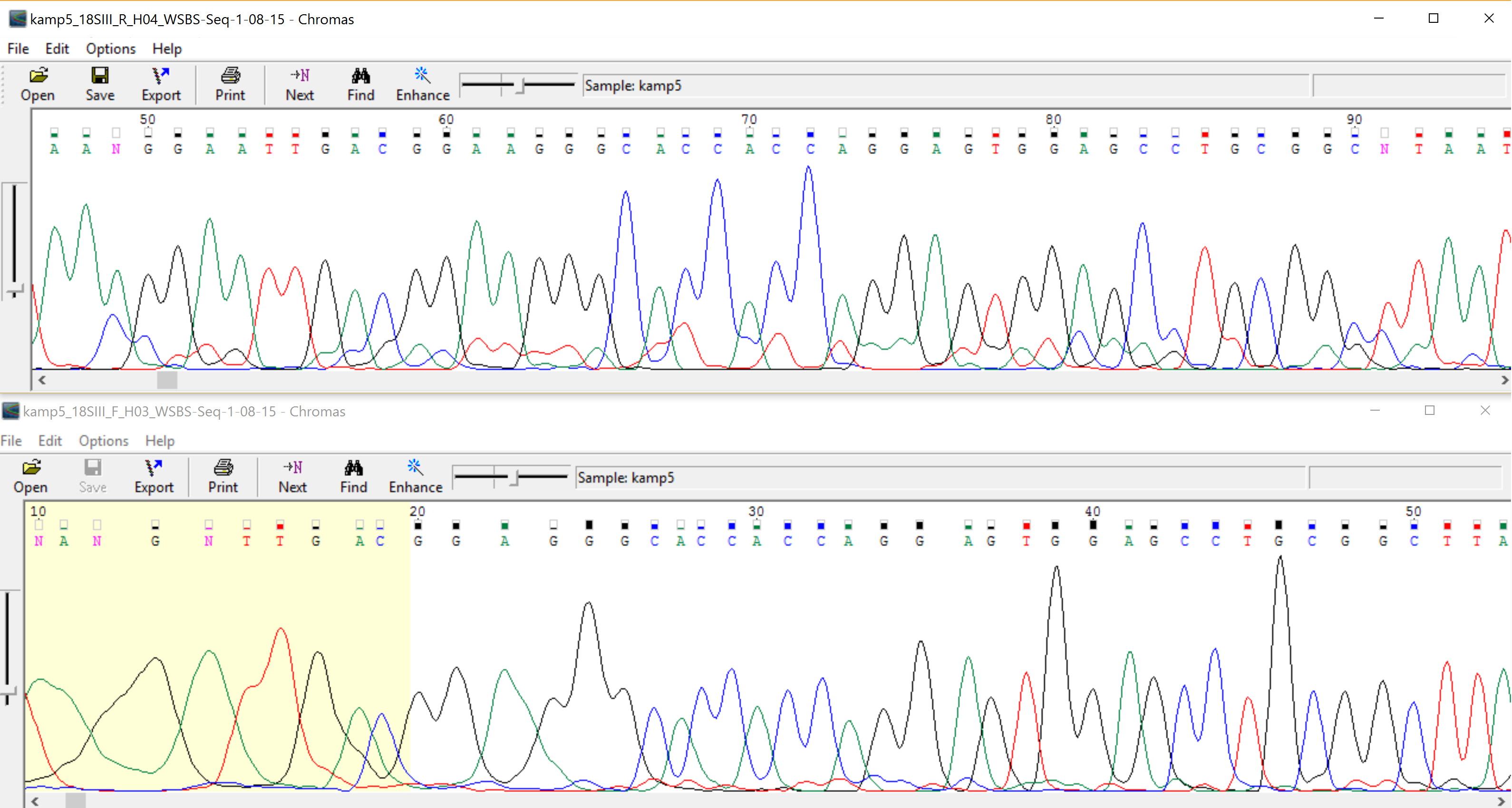
На рисунке 1 показаны выравненные друг под другой хроматограммы, с помощью поиска подслов (команда find).
|
|
| Рис. 1 Выравнивание прямой (внизу) и комплиментарной цепей (сверху) |
На рисунках 2-6 показаны типичные сложные места хроматограмм и методы их решения, так как программа не всегда способна выбрать правильное решение в спорном вопросе.
|
|
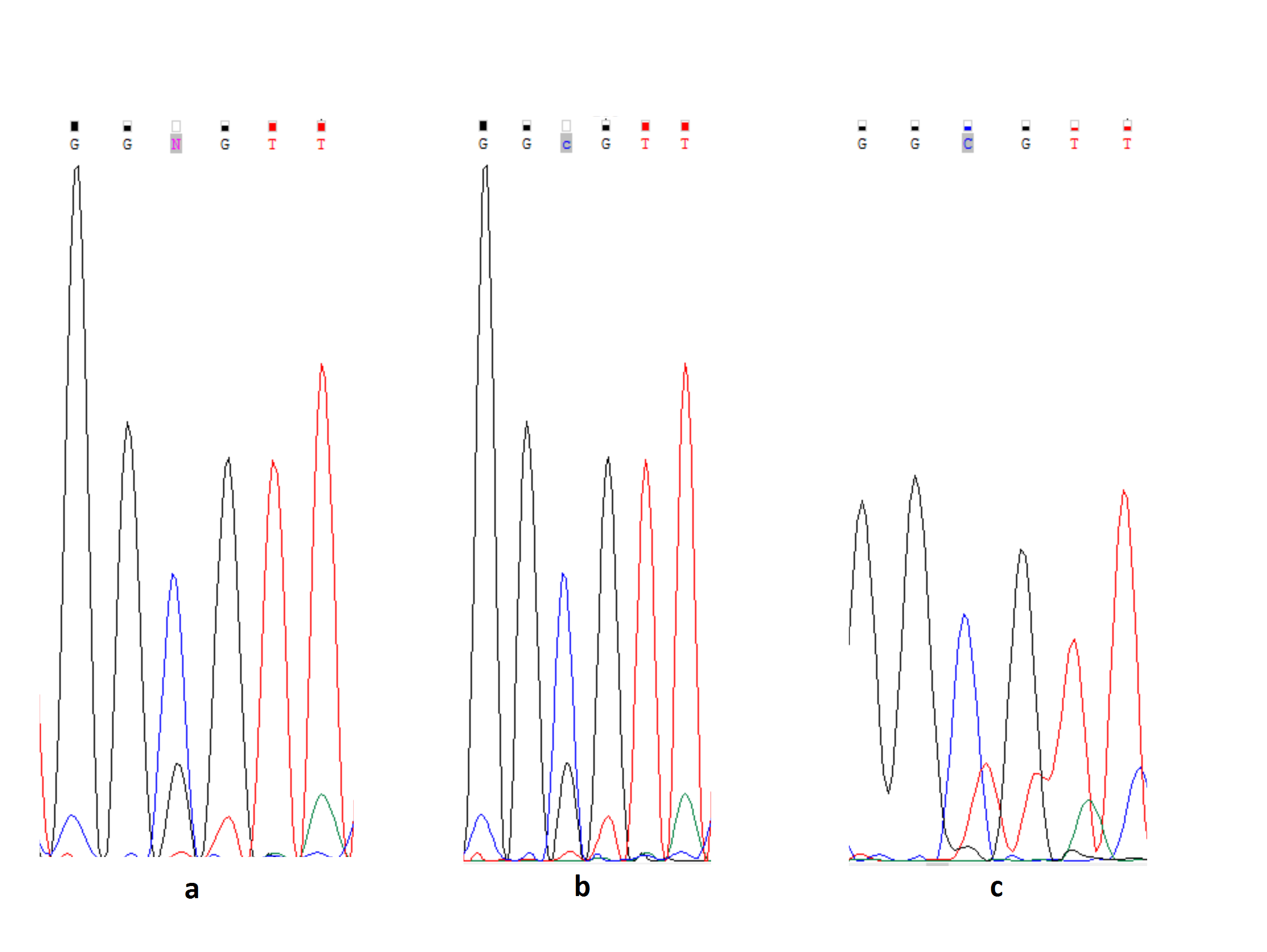
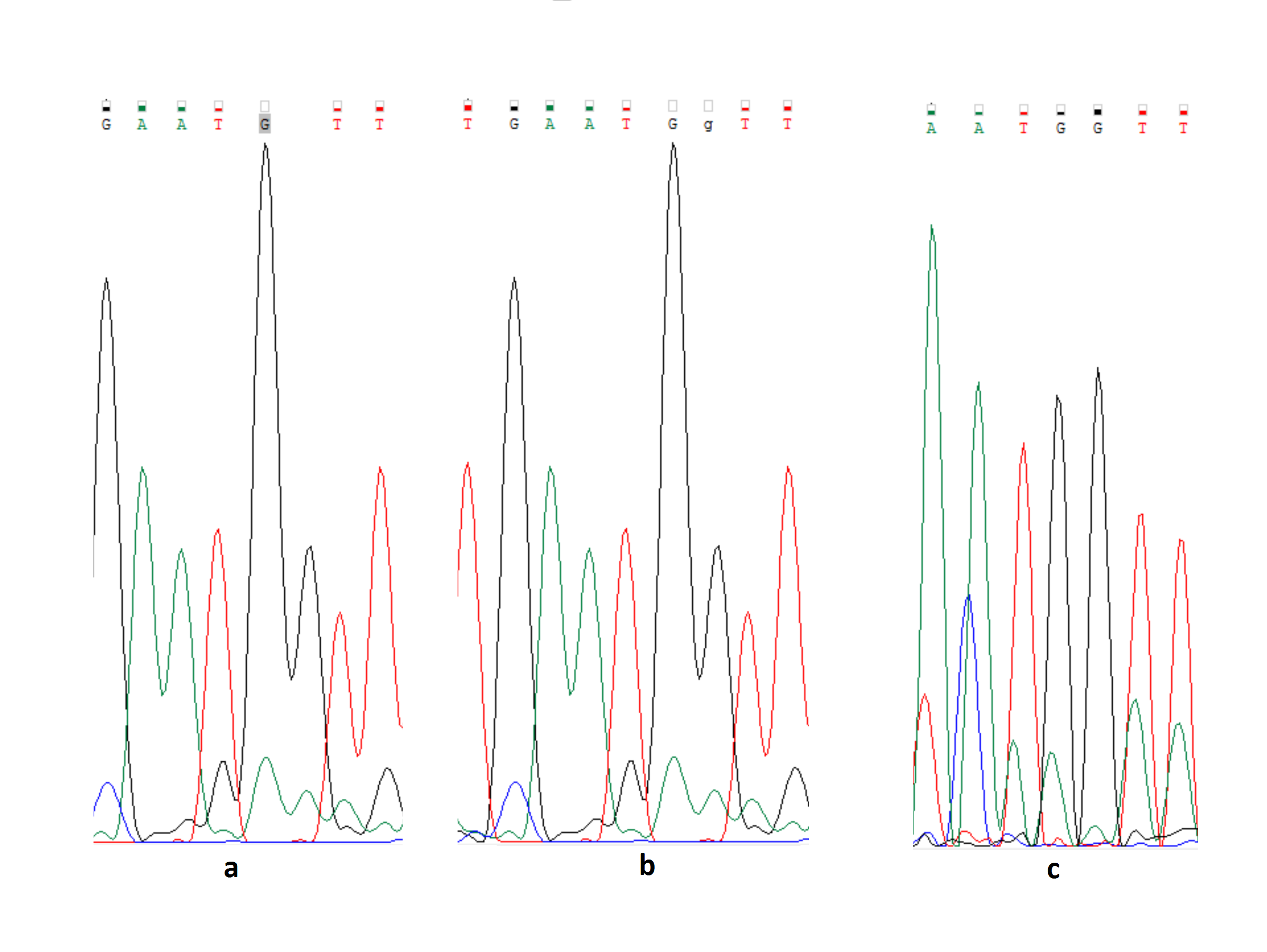
| Рис. 2 На месте 249-го нуклеотида прямой цепи программа поставила N (рис. 2а). Я же считаю, что поскольку шум меньше сигнала в 3 раза и соседные шумовые пики примерно такого же размера, то можно сказать, что на 249-м месте стоит C (рис. 2b). Также на перевернутой комплиментарной цепи сигнал однозначен - 307 C (рис. 2с) |
|
|
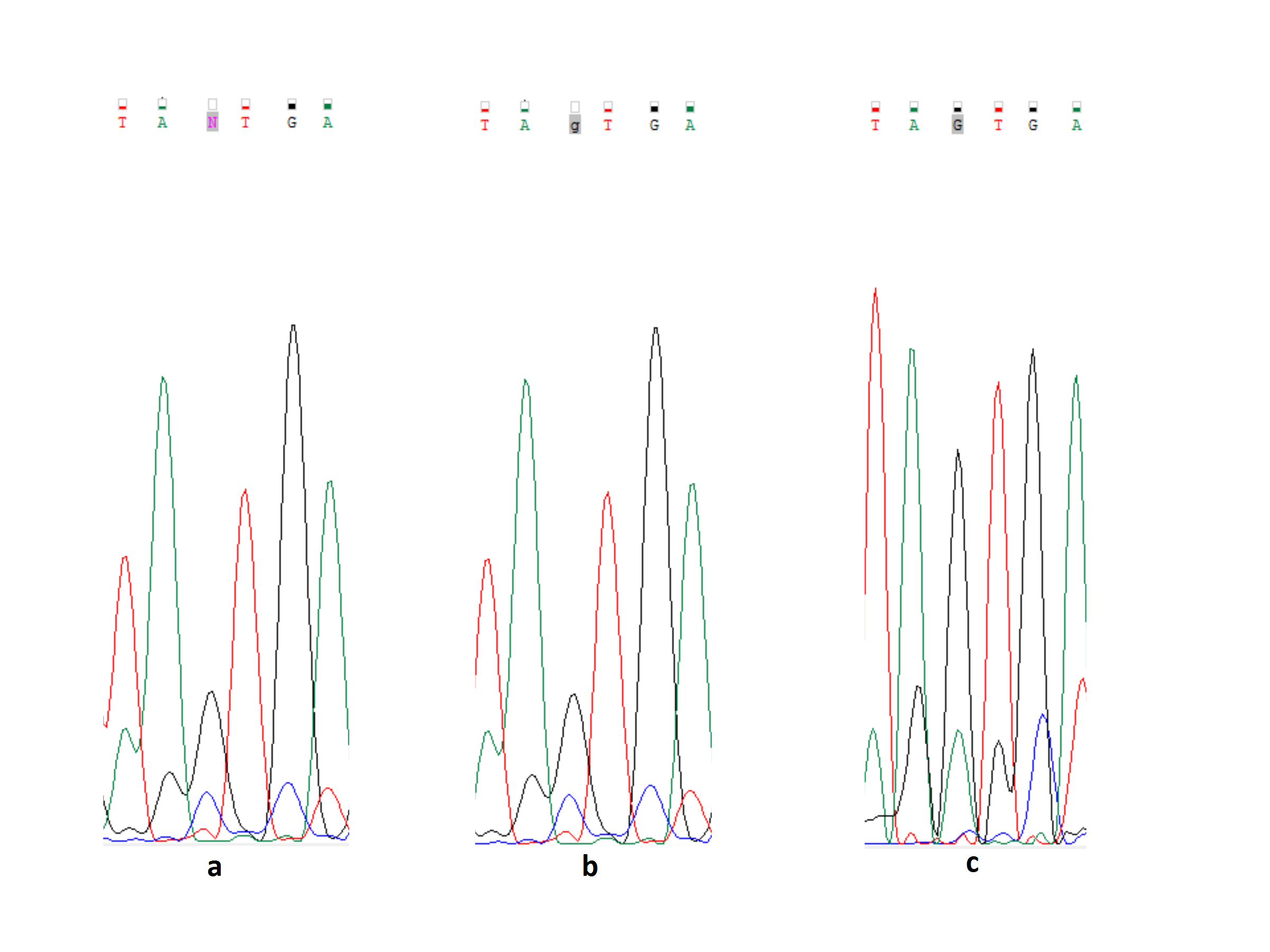
| Рис. 3 Я изменила N на месте 521-го нуклеотида прямой цепи (рис. 3а) на G (рис. 2b), так как шум достаточно маленький, сопостовим с соседними шумами и анализ комплиментарной цепи вносит ясность - 534 G(рис. 3с). |
|
|
| Рис. 4 Между 433-м C и 434-м С прямой цепи вклинивается шумовой пик аденина (подчеркнут зеленым), примерно равный сигналу 454-го G (подчеркнут черным). Однако, я согласна с анализом программы (между 433 и 434 именно шум), так как иначе расстояние между нуклеотдидами было бы слишком мало. |
|
|
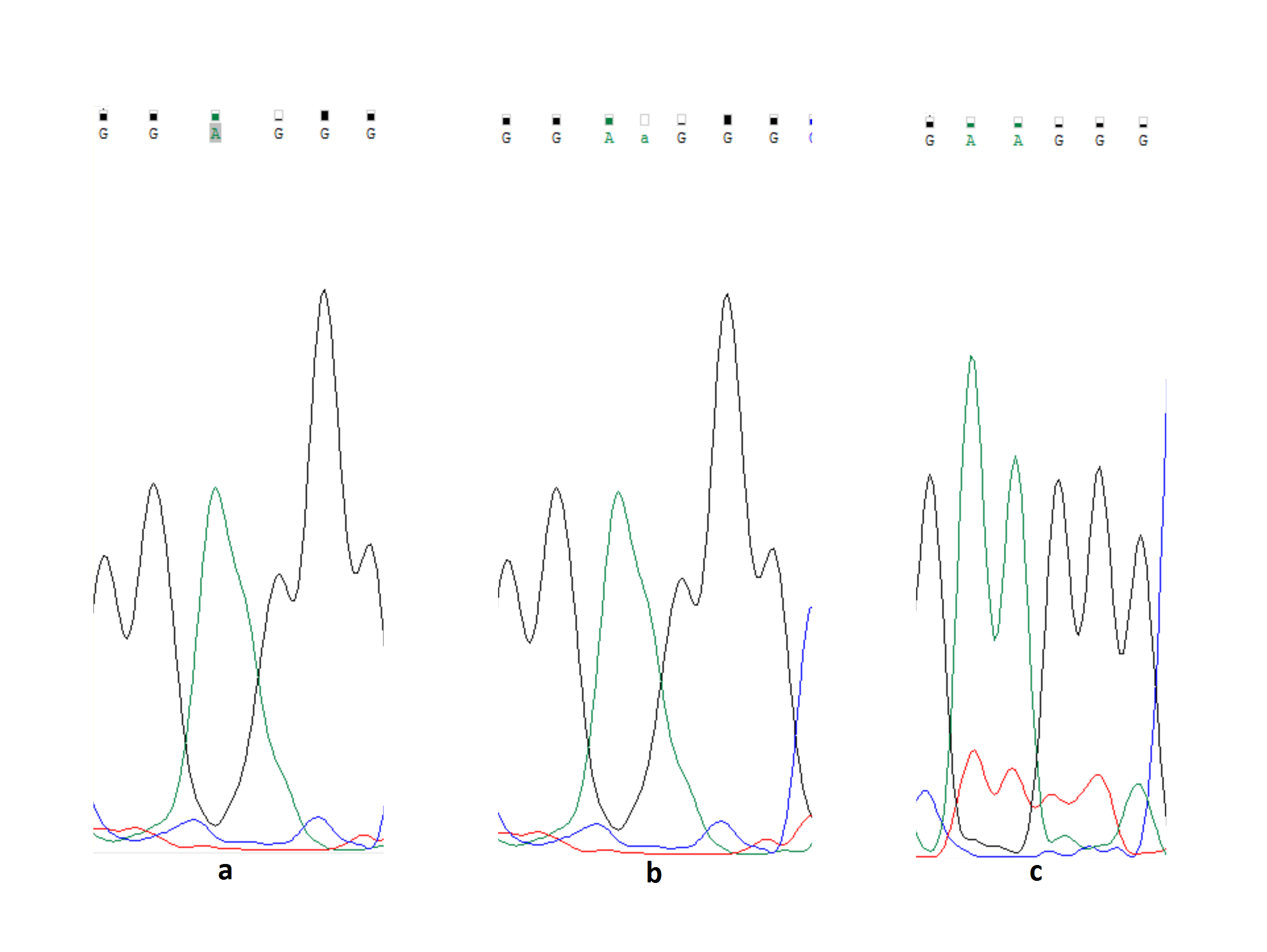
| Рис. 5 Широкий пик на месте 3 нуклеодида прямой цепи интерпретирован программой как один аденин (рис. 5a). Однако, при сравнении с комплиментарной цепью (рис. 5c) видно, что этот пик соответствует двум подряд идущим A (рис. 5b - исправленное). Ошибка допущена из-за неотчетливости начальных сигналов. |
|
|
| Рис. 6 Программа сочла пик после 515-го нуклеотида прямой цепи - шумовым (рис. 6а), но в таком случае расстояние между 516-м и 516-м слишком большое и этот пик даже больше сигнала 517-го тимина. По комплиментарной цепи видно (рис. 6c), что этот пик соответствуем гуанину. Исправлено на рис. 6b. |
При наложение прямой и перевернутой комплиментарной цепей получаем максимально длинную верную (более менее) проанализированную последовательность.
Полученные файлы:
выравнивание в формате fasta, выравнивание в формате jvp с окраской по нуклеотидам,
исправленная последовательность.
Исправленные нуклеотиды написаны строчными буквами.
Пример не читаемого фрагмента хроматограммы
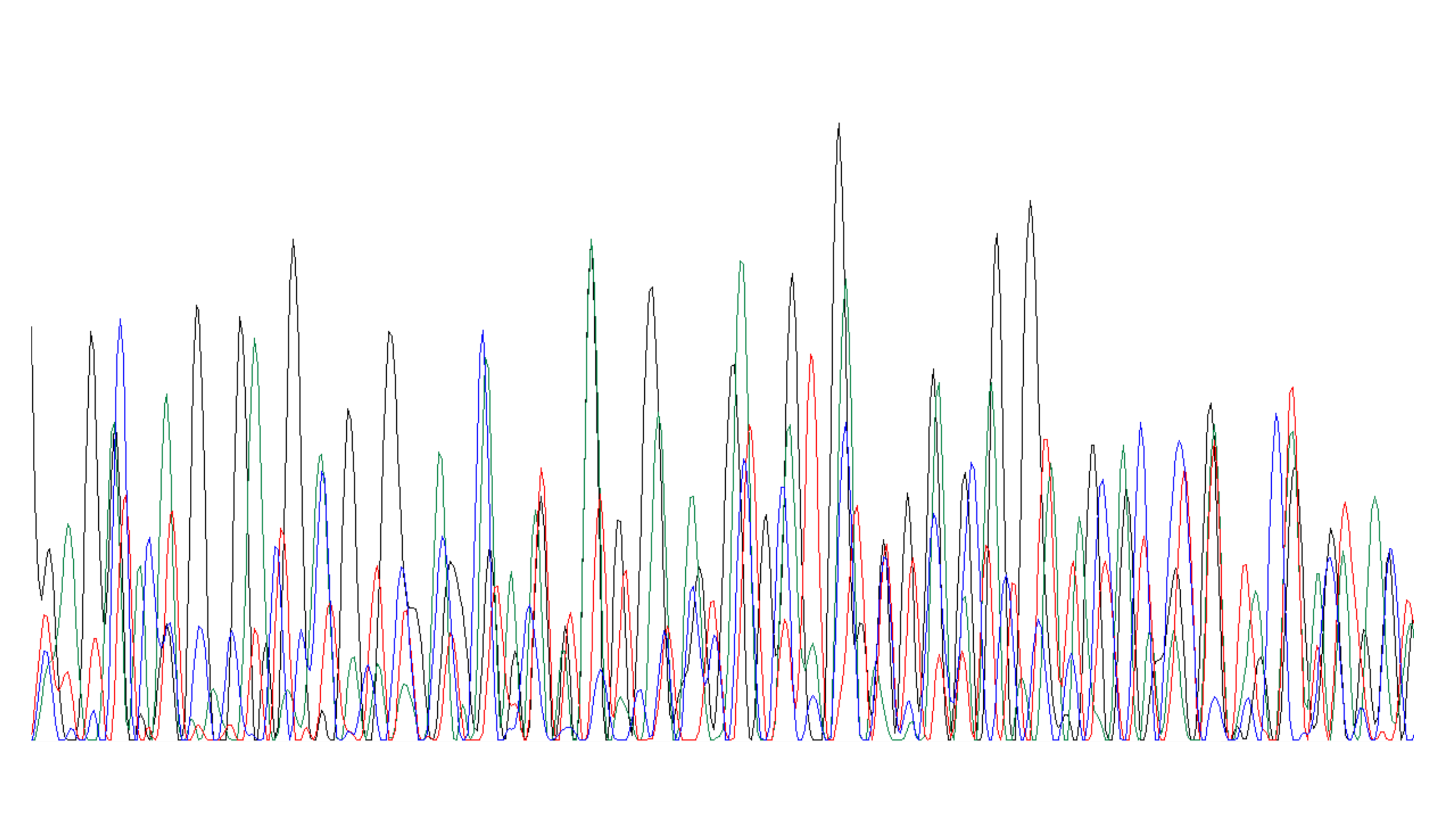
В качестве примера был взят файл NN_G10.ab1. Участок хроматограммы представлен на рисунке 7.
|
|
| Рис. 7 Видно, что хроматограмма абсолютно нечитаема. Такое бывает, если в секвенаторе читаются одновременно две разные последовательности: праймер для секвенирования отжегся на два разных участка или при ПЦР амплифицировались два фрагмента ДНК. Возможно произошло проскальнывание полимеразы, что привело к наложению пиков. |