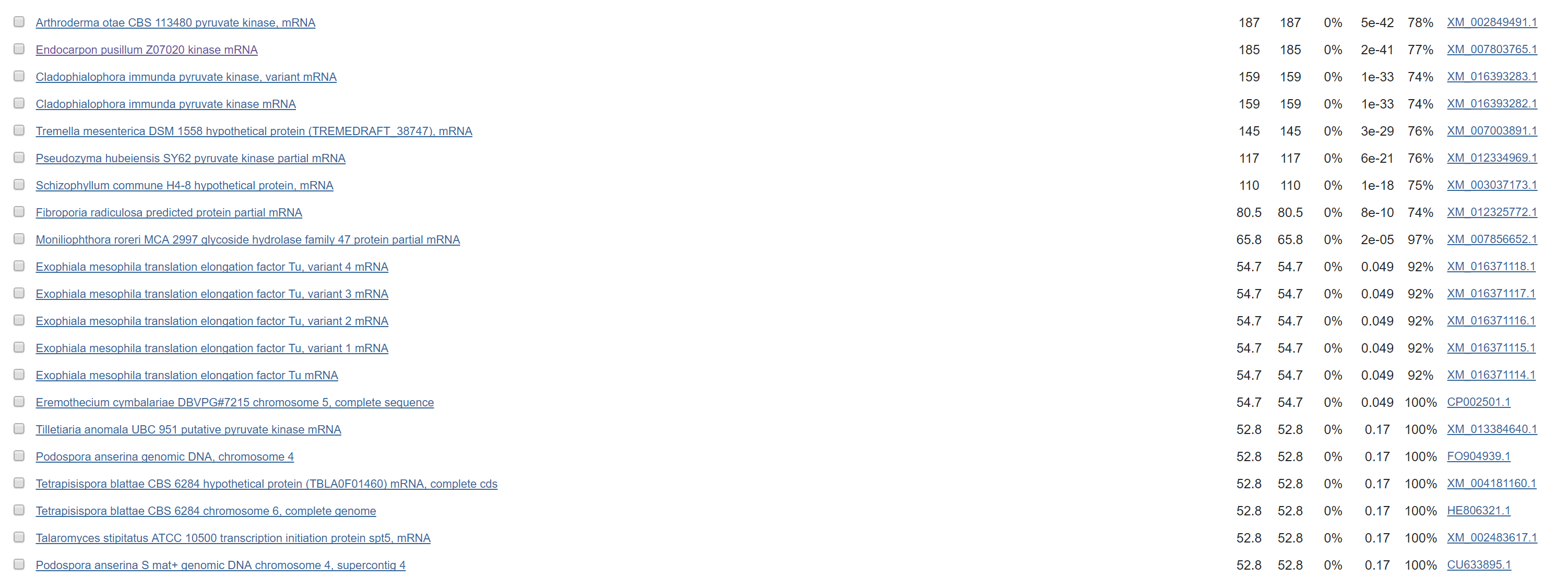Нуклеотидный blast
1. Определение таксономии и функции прочтенной нуклеотидной последовательности
Чтобы определить таксономию, я запустила нуклеотидный бласт (BLASTN) с параметрами по умолчанию и поиском по базе данных Nucleotide collection (nr/nt) для исправленной последовательности прямой цепи из 6-го практикума
(последовательность).
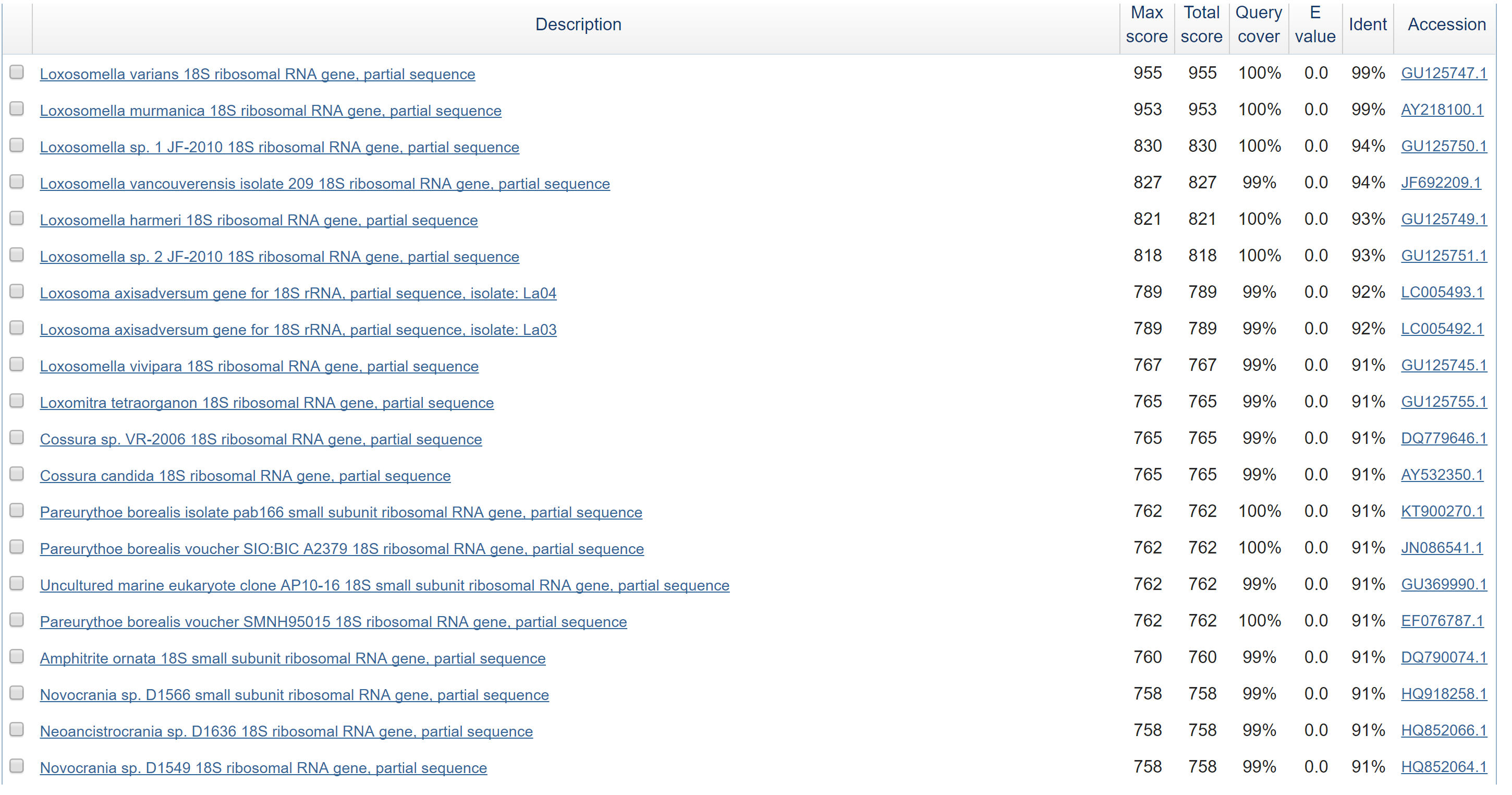
На рисунке 1 представлен топ-20 находок. Видно (в том числе по выравниванию), что самые большые совпадения с родами Loxosomella и Loxosoma из семейства Loxosomatidae.
Поэтому можно утверждать, что анализируемая последовательность принадлежит организму из того же семейства. Скорее всего, объект относится к роду Loxosomella, так как находки с максимальными параметрами
Bit-score, query cover и ident относятся именно к нему.
Также можно заключить, что данная последовательность принадлежит гену, кодирующему 18S рРНК. Выравнивания с максимально близкими последовательностями представлены на рисунке 2.
Ссылка на проект выравнивания в JalView.
|
|
| Рис. 1 Находки при поисками с параметрами по умолчанию |
|
Таблица №1 Таксономия |
|
| Домен | Eukaryota |
| Царство | Metazoa |
| Надтип | Lophotrochozoa |
| Тип | Entoprocta |
| Семейство | Loxosomatidae |
| Род | Loxosomella |
|
|
| Рис. 2 Выравнивание с максимально похожими последовательностями |
2. Сравнение списков находок 3-мя разными алгоритмами BLAST
Чтобы сузить поиск, я органичила его типом Entoprocta (Внутрипорошицевые).
Blastn ищет любые гомологи.
Длина слова по умолчанию: 11 нуклеотидов.
Всего найдена 31 сходная последовательность (рис. 2). Две худшие находки имеют покрытие всего 3% и e-value 0.71, что больше значения, позволяющего считать последовательности гомологичными.
Discontiguous megablast ищет близкие гомологи.
Длина слова по умолчанию: 11 нуклеотидов.
Найно 29 последовательностей (рис. 3). Даже у худшей находки e-value достаточно маленький, чтобы говорить о некой гомологии.
В общем, discontiguous megablast просто исключил из нахлдок blastn две последних, однозначно плохих находки.
Megablast ищет очень близкие гомологи.
Длина слова: 28
Матрица весов также отличается от предыдущих стратегий поиска. Match/Mismatch: 1/-2, штраф за гэп - линейный.
Находки те же самые, что и в discontiguous megablast, но с другими значениями bit-score, e-value, покрытия (рис. 4).
2. Описание ключей, используемых в таблицах особенностей
Ключи и аннатации к ним взяты с сайта INSDC.
Примеры - с сайта NCBI.
|
|
|
Рис. 2 Выдача blastn |
|
|
|
Рис. 3 Выдача discontiguous megablast |
|
|
|
Рис. 4 Выдача megablast |
3. Гомологи трех белков в геноме одного организма
Для поиска гомологов были выбраны 3 белка: CISY_HUMAN, RPB1_HUMAN, PABP2_HUMAN.
Задание было выполнено с помощью пакета BLAST+. Командой
makeblastdb -in X5.fasta -dbtype nucl
была создана локальная база данных.
Далее был запущен tblastn (ищет гомологов белка в транслированной нуклеотидной базе данных). Командой tblastn -db X5.fasta -query
были получены таблица №3, 4, 5.
А командой
tblastn -db X5.fasta -query
получены собственно выравнивания.
Первый белок CISY_HUMAN (ссылка в формате fasta, рис. 5) - цитратсинтетаза человека.
Этот фермент катализирует реакцию конденсации ацетата (ацетил-CoA) и оксалооцетата с образование цитрата.
Обнаружен почти во всех анаэробных организмах. Расположен в митохондриальном матриксе, но кодируется ядерным геномом и синтезируется рибосомами в цитоплазме, а затем переносится в митохондрии.
Всего tblastn нашел 6 предполагаемых гомологов (таблица №3, выравнивание), но рассматривать имеет смысл только два выравнивания (1 и 3) для scaffold-693 и scaffold-157.
В этих выравниваниях, как видно из таблицы, сымые большие значения identity и bit-score и самые маленькие evalue. И судя по выравниванию последовательности, действительно, сходны.
Такие данные позволяют говорить о наличии гомолога человеческой цитратсинтетазы у нашего организма (хотя возможно функция не сохранилась).
|
|
|
Рис. 5 Цитратсинтетаза |
| Таблица №3 Данные по выравниваниям |
|||||||||||
| query id | subject id | % identity | alignment length | mismatches | gap open | q. start | q. end | s. start | s. end | evalue | bit score |
| sp|075390|CISY_HUMAN | scaffold-693 | 69,5 | 377 | 122 | 3 | 90 | 464 | 1243882 | 1245009 | 2e-180 | 565 |
| sp|075390|CISY_HUMAN | scaffold-693 | 48,44 | 64 | 33 | 0 | 26 | 89 | 1243623 | 1234814 | 7e-14 | 74,3 |
| sp|075390|CISY_HUMAN | scaffold-157 | 69,5 | 377 | 112 | 3 | 90 | 464 | 314582 | 315709 | 5e-180 | 564 |
| sp|075390|CISY_HUMAN | scaffold-158 | 46,88 | 64 | 34 | 0 | 26 | 89 | 314323 | 314514 | 1e-13 | 73,2 |
| sp|075390|CISY_HUMAN | scaffold-287 | 25,76 | 396 | 234 | 15 | 83 | 442 | 458091 | 546976 | 3e-22 | 100 |
| sp|075390|CISY_HUMAN | scaffold-212 | 28,08 | 260 | 159 | 10 | 206 | 452 | 460007 | 45273 | 1e-19 | 92,4 |
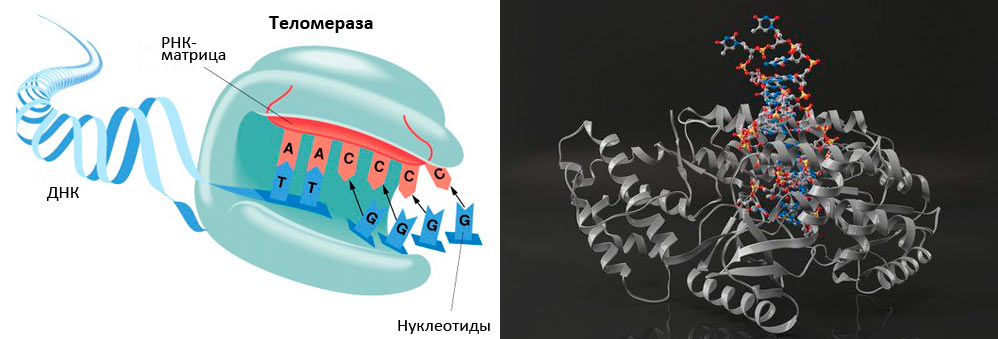
Второй белок TERT_HUMAN (ссылка в формате fasta) - человеческая теломера (рис. 6) - фермент, необходимый для репликации терминальной части хромосом.
Активен в стволовых и раковых клетках, малоактивен или неактивен совсем в соматических.
Две субъединицы теломеразы кодируются разными генами. Фермент состоит из теломеразной обратной транскриптазы, теломеразной РНК и дискерина.
Основная функция теломеразного комплекса - удлинение теломерных участков хромосом за счет функционирования, как обратная транкриптаза.
Фермент добавляет повторяющие короткое последовательности (5'-TTAGGG-3' у позвоночных, у других классов организмой последовательности другие) на 3'конец ДНК цепи, копируя свою РНК-компоненту.
Tblastn нашел 3 предполагаемых гомолога (таблица №4) и составил выравнивание. Последнее стоит совсем убрать из рассмотрения, судя по evalue.
Два остальных тоже не настолько хорошие, чтобы утверждать гомологию. Выравнивание случайное (blast еще и гэпов понаставил кучу) и в данном геноме нет гомолога человеческой теломеразы.
|
|
| Рис. 6 Теломераза |
| Таблица №4 Выравнивание с TERT_HUMAN |
|||||||||||
| query id | subject id | % identity | alignment length | mismatches | gap open | q. start | q. end | s. start | s. end | evalue | bit score |
| sp|O14746|TERT_HUMAN | scaffold-17 | 26,58 | 568 | 374 | 17 | 452 | 1007 | 610942 | 612552 | 8E-23 | 105 |
| sp|O14746|TERT_HUMAN | unplaced-307 | 24,87 | 579 | 372 | 17 | 452 | 1007 | 14902 | 16518 | 5E-18 | 90,1 |
| sp|O14746|TERT_HUMAN | scaffold-361 | 29,63 | 81 | 54 | 2 | 409 | 487 | 82346 | 82107 | 42614 | 32 |
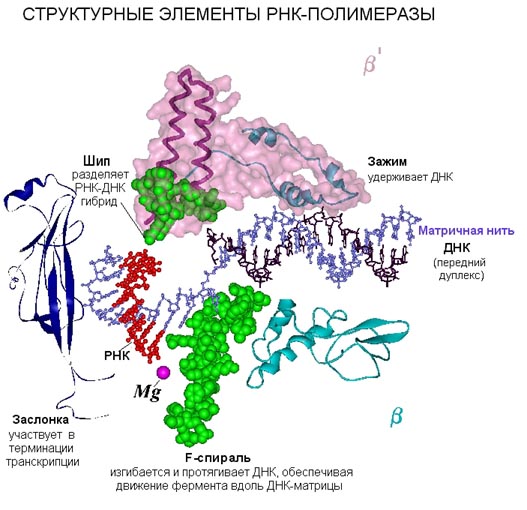
Третий белок RPB1_HUMAN (ссылка в формате fasta) - РНК-полимераза II.
Фермент эукариотических клеток, который катализирует транскрипцию ДНК в РНК, используя в качестве субстрата рибонуклеозидтрифосфаты.
Самый большой компонет РНК-полимеразы - наш белок (ДНК-зависимая субъединица RPB1 РНК-полимеразы II) -
содержит в C-концевом домене 52 повтора YSPTSPS, что необходимо для полимеразной активности.
RPB1 закрывает и открывает расселину в центре ядра полимеразы.
Найден 21 гомолог. Первая и третья находки (для scaffold-300 и scaffold-157) самые неплохие: evalue близко к нулю, а bit-score 1495 и 1390 соответственно.
Identity около 50% может говорить о гомологии только отдельных доменов белка. И судя по выравниванию, где некоторые фрагменты очень сходны,
а другие почти совсем не гомологичны, можно прийти к тому же выводу.
|
|
|
Рис. 6 РНК-полимераза |
| Таблица №5 Выравнивание с PABP2_HUMAN |
|||||||||||
| query id | subject id | % identity | alignment length | mismatches | gap open | q. start | q. end | s. start | s. end | evalue | bit score |
| sp|P24928|RPB1_HUMAN | scaffold-300 | 51,65 | 1547 | 683 | 18 | 10 | 1520 | 173104 | 177657 | 0 | 1495 |
| sp|P24928|RPB1_HUMAN | scaffold-300 | 36,41 | 868 | 510 | 14 | 16 | 854 | 142461 | 139897 | 3E-146 | 509 |
| sp|P24928|RPB1_HUMAN | scaffold-300 | 29,48 | 424 | 230 | 10 | 1058 | 1474 | 138270 | 137185 | 2E-39 | 161 |
| sp|P24928|RPB1_HUMAN | scaffold-300 | 49,06 | 53 | 27 | 0 | 854 | 906 | 138639 | 138481 | 0,00000005 | 58,2 |
| sp|P24928|RPB1_HUMAN | scaffold-157 | 53,04 | 1397 | 616 | 16 | 137 | 1520 | 534515 | 530406 | 0 | 1390 |
| sp|P24928|RPB1_HUMAN | scaffold-157 | 36,75 | 868 | 507 | 14 | 16 | 854 | 562218 | 564782 | 2E-151 | 526 |
| sp|P24928|RPB1_HUMAN | scaffold-157 | 30,14 | 418 | 235 | 10 | 1058 | 1474 | 566409 | 567494 | 1E-38 | 159 |
| sp|P24928|RPB1_HUMAN | scaffold-157 | 52,25 | 111 | 51 | 1 | 11 | 121 | 534958 | 534632 | 1E-30 | 133 |
| sp|P24928|RPB1_HUMAN | scaffold-157 | 51,02 | 49 | 24 | 0 | 854 | 902 | 566040 | 566186 | 0,00000008 | 57,4 |
| sp|P24928|RPB1_HUMAN | scaffold-157 | 33,85 | 65 | 41 | 1 | 1011 | 1073 | 326645 | 326451 | 0,036 | 38,9 |
| sp|P24928|RPB1_HUMAN | scaffold-44 | 34,28 | 388 | 211 | 8 | 343 | 694 | 232854 | 233993 | 2E-50 | 197 |
| sp|P24928|RPB1_HUMAN | scaffold-44 | 45,28 | 159 | 85 | 2 | 764 | 920 | 234665 | 235141 | 2E-33 | 142 |
| sp|P24928|RPB1_HUMAN | scaffold-44 | 37,24 | 145 | 83 | 5 | 1056 | 1197 | 235352 | 235771 | 4E-17 | 88,2 |
| sp|P24928|RPB1_HUMAN | scaffold-44 | 31,18 | 170 | 109 | 4 | 1310 | 1478 | 236450 | 236938 | 8E-16 | 84 |
| sp|P24928|RPB1_HUMAN | scaffold-44 | 32,69 | 104 | 65 | 3 | 14 | 114 | 231513 | 231818 | 0,000000004 | 61,6 |
| sp|P24928|RPB1_HUMAN | scaffold-100 | 34,28 | 388 | 211 | 8 | 343 | 694 | 317738 | 318877 | 3E-49 | 194 |
| sp|P24928|RPB1_HUMAN | scaffold-100 | 45,28 | 159 | 85 | 2 | 764 | 920 | 319548 | 320024 | 3E-33 | 141 |
| sp|P24928|RPB1_HUMAN | scaffold-100 | 37,86 | 140 | 78 | 4 | 1056 | 1192 | 320235 | 320636 | 1E-16 | 86,7 |
| sp|P24928|RPB1_HUMAN | scaffold-100 | 31,18 | 170 | 109 | 4 | 1310 | 1478 | 321333 | 321821 | 4E-16 | 85,1 |
| sp|P24928|RPB1_HUMAN | scaffold-100 | 32,69 | 104 | 65 | 3 | 14 | 114 | 316391 | 316696 | 0,00000006 | 57,8 |
| sp|P24928|RPB1_HUMAN | unplaced-712 | 52,25 | 111 | 51 | 1 | 11 | 121 | 1 | 327 | 6E-35 | 132 |
4. Поиск гена белка, закодированного в одном скэффолде ''Amoboaphelidium''
Amoeboaphelidium protococarum - протист, родственный грибам, относится к Aphelida. Последовательность его генома находтся в файле X5.fasta.
Сначала была получена информация о длинах скэффолдов в файле X5.fasta с помощью команды
infoseq X5.fasta -only -name -length >> scaffolds.out
Для анализа я выбрала скэффолд 115 длины 160031. С помощью команды
seqret X5.fasta:scaffold-115 -out scaffold115.fasta
получила в файле scaffold115.fasta его последовательность.
С помощью megablast с ограничение поиска только по Fungi была найдена 21 сходная последовательность (рис. 10), причем большинство находок оказались генами, кодирующими
пируват киназу - фермент, катализирующий финальную стадию гликолиза (рис. 9). Исходя из этого, а также вполне приемлимых значений cover, identity и e-value можно заключить, что именно этот ген закодирован в скэффолде 115.
|
|
|
Рис. 10 Результат поиска |