Филогенетическое дерево
В таблице №1 представлены выбранные мною организмы.
| Таблица №1 Отобранные бактерии |
|
| Название | Мнемоника |
| Bacillus anthracis | BACAN |
| Clostridium botulinum | CLOBA |
| Clostridium tetani | CLOTE |
| Geobacillus kaustophilus | GEOKA |
| Lactobacillus acidophilus | LACAC |
| Staphylococcus aureus | STAAR |
| Streptococcus pyogenes serotype M1 | STRP1 |
Филогенетическое дерево в скобочной форме: ((CLOTE,CLOBA),((LACAC,STRP1),((BACAN,GEOKA),STAAR)))
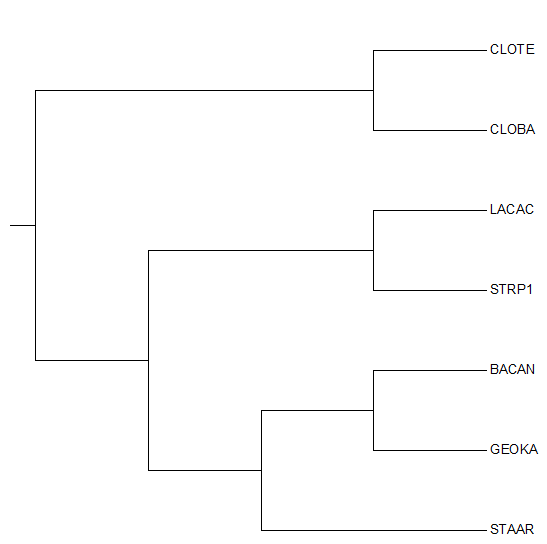
Изображение дерева представлено на рисунке 1.
|
|
| Рис. 1 Изображение дерева |
Нетривиальные ветви:
1) {CLOTE, CLOBA} vs {LACAC, STRP1, BACAN, GEOKA, STAAR};
2) {CLOTE, CLOBA, LACAC, STRP1, STAAR} vs {BACAN, GEOKA};
3) {CLOTE, CLOBA, LACAC, STRP1} vs {BACAN, GEOKA, STAAR};
4) {CLOT, CLOBA, BACAN, GEOKA, STAAR} vs {LACAC, STRP1}.
Реконструкция филогении
Пользуясь таксономическим сервисом NCBI (http://www.ncbi.nlm.nih.gov/taxonomy/) я определила, к каким таксонам относятся отобранные бактерии (таблица №2).
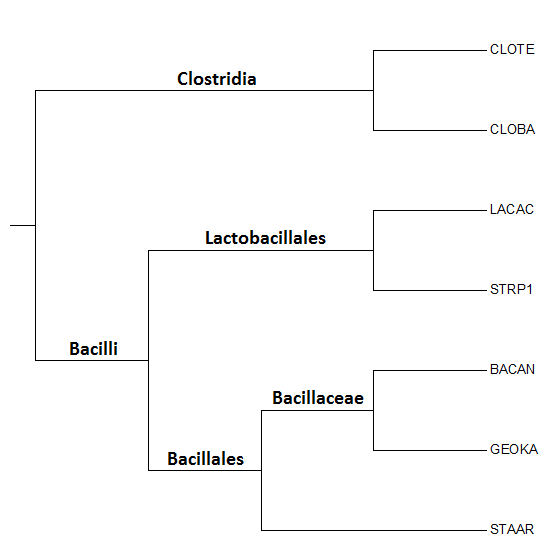
Из рисунка 2 видно, что все нетривиальные ветви разделяют различные таксоны: класс Clostridia с классом Bacilli,
отряд Lactobacillales с Bacillales (и др.), семейство Bacillaceae с прочими семествами.
| Таблица №2 Таксономия выбраных бактерий | ||
| Название | Мнемоника | Таксономия |
| Bacillus anthracis | BACAN | Bacteria; Terrabacteria group; Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus cereus group; Bacillus anthracis |
| Clostridium botulinum | CLOBA | Bacteria; Terrabacteria group; Firmicutes; Clostridia; Clostridiales; Clostridiaceae; Clostridium |
| Clostridium tetani | CLOTE | Bacteria; Terrabacteria group; Firmicutes; Clostridia; Clostridiales; Clostridiaceae; Clostridium |
| Geobacillus kaustophilus | GEOKA | Bacteria; Terrabacteria group; Firmicutes; Bacilli; Bacillales; Bacillaceae; Geobacillus; Geobacillus thermoleovorans group |
| Lactobacillus acidophilus | LACAC | Bacteria; Terrabacteria group; Firmicutes; Bacilli; Lactobacillales; Lactobacillaceae; Lactobacillus |
| Staphylococcus aureus | STAAR | Bacteria; Terrabacteria group; Firmicutes; Bacilli; Bacillales; Staphylococcaceae; Staphylococcus |
| Streptococcus pyogenes serotype M1 | STRP1 | Bacteria; Terrabacteria group; Firmicutes; Bacilli; Lactobacillales; Streptococcaceae; Streptococcus; Streptococcus pyogenes |
|
|
| Рис. 2 Изображение дерева с указанием таксонов |
Далее требовалось реконструировать филогенетическое дерево по заданному семейству белков. В качестве белка я выбрала рибосомный белок S4 (RS4).
File -> Fetch sequences, Select Database -> Uniprot.
Далее в строку вписала выражение вида (мнемоника функции)_(мнемоника организма) для каждой выбранной бактерии через ';' (RS4_BACAN ; RS4_CLOBA ; RS4_CLOTE ; RS4_GEOKA ; RS4_LACAC ; RS4_STAAR ;RS4_STRP1).
Полученные оследовательности выравнены при помощи Muscle (Сalculate -> Calculate Tree -> Neighbour Joining Using % Identity).
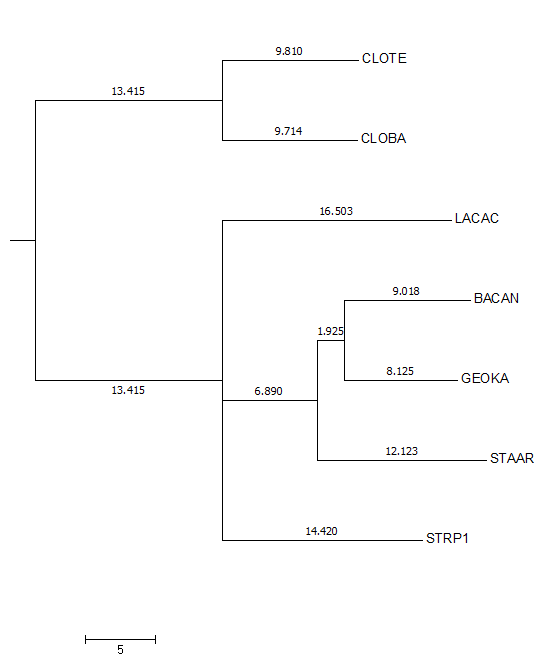
По выравниванию было построено дерево методом "Neighbor Joining Using % Identity" (рис. 3).
Оно было сохранено в Newick-формате и открыто в программе MEGA. Выравнивание в fasta-формате: pr2_alignment.fasta, проект Jalwiew: pr2.jvp, дерево в Newick-формате: pr2_tree.tre.
|
|
| Рис. 3 Изображение дерева, полученное после выравниания |
На рисунке 3 представлено полученное с помощью Jalview изображение построенного дерева. Важно заметить, что дерево получилось небинарным.
Возможно из-за того, что расстояние получилось слишком маленьким м Jalwiew
округлил его до нуля.
Поэтому в нетривиальных ветвях ветвях привожу возможное разрешения:
1) {CLOTE, CLOBA} VS {LACAC, STRP1, STAAR, GEOKA, BACAN};
2) {BACAN, GEOKA} VS {STAAR, STRP1, LACAC, CLOBA, CLOTE};
3) {BACAN, GEOKA, STAAR} VS {STRP1, LACAC, CLOBA, CLOTE}
4.1) {BACAN, GEOKA, STAAR, LACAC} VS {STRP1, CLOBA, CLOTE};
4.2) {BACAN, GEOKA, STAAR, STRP1} VS {LACAC, CLOBA, CLOTE};
4.3) {LACAC, STRP1} VS {CLOT, CLOBA, BACAN, GEOKA, STAAR}.
Видно, что три первых нетривиальных ветви совпали, то есть топология деревьев совпала (ну почти). Правильное разрешение соответсвует нетривиальной ветви (4.3).