Построение профиля подсемейства
1. Выбoр подсемейства
Для выполнения практикума мне был дан файл с ридами chipseq_chunk18.fastq.
Контроль качества был произведен командой fastq:
fastqc chipseq_chunk18.fastq
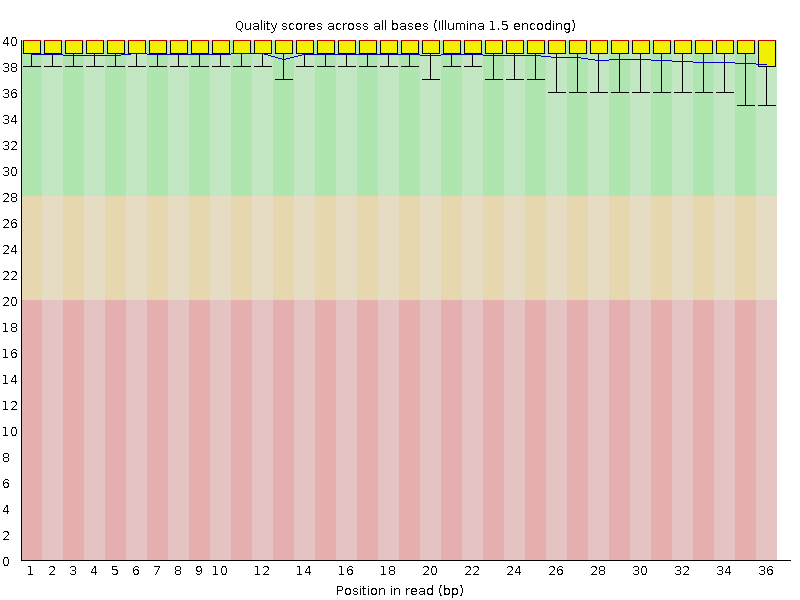
Так был получен HTML-файл, содержащий информацию о ридах. Всего 6546 ридов; по рисунку 1 можно оценить их качетсво.
Видно, что качество ридов очень хорошее, дополнительная чистка не требуется.
|
|
| Рис. 1 Per base sequence quality |
2. Картирование ридов на геном человека
Картирование выполнялось с помощью команды BWA для проиндексированного генома:
bwa mem /srv/databases/ngs/hg19/GRCh37.p13.genome.fa chipseq_chunk18_out.fastq >
chipseq_chunk18.sam
Командой:
samtools view chipseq_chunk18.sam -bSo chipseq_chunk18.bam
полученный файл был переведет в бинарный формат .bam.
Выравнивание ридов с референсом было отсортировано по координате в референсе начала рида:
samtools sort chipseq_chunk18.bam -T chip_temp -o chipseq_chunk18_sorted.bam
Полученный файл был проиндексирован:
samtools index chipseq_chunk18_sorted.bam
Было выяснено, сколько чтений откартировалось на геном:
samtools idxstats chipseq_chunk18_sorted.bam > chipseq.out
Из файла chipseq.out выяснилось,
6038 из 6546 были откартированы на хромосому 5, таким образом скорее всего прочтения с нее мне были предложены для анализа.
Всего было откартировано 6519 ридов.
3. Поиск пиков
Peak calling — компьютерный метод поиска областей генома, на которые откартировалось большое число ридов из данных эксперимента ChiP-Seq.
Для поиска пиков использовалась программа MACS (Model-based Analysis of Chip-Seq). Была использована команда (параметр --nomodel необходим, поскольку пиков слишком мало):
macs2 callpeak -n chipseq_chunk41 -t chipseq_chunk18_sorted.bam --nomodel
В результате были получены narrowPeak- xls- и bed-файлы, содержащие информацию о пиках (таблица 1).
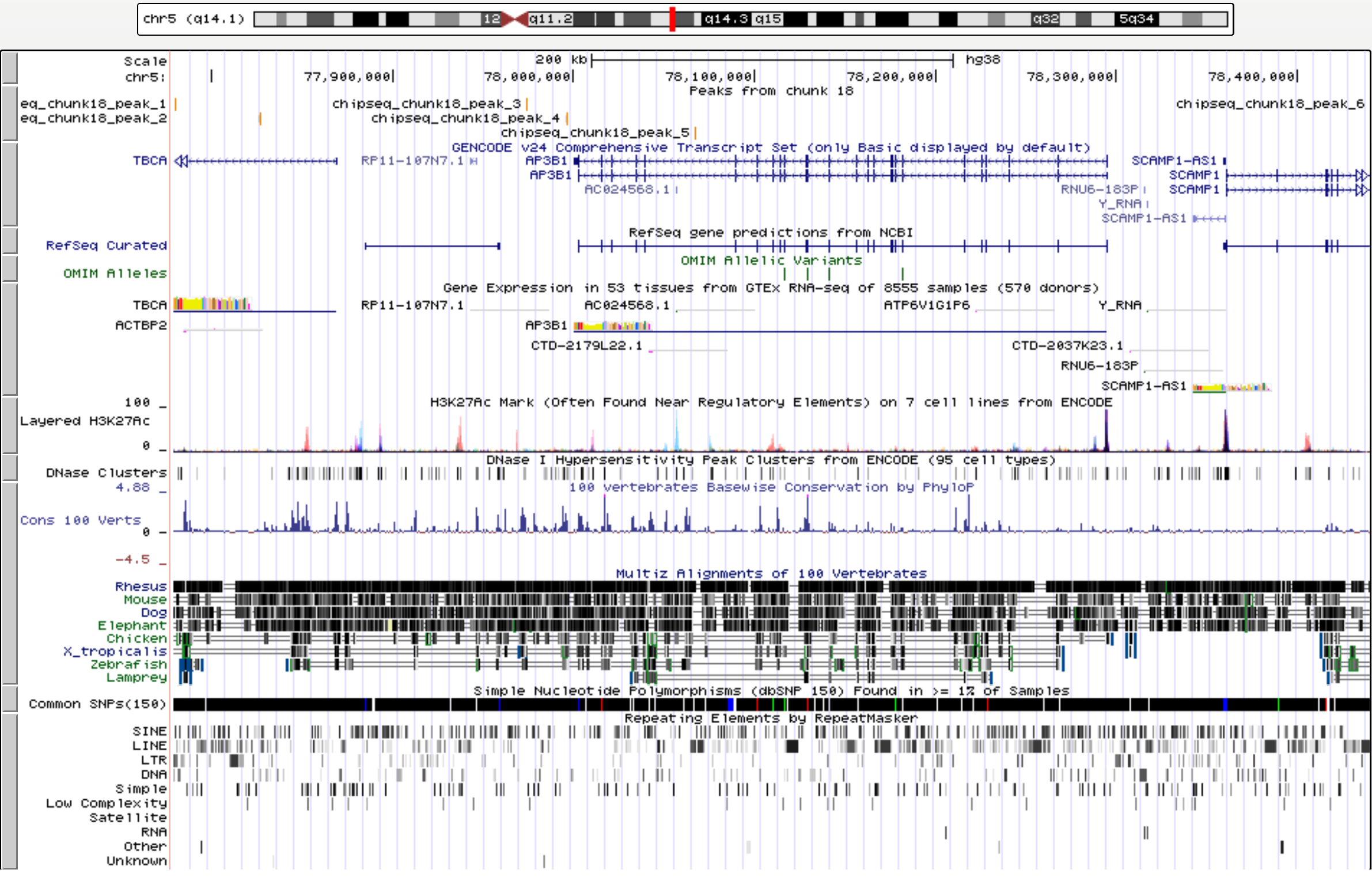
Всего найдено 6 пиков на 5-й хромосоме. С помощью геномного браузера (UCSC Genome Browser) они были визуализированны (рисунок 2).
| Таблица №1 Информация о найденных пиках |
|||||
| № | Начало | Конец | Длина | Расстояние от старта пика до вершины | -lg(p-value) |
| 1 | 77779687 | 77779970 | 284 | 92 | 20.42412 |
| 2 | 77826235 | 77826517 | 283 | 144 | 30.77239 |
| 3 | 77974151 | 77974405 | 255 | 128 | 15.04807 |
| 4 | 77995753 | 77995991 | 239 | 122 | 21.02363 |
| 5 | 78067225 | 78067471 | 247 | 108 | 18.16418 |
| 6 | 78439925 | 78440124 | 200 | 100 | 18.26694 |
|
|
| Рис. 2 Визуализация пиков в UCSC Genome Browser |
О достоверноси пиков можно судить по значениям p-value. В файле *_peaks.narrowPeak для каждого пика посчитаны значения -lg(p-value).
Чем -lg(p-value) больше, тем меньше p-value, а следовательно, достовернее находка.
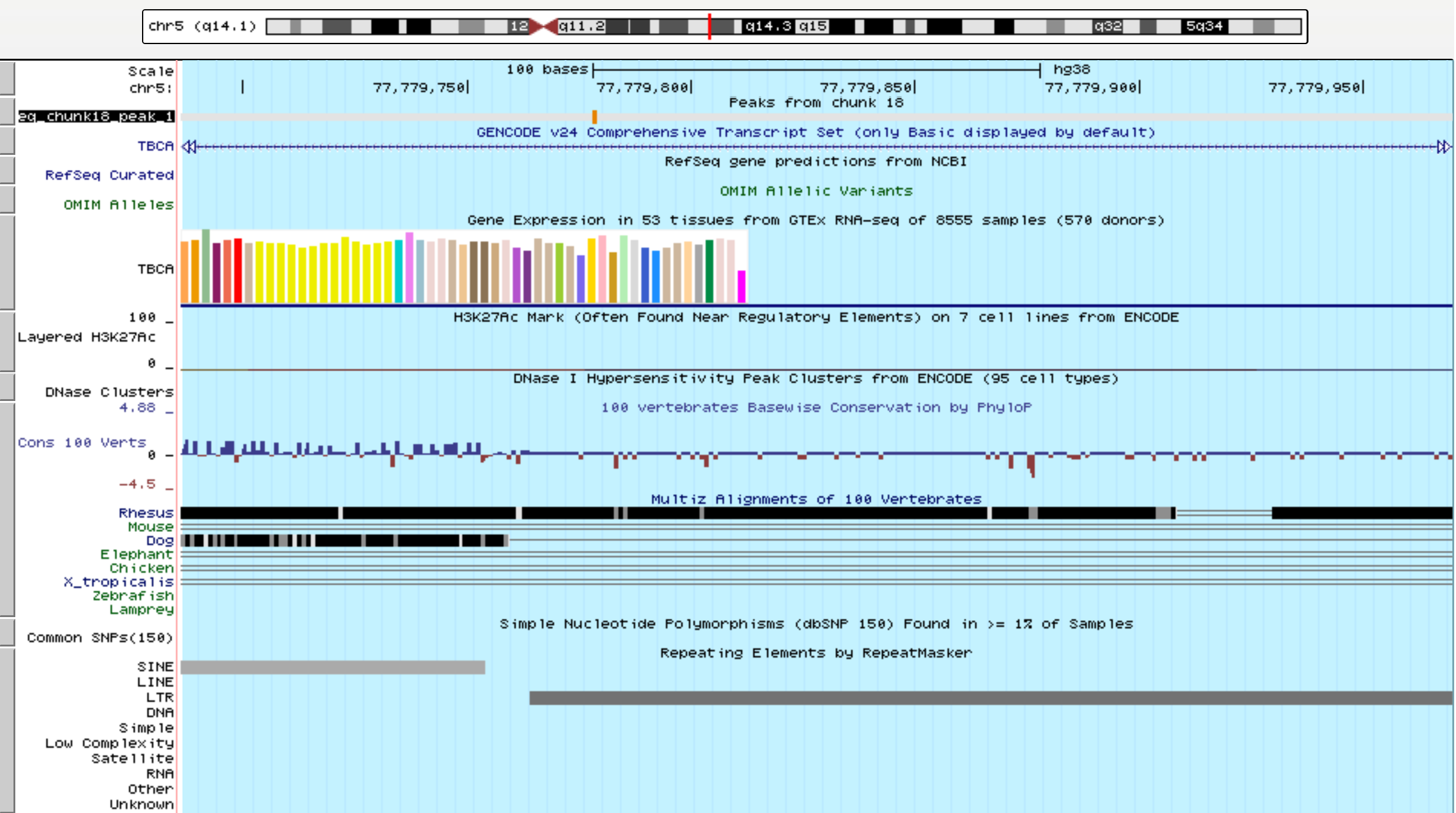
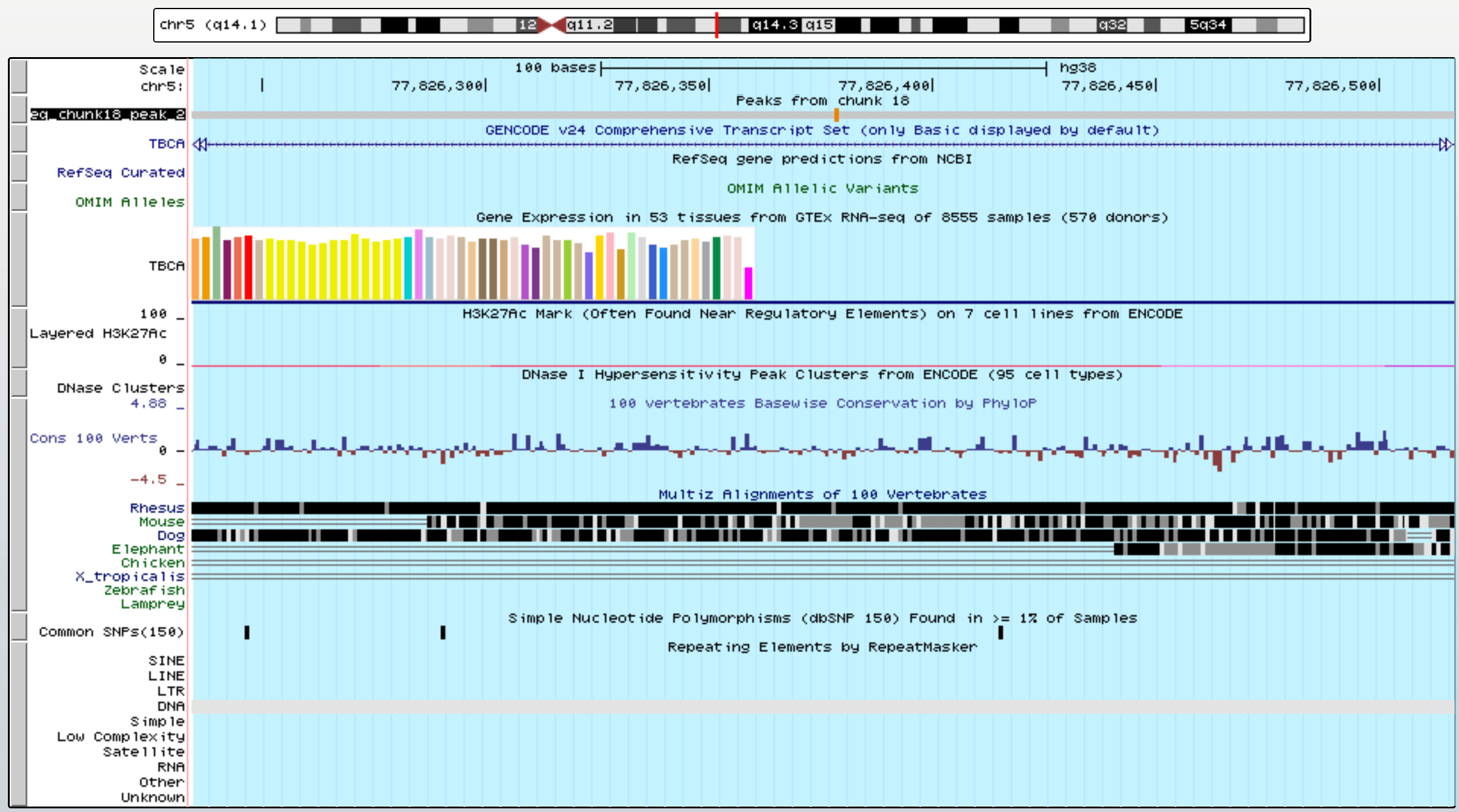
Все пики достаточно достоверные, но для анализа я возьму две самых широких пика (1 и 2), их p-value также низкие. На рисунках 3 и 4 представлены изображения двух этих пиков в UCSC Genome Browser.
|
|
| Рис. 3 Визуализация первого пика в UCSC Genome Browser |
|
|
| Рис. 4 Визуализация второго пика в UCSC Genome Browser |
Как видно, пики попали на ген TBCA - tubulin folding cofactor A, причем, оба пика попали на экзон. Продукт гена TBCA - один из 4-х белков, отвечающих за правильный фолдинг бета-тубулина из интермедиатов.