Реконструкция филогении по РНК. Паралоги
1. Построение дерева по нуклеотидным последовательностям
Нуклеотидные последовательности 16S рРНК всех выбранных бактерий были взяты в банке данных NCBI.
Последовательности были выравнены в Jalview; на основе выравнивания программой MEGA было построено дерево (методом Максимального правдоподобия). Результат представлен на рисунке 1.
|
|
| Рис. 1 Полученное дерево (ML) |
Видно, что построенное дерево не соответветствует реальному: вместо нетривиальной ветви {CLOTE, CLOBA, LACAC, STRP1, STAAR} vs {BACAN, GEOKA}
на дереве притствует ветвь {CLOTE, CLOBA, LACAC, STRP1, GEOKA} vs {BACAN, STAAR}.
Причиной неточности, вероятно, является то, что при анализе нуклеотидных последовательностей не учитываются синонимичные замены и замены на аминокислоу с аналогичными свойствами -
аргумент в пользу выравнивания белковых последовательнотей.
Также заметим, что у неправильной ветви довольно низкая поддержка бутстрепа.
2. Построение и анализ дерева, содержащего паралоги
Протеомы бактерий были записаны в один файл. Далее был проведен поиск гомологов белка CLPX_BACSU программой blastp. Первые 18 находок приведены в таблице №1.
| Таблица №1 Найденные гомологи |
|
| Мнемоника | Продукт |
| CLPX_BACAN | ATP-dependent Clp protease ATP-binding subunit ClpX |
| CLPX_GEOKA | ATP-dependent Clp protease ATP-binding subunit ClpX |
| CLPX_STAAR | ATP-dependent Clp protease ATP-binding subunit ClpX |
| CLPX_CLOTE | ATP-dependent Clp protease ATP-binding subunit ClpX |
| CLPX_CLOBA | ATP-dependent Clp protease ATP-binding subunit ClpX |
| Q5FKR6_LACAC | ATP-dependent Clp protease ATP-binding subunit ClpX |
| J7M389_STRP1 | ATP-dependent Clp protease ATP-binding subunit ClpX |
| CLPX_STRP1 | ATP-dependent Clp protease ATP-binding subunit ClpX |
| HSLU_LACAC | ATP-dependent protease ATPase subunit HslU |
| HSLU_GEOKA | ATP-dependent protease ATPase subunit HslU |
| HSLU_BACAN | ATP-dependent protease ATPase subunit HslU |
| HSLU_STAAR | ATP-dependent protease ATPase subunit HslU |
| Q5L436_GEOKA | ATP-dependent Clp protease ATPase subunit |
| Q81VV9_BACAN | ATP-dependent Clp protease ATP-binding subunit ClpC |
| Q890L5_CLOTE | Negative regulator of genetic competence mecB/clpC |
| CLPL_STAAR | ATP-dependent Clp protease ATP-binding subunit ClpL |
| CLPC_STAAR | ATP-dependent Clp protease ATP-binding subunit ClpC |
| Q899V4_CLOTE | Negative regulator of genetic competence mecB |
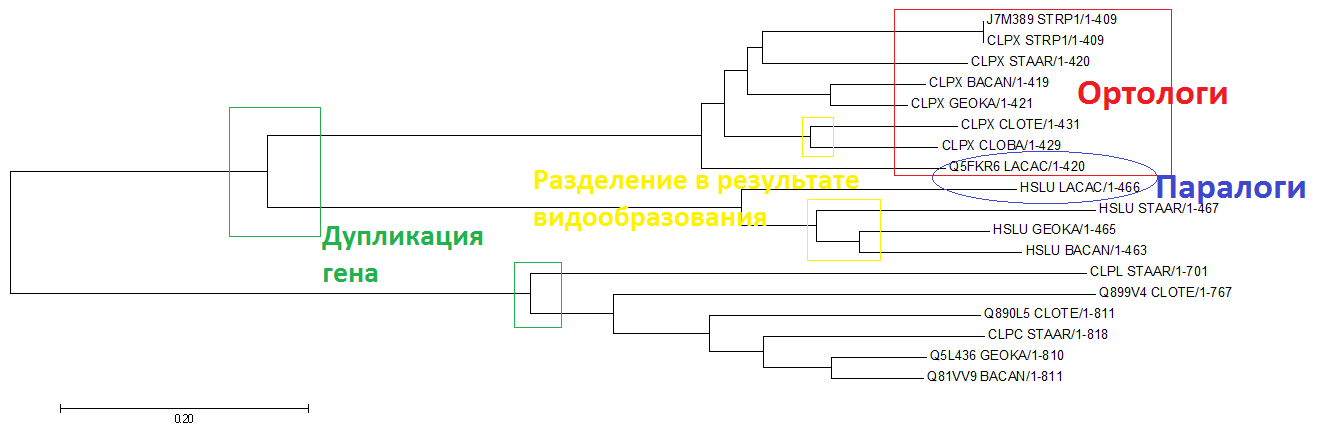
Вышеперечисленные белковые последовательности были выравнены в Jalview, затем по ним было построено дерево программой MEGA методом Neighbor-joining (рис. 3)
|
|
| Рис. 1 Полученное дерево (NJ) |
На рисунке 2 приведены примеры ортологов и паралогов; также отмечены такие эволюционные события как дупликаци гена и разделение путей эволюции белков в результате видообразования.