Геномное окружение. База данных GO
1. Получение информации о КОГе, к которому относится белок
В данном задании нужно использовать для работы данный мне в первом семестре белок YP_400610. Это альдегид декарбонилаза Synechococcus elongatus PCC 7942.
Однако после изменения системы идентификаторов в NCBI и т.д. по этому идентификатору последовательности нет, и со страницы генома данного организма при поиске этого белка идет ссылка по идентификатору
WP_011378104.
Но сервис CDD не выдал КОГов для данного белка, поэтому пришлось использовать другой. NP_953118 -
гидролаза HAD суперсемейства Geobacter sulfurreducens PCA. При помощи сервиса CDD (Conserved Domain Database) был получен список находок, некоторые из которых относили белок к определенному КОГу.
Такие хиты были отобраны, и для каждого из соответствующих КОГов было получено его название и функциональная категория, к которой он принадлежит.
Это проводилось при использовании последнего релиза базы данных по КОГам (Таблица 1).
| Таблица №1 Краткая информация о найденных КОГах |
||||
| ID | E-value | Интервал | Название | Функциональная категория |
| COG0546 | 7.31e-30 | 8-188 | Phosphoglycolate phosphatase Фосфоглюколат фосфатаза | С - Energy production and conversion Производство и преобразование энергии |
| COG0637 | 5.43e-26 | 8-209 | Beta-phosphoglucomutase бета-фосфоглюкомутаза | GR - Carbohydrate transport and metabolism Транспорт и метаболизм углеводов |
| COG1011 | 1.69e-19 | 8-209 | FMN phosphatase YigB FMN фосфатаза YigB | H - Coenzyme transport and metabolism Транспорт и метаболизм коферментов |
| COG0241 | 2.95e-03 | 146-209 | Histidinol phosphatase Гистидинол фосфатаза | E - Amino acid transport and metabolism Транспорт и метаболизм аминокислот |
2. Визуализация геномного окружения
Для визуализации был выбран КОГ с наилучшим значением e-value – COG0546. С помощью сервиса
STRING были предсказаны функциональные взаимодействия между КОГами в виде графа (Рис.1).
Входным параметром служил COG ID = 'COG0546'. Настройки же были оставлены по умолчанию.
|
|
| Рис. 1 Граф всех взаимодействий COG0546 |
Цвет ребер также имет смысл (Рис. 2).
|
|
| Рис. 2 Часть легенды изображенного на Рис. 1 графа. Розовые ребра отражают экспериментально доказанные взаимосвязи, голубые - взаимосвязи, информация о которых получена из курируемых баз данных. Ярко-зеленые - соседство в геноме, красные - слияние генов, синие - совместную встречаемость. Светло-зеленые ребра означают совместное упоминание данных белков в Pub-Med, черные - коэкспрессию, а светло-синие - гомологию. |
|
|
| Рис. 3 Обозначения КОГов и сила свидетельств связанности |
|
|
| Рис. 4 Геномное окружение для COG0546 |
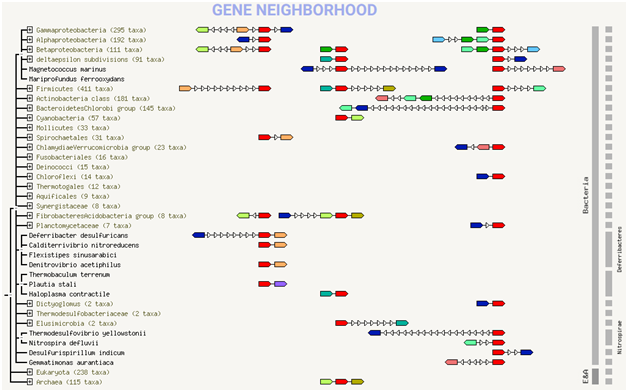
По Рис.4 видно, что у COG0546 отсутствует консервативное геномное окружение, т.е. нет постоянных соседей.
|
|
|
| Рис. 5 Совместная встречаемость в бактериях | Рис. 6 Совместная встречаемость в археях |
На Рис.5 видно, что и совместной встречаемости COG0546 с каким-то другим КОГом нет.
3. Отнесение гидролазы HAD суперсемейства из Geobacter sulfurreducens PCA к терминам GO
С помощью инструмента AmiGO поиском BLAST необходимо обнаружить в БД GO белок, который наиболее похож на исследуемый.
Первой находкой blast оказался именно мой белок – HAD-superfamily hydrolase, subfamily IA, variant 1 из Geobacter sulfurreducens PCA с e-value = 1,8е-110.
В Таблице 2 указаны термины, ассоциированные с данным белком.
| Таблица №2 Термины, ассоциированные с данным белком |
|||
| Аспект | Идентификатор GO | Название термина | Код типа достоверности |
| Биологический процесс | GO:0008152 | Metabolic process, Метаболический процесс | ISS |
| Молекулярная функция | GO:0016787 | Hydrolase activity, гидролазная активность | ISS |
Объяснения встречающихся в Таблице 2 кодов достоверности представлены в Tаблице 3.
| Таблица №3 | ||
| Код типа достоверности | Расшифровка кода типа достоверности | Объяснение |
| ISS | Inferred from Sequence or structural Similarity | Это код достоверности хороший и, как видно из названия кода, строит связи с термином на основе последовательностей. В ISS входят еще 3 типа: inferred from Sequence Orthology (ISO), inferred from Sequence Alignment (ISA), inferred from Sequence Model (ISM). |