Определение вторичной структуры
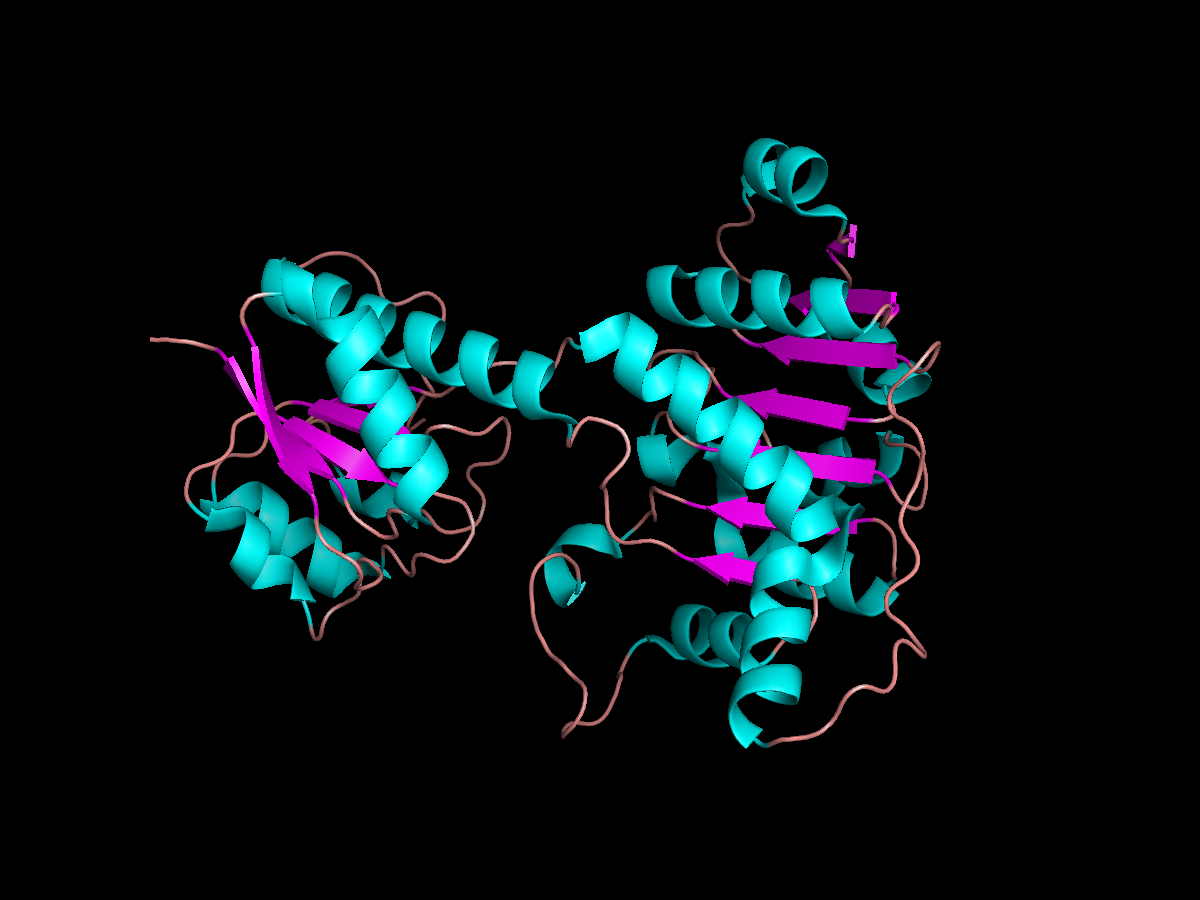
На рисунке 1 представлены вторичные структуры в составе дегидрогеназы.

Для анализа были выбраны 4 структуры: 2 альфа-спирали (helix 1 и 2) и 2 бета-листа (sheet 1 и 2) (рис. 2, выделены цветами). Информация о границах вторичных структур указана в pdb-файле. Также вторичные структуры были предсказаны при помощи программы DSSP (установлена на kodomo как mkdssp). В результате работы программы был получен файл 3wwy_dssp.out. На основании сравнения границ четырёх элементов вторичной структуры была составлена таблица №1. Также на рисунке 2 представлено расположение выбранных элементов по pdb-файла и по предсказанию программой.
|
Таблица №1. Сравнение границ некоторых структур в исходном pdb-файле и в файлах с предсказаниями программы DSSP |
||
Структура |
Границы в PDB |
Границы в DSSP |
Helix 1 (красная) |
GLY`155-PHE`168 |
LYS`156- GLY`167 |
Helix 2 (желтая) |
THR`303-GLU`323 |
LYS`304- VAL`322 |
Sheet 1 (синий) |
THR`4-PHE`8 PHE`28-PHE`32 VAL`50-GLY`53 LEU`74-MET`77 LYS`95-VAL`97 |
THR`4-PHE`8 PHE`28-PHE`32 VAL`50-GLY`53 LEU`74-MET`77 LYS`95-VAL`97 |
Sheet 2 (зеленый) |
THR`148-ILE`152LYS`171-TYR`175ILE`203-LEU`206 VAL`230-ASN`234 ILE`255-LEU`260 VAL`293-ILE`295 |
THR`148-ILE`152LYS`171-TYR`175ILE`203-LEU`206 VAL`230-ASN`234 ILE`255-LEU`260 VAL`293-ILE`295 |
Из таблицы видно, что определение мало отличается от pdb-файла. Бета-листы были определены абсолютно так же, как и в PDB, а в случае альфа-спиралей - разметка отличается всего на 1 аминокислоту: не включаются концы спиралей. Это можно считать не ошибкой программы, а тем, как альфа-спираль определяется.
|
|
|
Рис. 2. Положение выбранных элементов вторичной структуры на основании разметки из pdb-файла (слева) и предсказаний DSSP (справа)