Uronate dehydrogenase (isolated from Agrobacterium fabrum (A. tumefaciens) strain С58 / ATCC 33970)
Общая информация о белке
Краткое описание белка
Уронатдегидрогеназа - НАД-связанный фермент, принадлежащий к классу оксидоредуктаз. Он функционирует в цитоплазме клетки и представляет собой гомодимер из двух субъединиц [1].Функциональная роль
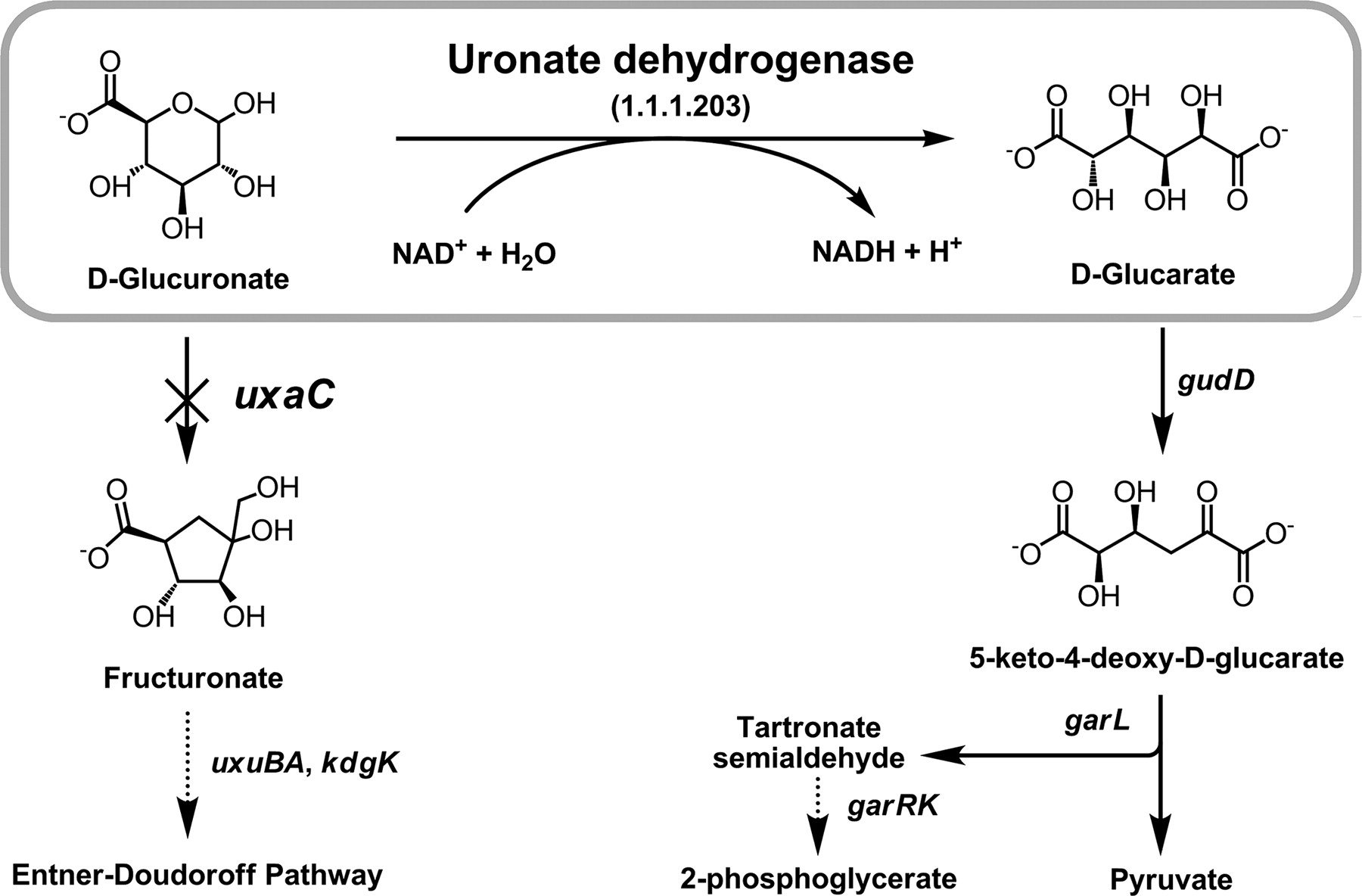
Катализирует реакцию окисления D-глюкуроновой кислоты до D-глюконо-1,5-лактона. Переносит протоны на никотинамидадениндинуклеотид (далее - NAD+). D-глюкуроновая и D-галактоуроновая кислоты способны изомеризоваться в D-фруктоуронат и D-тагатуронат соответственно и вступать в метаболический путь Энтнера-Дудорова [3]. Продукт, преобразовываясь в пируват, вступает в гликолиз [1].Структура
Белок состоит из 265 аминокислотных остатков. Как уже было сказано, он состоит из двух идентичных субъединиц (рис.2B). Каждая из них содержит три одинаковые цепи. Цепь в свою очередь содержит 7 бета-листов и 7 альфа-спиралей (рис. 2A) [2].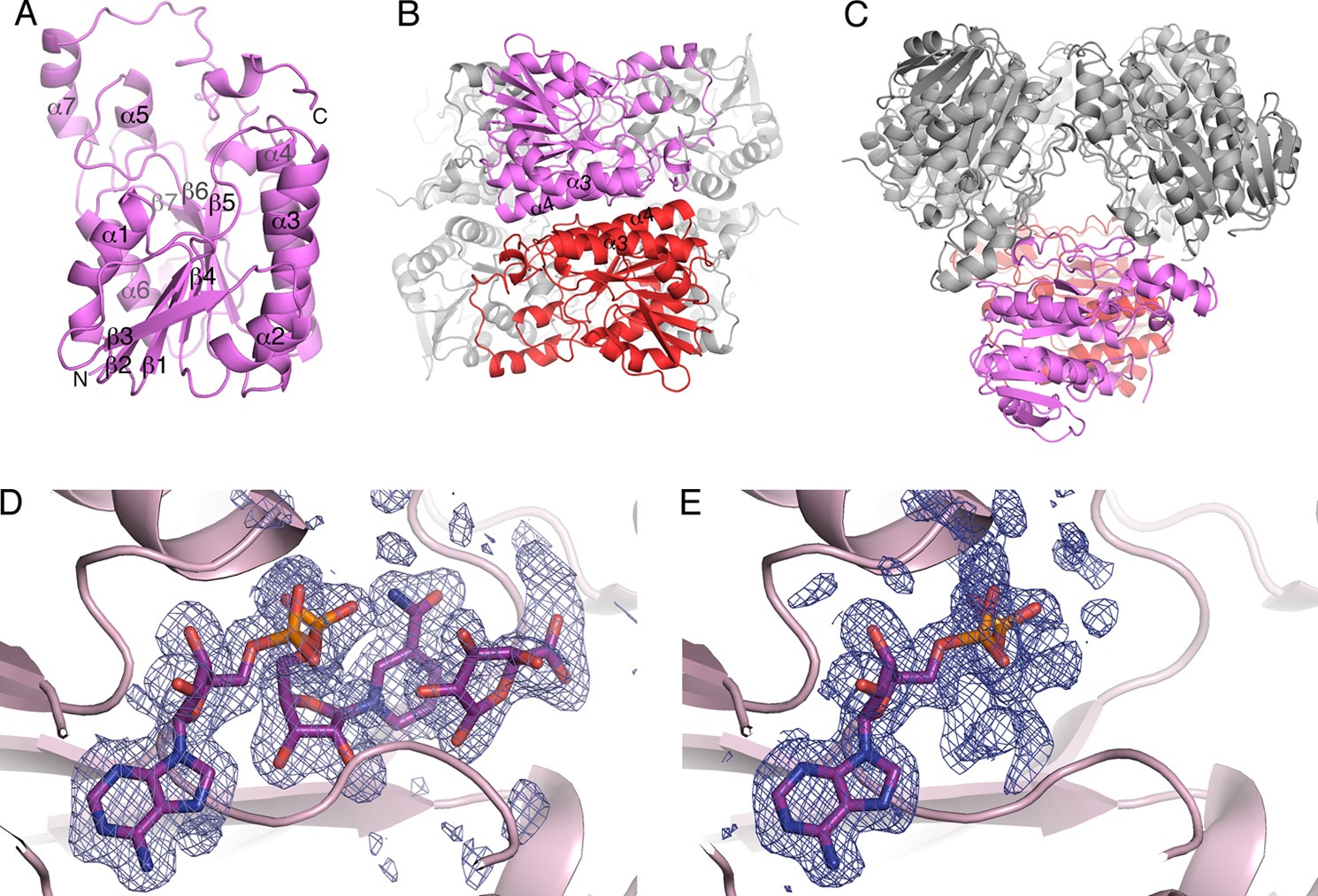
Организм, которому принадлежит белок
Белок был выделен из бактерии Agrobacterium fabrum (A. tumefaciens), штамм С58 / ATCC 33970.Систематическое положение организма
BacteriaProteobacteria
Alphaproteobacteria
Rhizobiales
Rhizobiaceae
Rhizobium/Agrobacterium group
Agrobacterium
Образ жизни и местообитание организма
Род Agrobacterium - грам-отрицательные бактерии, являющиеся фитопатогенами, способными провоцировать ненормальные разрастания тканей растений - опухоли и галлы.А. tumefaciens вызывает появление галлов в кроне древесных двудольных растений.
Вирулентность бактерии обуславливается переносом так называемой Ti-плазмиды в геном растительного организма. Эта способность может быть использована на практике для встраивания любого гена в ДНК растения, поэтому секвенирование генома A. tumefaciens представляет особый интерес [4].
| Информация о белке, взятая из UniProt | ||||
| UniProtKB | Swiss-Prot | |||
| UniProt ID | URODH_AGRFC | |||
| UniProt AC | Q7CRQ0 | |||
| EMBL AC | AE007870, bk006462 | |||
| PDB ID | 3RFT, 3RFV, 3RFX | |||
| Длина белка | 265 аминокислотных остатков | |||
| Молекулярная масса белка | 29047 MW (масса одной субъединицы) | |||
| Рекомендуемое название | Uronate dehydrogenase | |||
| Поиск информации в UniProt | ||||
| Запрос | Swiss-Prot | TrEMBL | Выдача | |
| name:"uronate dehydrogenase" AND existence:"evidence at protein level" | 3 | 0 | Все записи об уронатдегидрогеназах, чье существование подтверждено непосредственным наблюдением | |
| name:"uronate dehydrogenase" existence:"Evidence at protein level [1]" NOT length:[1 TO 265] | 2 | 0 | Все записи об уронатдегидрогеназах, чье существование подтверждено непосредственным наблюдением и чья длина превышает длину исследуемого белка | |
| name:"uronate dehydrogenase" existence:"Evidence at protein level [1]" database:(type:pdb) | 1 | 0 | Все записи об уронатдегидрогеназах,чье существование подтверждено непосредственным наблюдением и для которых известна PDB-запись | |
| annotation:(type:pathway "d-galacturonate degradation") | 5 | 0 | Все записи о белках, вовлеченных в деградацию D-галактоуроната | |
| family:"nad p -dependent epimerase dehydratase family" annotation:(type:"ph dependence" "optimum ph is 8") | 8 | 0 | Все записи о белках из того же семейства, имеющие оптимальный рН, равный 8 | |
| organism:"agrobacterium fabrum strain c58 atcc 33970" existence:"Evidence at protein level [1]" | 60 | 125 | Все записи о белках исследуемого организма, чье существование подтверждено непосредственным наблюдением | |
Работа с белком в UniRef
В нижней части страницы по ссылке “Sequence clusters (UniRef)” была найдена информация о кластерахИз каких организмов взяты белки, содержащиеся в кластерах?
Известно, что, в соответствии с молекулярными данными, род Rhizobium на самом деле содержит множество видов рода Agrobacterium [5], так что можно сделать вывод, что белок широко распространен у бактерий, относящихся к симбионтам или паразитам высших растений.
Каждый из трех кластеров содержит как просмотренные человеком (reviewed), так и автоматически сгенерированные с нуклеотидной последовательности (unreviewed) последовательности белков.
В кластере, относящимся к UniRef100, находятся самая длинная (seed) и наиболее хорошо аннотированная последовательности (representative).
Изучаемый белок является репрезентативной последовательностью. Это может быть связано с тем, что Agrobacterium tumefaciens легче всего выращивать в культуре, значит, проще всего изучать ее протеом. Бактерия является эндобионтом, так что средой для роста могут служить клетки растения, т.е. задача заключается в том, чтобы вырастить растительные клетки в искусственной среде. Это, в свою очередь, не должно быть очень сложно, так как бактерия, живущая в них, запускает быстрый рост клеток и вызывает разрастания тканей.
Сид принадлежит тому же виду бактерий. Длина сида превышает длину репрезентативной последовательности всего на два нуклеотида, при этом он имеет статус unreviewed. Поэтому возникающее несоответствие можно счесть ошибкой. Значит, одна и та же последовательность (UniProt ID: Q7CRQ0) является репрезентативной и самой длинной одновременно.
| Записи UniRef о кластерах, содержащих уронатдегидрогеназу | ||||
| Раздел UniRef | Название | Размер | ID | |
| UniRef50 | Cluster: Uronate dehydrogenase | 340 | UniRef50_Q7CRQ0 | |
| UniRef90 | Cluster: Uronate dehydrogenase | 99 | UniRef90_Q7CRQ0 | |
| UniRef100 | Cluster: Uronate dehydrogenase | 3 | UniRef100_Q7CRQ0 | |
Сравнение протеомов
В данном разделе сравниваются протеомы Agrobacterium tumefaciens и Pseudomonas syringae pv. tomato (strain ATCC BAA-871 / DC3000). Оба протеома являются референсными.| Информация о сравниваемых протеомах | ||||
| Proteome ID | Organism | Protein count | CPD (Complete Proteome Detector) | Genome representation (RefSeq) |
| UP000000813 | Agrobacterium fabrum (strain C58 / ATCC 33970) (Agrobacterium tumefaciens (strain C58)) | 5344 | Standart | full |
| UP000002515 | Pseudomonas syringae pv. tomato (strain ATCC BAA-871 / DC3000) | 5431 | Standart | full |
| Результаты поиска функциональных групп белков | ||||
| Запрос | Reviewed | Unreviewed | Выдача | Процент от общего числа белков, % |
| annotation:(type:transmem) AND organism:"Agrobacterium fabrum (strain C58 / ATCC 33970) (Agrobacterium tumefaciens (strain C58)) [176299]" AND proteome:up000000813 | 64 | 997 | Трансмембранные белки Agrobacterium fabrum (strain C58 / ATCC 33970) (Agrobacterium tumefaciens (strain C58)) | 19,85 |
| ec:* AND organism:"Agrobacterium fabrum (strain C58 / ATCC 33970) (Agrobacterium tumefaciens (strain C58)) [176299]" AND proteome:up000000813 | 411 | 384 | Ферменты Agrobacterium fabrum (strain C58 / ATCC 33970) (Agrobacterium tumefaciens (strain C58)) | 14,88 |
| d-galacturonate degradation via prokaryotic oxidative pathway AND organism:"Agrobacterium fabrum (strain C58 / ATCC 33970) (Agrobacterium tumefaciens (strain C58)) [176299]" | 3 | 0 | Белки, вовлеченные в окислительную деградацию D-галактоуроната Agrobacterium fabrum (strain C58 / ATCC 33970) (Agrobacterium tumefaciens (strain C58) | 0,056 |
| annotation:(type:transmem) AND organism:"Pseudomonas syringae pv. tomato (strain ATCC BAA-871 / DC3000) [223283]" AND proteome:up000002515 | 49 | 965 | Трансмембранные белки Pseudomonas syringae pv. tomato (strain ATCC BAA-871 / DC3000) | 18,67 |
| ec:* AND organism:"Pseudomonas syringae pv. tomato (strain ATCC BAA-871 / DC3000) [223283]" AND proteome:up000002515 | 455 | 477 | Ферменты Pseudomonas syringae pv. tomato (strain ATCC BAA-871 / DC3000) | 17,16 |
| d-galacturonate degradation via prokaryotic oxidative pathway organism:"pseudomonas putida strain atcc 47054 dsm 6125 ncimb 11950 kt2440" | 1 | 0 | Белки, вовлеченные в окислительную деградацию D-галактоуроната Pseudomonas syringae pv. tomato (strain ATCC BAA-871 / DC3000) | 0,018 |
Выводы
В двух протеомах содержится примерно одинаковое количество белков, относящихся к группе трансмембранных и обладающих энзиматической активностью. Однако в протеоме A. tumefaciens нашлось аж три белка, способных участвовать в окислительной деградации D-галактоуроната, в то время как у P. syringae такой белок всего один.Также хочется отметить, что среди трансмембранных белков соотношение последовательностей, имеющих статус "reviewed", и последовательностейсо статусом "unreviewed" равно 0,064 и 0,05 у двух бактерий, а для белков с энзиматической активностью этот показатель примерно равен единице у обоих организмов. Это говорит о том, что ферменты изучаются и аннотируются больше трансмембранных белков.