Работа с пангеномами в NPG explorer
Подготовка к работе
| Выбранный штамм | Число хромосом | AC сборки в EMBL-EBI | AC последовательности в EMBL-EBI |
| Mycobacterium tuberculosis H37Rv | 1 | GCF_000195965.2 | AL123456 |
| Mycobacterium tuberculosis strain CG24 | 1 | GCF_017901105.1 | CP072761 |
| Mycobacterium tuberculosis strain 3-0096P6C4 | 1 | GCF_014899995.1 | CP041827.1 |
Запуск NPGE
| Файл | Значение |
| pangenome.info | Общая информация о всех блоках |
| npge.conf | Файл с параметрами MIN_IDENTITY, MIN_LENGTH и др. |
| nj-global-tree.tre | Древо геномов |
| genomes.tsv | Файл с информацией о геномах |
| features.bs | Информация о генах |
| pangenome.bs,pangenome.bi | Подробное описание блоков |
| consensuses.fasta | Консенсусы белков |
Стабильное ядро нуклеотидного пангенома
| Число s-блоков | 371 (12780172 нуклеотидов) |
| Размер нуклеотидного ядра (процент нуклеотидов в ядре от числа нуклеотидов во всех геномах) | 95.93% |
| Процент консервативных колонок в объединенном выравнивании | 99,9043% |
| Длина построенных фрагментов | min: 100 max: 111720 |
| Процент числа колонок в объединенном выраванивании s-блоков от суммарного числа колонок во всех блоках | 96.79% |

Крупные делеции
В результате были найдены 2 самых крупных h-блока, отсутствующие у штамма Mycobacterium tuberculosis strain 3-0096P6C4:
Информация о делятированных белках
| Блок | Белки |
| h2x869 | |
| h2x869h2x2399 |
В третьем штамме данные белки, возможно, переместились в другое место генома или были заменены на их альтернативные формы, т.к. большая часть их них выполняет довольно важные функции. Также возможно, что произошел горизонтальный перенос генов, за счет которого штаммы H37Rv и CG24 получили эти гены.
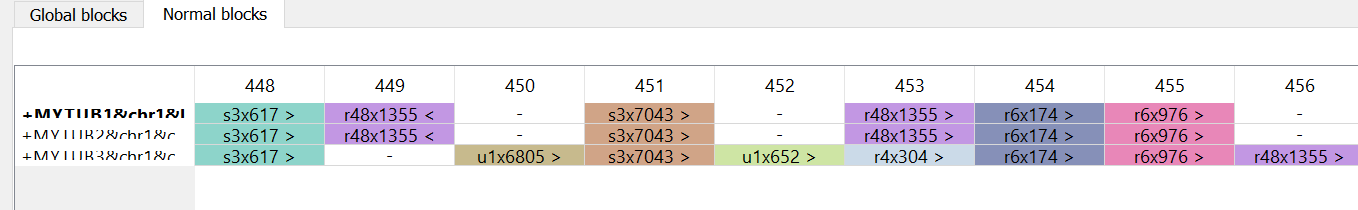
Перестановка синтений в g-блоках
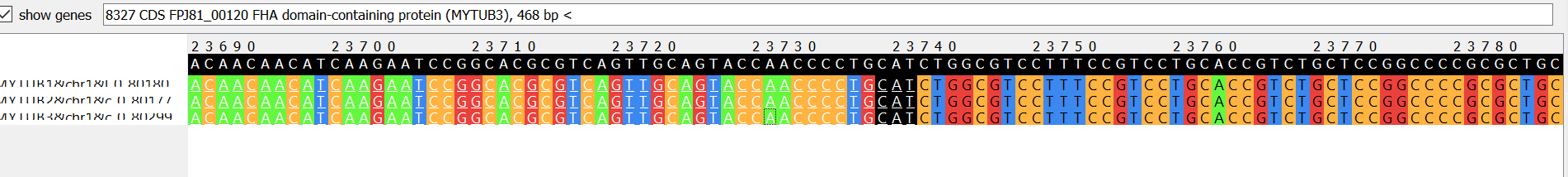
Ошибки в аннотации

Пример 2.В блоке s3x80320 у одной последовательности не уточнена в аннотации функция белка, у третьей описано только наличие FHA-домена (аннотации: CDS Rv0019c Conserved protein with FHA domain, FhaB; CDS J8670_00115 growth/cell division-associated protein FhaB; CDS FPJ81_00120 FHA domain-containing protein) несмотря на то, что они хорошо выравниваются, т.е. аннотация неполная.