Сборка de novo
Подготовка к работе
Архив с чтениями был скачан с помощью команды:
wget https://www.ebi.ac.uk/ena/browser/view/SRR4240358
Подготовка чтений программой trimmomatic
Объединение файлов с информацией об адаптерах:
cat *.fa > adapters.fasta
Удаление адаптеров:
java -jar /usr/share/java/trimmomatic.jar SE SRR4240358.fastq.gz -threads 5 no_adapter.fastq.gz -trimlog trim_adapter.log ILLUMINACLIP:adapters.fasta:2:7:7
Удаление нуклеотидов с низким качеством:
java -jar /usr/share/java/trimmomatic.jar SE no_adapter.fastq.gz -threads 5 trimmed.fastq.gz -trimlog trim_poorq.log TRAILING:20 MINLEN:32
Результаты
Размеры:
Адаптеры занимали небольшую часть чтений (менее 2%), однако чистка чтений привела к удалению более чем 22% нуклеотидов.
Работа с программой velveth
velveth kmers_velvet 31 -fastq.gz trimmed.fastq.gz -short
Сборка по созданным k-мерам:
velvetg kmers_velvet &> velvetg.log
Длина N50 - 8600 bp. В таблице ниже приведены самые длинные контиги.
| ID | Длина | Покрытие | Ссылка на последовательность |
| 56 | 19821 | 29.475859 | contig56.fasta |
| 34 | 18714 | 29.922678 | contig34.fasta |
| 40 | 16436 | 30.793624 | contig40.fasta |
Выравнивание
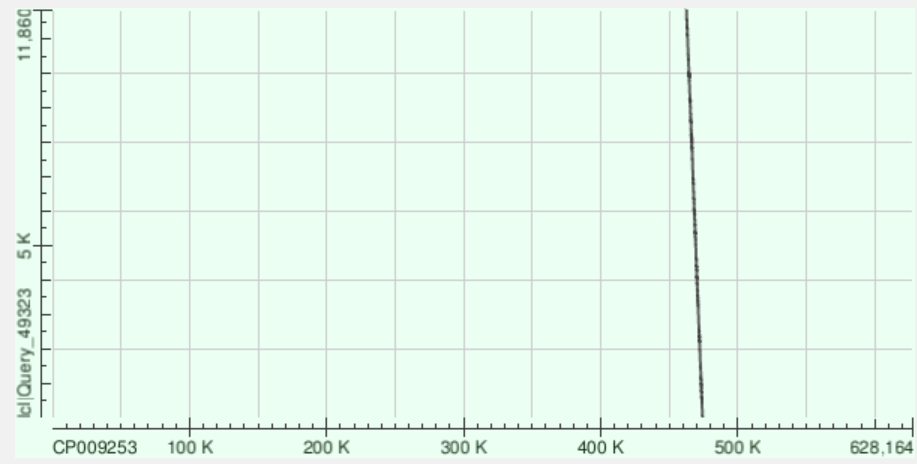
Далее был запущен BLAST с каждым из вышеописанных контигов и геномом бактерии Buchnera aphidicola.
Контиг 56
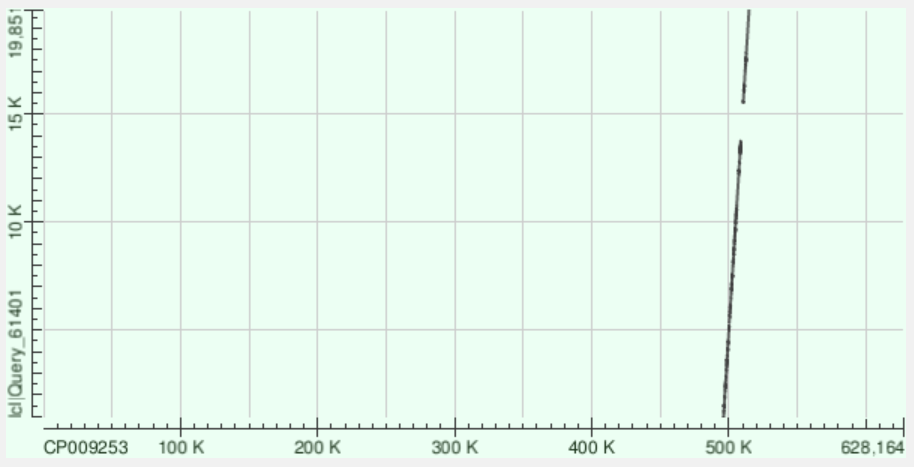
Контиг 34
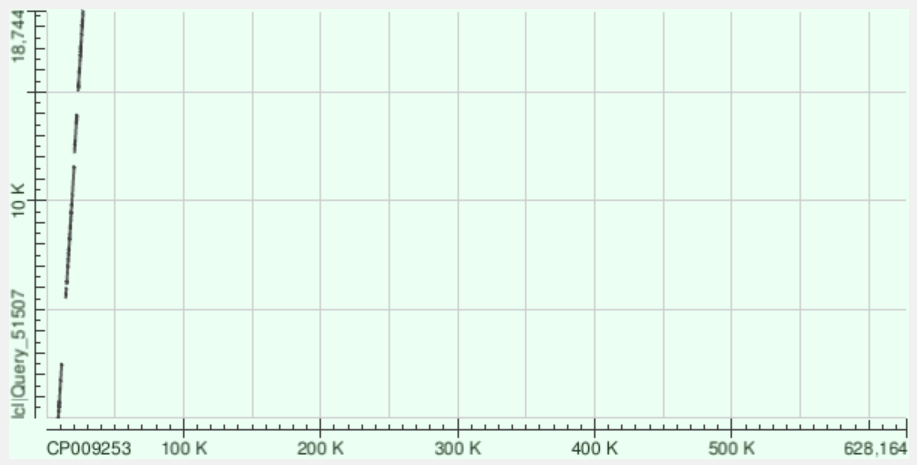
Контиг 40