Задание 1: Попробовать разные варианты BLAST для фрагмента ДНК
Выбор участка генома Danio Rerio с по крайней мере одной CDS и с не кодирующей областью:
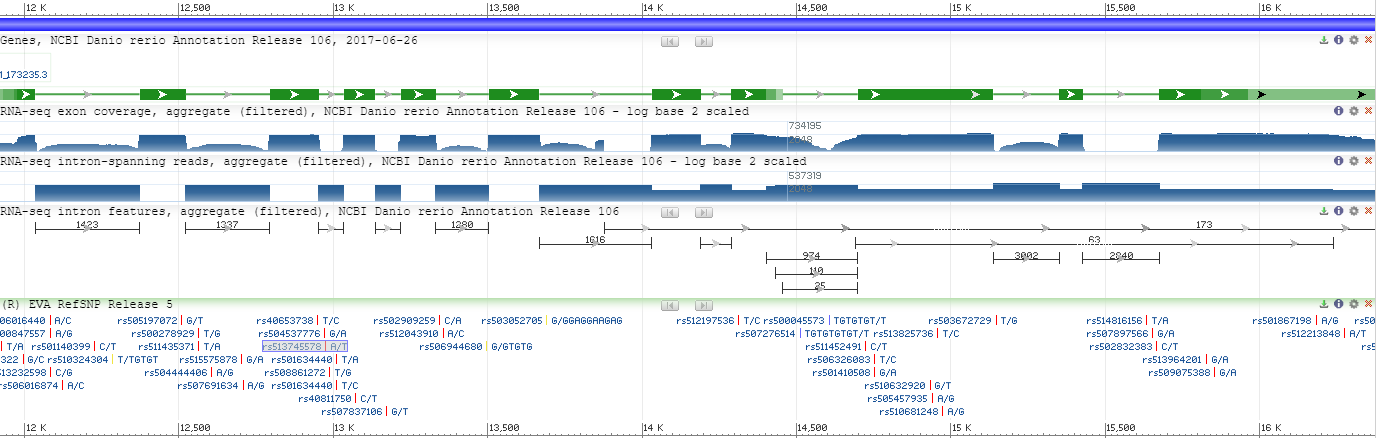
Идентификатор нуклеотидной записи: NC_007112.7
Фрагмент был взят из 1 хромосомы из сборки GCF_000002035.6
Координаты фрагмента: 11922..16373
Длина фрагмента: 4452

Данный фрагмент представляет собой ген cep97, зелёным выделены кодирующие фрагменты - экзоны, а между ними находятся интроны.
Кодируются: 2 белка (на самом деле один белок в 2-х изоформах, всё зависит от начальной координаты, кодирующей белок: либо 11932, либо 11977. мРНК начинаются на 10 нуклеотидов раньше, т.е. эти 10 нуклеотидов не кодируют белок) и одна мяРНК. Последний кусочек с чёрными стрелочками, кодирующий мРНК, не кодирует белок.
NC_007112.7[11922..16373].fasta
Поиск BLAST по эукариотическим последовательностям из родственного таксона для выбранной последовательности:
Danio rerio принадлежит к отряду Cypriniformes(Карпообразные), классу Actinopteri(Лучепёрые рыбы).
Целевой таксон: отряд Acipenseriformes(Осетрообразные)
Именения в настройках алгоритма:
blastn - искать до 1000 последовательностей
megablast - искать до 1000 последовательностей, длина слова понижена до 20
blastx - искать до 1000 последовательностей
tblastx - большое количество разных настроек алгоритма
Результаты:
blastn - 110 находок
megablast - 10 находок
blastx - 12 находок
tblastx - как бы я ни уменьшал круг поиска, процессор BLAST перегружается и выдаёт ошибку. (Добился результата работы больше 2-х часов с оптимизацией настроек, но процессор всё равно не выдерживал)
blastn - есть одинаковые результаты с megablsast, но нашёл и лучше
megablast - нашёл меньше, чем blastn, но быстрее
blastx - нашёл белки с такой же функцией, большинство из них одинакового веса (97 kDa)
tblastx - польза сомнительна, подобрать настройки может быть крайне сложно.
Основными организмами, гомологичными по данному фрагменту Danio rerio(Рыба-зебра) являются Polyodon Spathula(Веслонос) и Acipenser ruthenus(Стерлядь).
Примеры использования алгоритмов:
blastn - Поиск гомологичных участков хромосом у какой-то группы живых существ.
megablast - Секвенировали геном нового животного, надо определить к какому таксону оно ближе всего из всех возможных животных.
blastx - Определить степень консервативности белка с какой-то функцией у разных существ, ведь ДНК мутирует.
tblastx - Какими разными последовательностями кодируется белок? Мы знаем кодирующую последовательность, но пока никто не занимался последовательностью белка.
Задание 2: Найти в геноме эукариота гены основных рибосомальных РНК по далёкому гомологу
Использованные команды:
makeblastdb -in GCF_000002035.6_GRCz11_genomic.fna -dbtype nucl blastn -task blastn -query 16s_rRNA_Ecoli.fasta -db GCF_000002035.6_GRCz11_genomic.fna -out 16s_out.txt -evalue 0.001 -word_size 7 -outfmt 7 blastn -task blastn -query 23s_rRNA_Ecoli.fasta -db GCF_000002035.6_GRCz11_genomic.fna -out 23s_out.txt -evalue 0.001 -word_size 7 -outfmt 7
Названия последовательностей рРНК (они входят в состав рибосомы, их функция - помощь в трансляции), по которым проводился поиск:
>CP014225.1:complement(926804-928359)|16S_rRNA|Escherichia coli str. K-12 substr. MG1655 strain K-12
>CP014225.1:2234710-2237641|23S_rRNA|Escherichia coli str. K-12 substr. MG1655 strain K-12
Был выбран blastn. Так как надо искать гомологи в нуклеотидных последовательностях.
BLAST выдал 30 результатов для 16s и 76 результатов для 23s.
16s_out.txt
23s_out.txt
Рассмотрим выходные файлы. Начнём с 16s рРНК: значения e-value и score довольно низкие, однако мы можем выделить 9 самых длинных выравниваний (114) с приемлемым значением e-value (2.46e-09) для BLASTа таксономически далёких организмов. Однако все эти 9 выравниваний являются выравниваниями одного и того же участка 16s рРНК (887..998) E. coli на разные участки генома Danio rerio, а именно, на скаффолд и на 5 хромосому.
Теперь рассмотрим 23s рРНК. E-value и score здесь значительно больше, длина выравниваний и их количество тоже. Если анализировать аналогично с тем, как мы это делали при работе с выравниваниями 16s рРНК, то можно сделать вывод, что есть 3 участка последовательности 23s рРНК E. coli (190..290; 428..528; 2442..2613), которые скорее всего являются гомологичными участками между E. coli и Danio rerio.
Я не нашёл аннотации для объяснения результатов.
Назад