Необходимо найти достоверные гомологи белка CLPX_ECOLI среди протеомов выбранных бактерий(см. Практикум 1-2).
Сделаем локальный blastp:
makeblastdb db.fasta -dbtype prot blastp -task blastp -query CLPX_ECOLI.fasta -db db.fasta -out out.txt -evalue 0.001 -word_size 7 -outfmt 7После анализа выходной таблицы был создан список возможных гомологов (в том числе и сам CLPX_ECOLI из ECOLI со 100% совпадением):
ID: Identity: E-value: sp|P0A6H1|CLPX_ECOLI 100.000 0.0 sp|B4EU54|CLPX_PROMH 88.679 0.0 sp|Q12LA2|CLPX_SHEDO 81.412 0.0 sp|Q3SI99|CLPX_THIDA 77.349 0.0 sp|Q8UFY5|CLPX_AGRFC 70.531 0.0 sp|Q165G0|CLPX_ROSDO 68.750 0.0 sp|A1B1H7|CLPX_PARDP 71.154 0.0 sp|A1B5T0|HSLU_PARDP 40.278 3.08e-24 sp|A1B5T0|HSLU_PARDP 32.618 3.93e-21 sp|B4F171|HSLU_PROMH 46.000 8.24e-22 sp|B4F171|HSLU_PROMH 34.211 5.80e-21 sp|Q12IT8|HSLU_SHEDO 47.000 5.12e-21 sp|Q12IT8|HSLU_SHEDO 32.093 1.96e-18 sp|P0A6H5|HSLU_ECOLI 45.000 6.92e-21 sp|P0A6H5|HSLU_ECOLI 33.488 1.73e-19 sp|Q16CY2|HSLU_ROSDO 33.191 8.53e-21 sp|Q16CY2|HSLU_ROSDO 46.154 9.90e-21 sp|Q8UJ87|HSLU_AGRFC 46.078 2.27e-20 sp|Q8UJ87|HSLU_AGRFC 30.698 3.49e-18 tr|Q3SFW1|Q3SFW1_THIDA 33.475 1.68e-18 tr|Q3SFW1|Q3SFW1_THIDA 44.231 2.42e-17 tr|B4EV83|B4EV83_PROMH 25.538 1.32e-06 tr|A1B8N4|A1B8N4_PARDP 23.810 1.33e-06 tr|B4F2B3|B4F2B3_PROMH 35.897 1.65e-05 sp|P0AAI3|FTSH_ECOLI 34.615 1.91e-05 sp|Q168A2|RUVB_ROSDO 26.846 2.82e-05 tr|Q7CT50|Q7CT50_AGRFC 35.065 4.11e-05 tr|A1AZV8|A1AZV8_PARDP 36.842 8.17e-05 tr|Q3SJR4|Q3SJR4_THIDA 33.333 1.40e-04 sp|A1AZW1|RUVB_PARDP 29.921 1.49e-04 sp|P0ABH9|CLPA_ECOLI 38.202 2.08e-04 tr|Q3SJH1|Q3SJH1_THIDA 24.000 2.91e-04 tr|A1BBJ2|A1BBJ2_PARDP 30.588 5.95e-04 tr|A1AY35|A1AY35_PARDP 22.701 7.03e-04 tr|Q16C81|Q16C81_ROSDO 28.571 7.79e-04 tr|Q167Z2|Q167Z2_ROSDO 36.842 7.90e-04 tr|Q12QI8|Q12QI8_SHEDO 31.250 8.59e-042. Реконструкция и визуализация
Создадим FASTA файл с нужными последовательностями, построим дерево с помощью FastME (c параметрами: "Gamma distributed rates across sites" — No, "Starting tree" — BIONJ, "No refinement", 100 бутстреп реплик.)
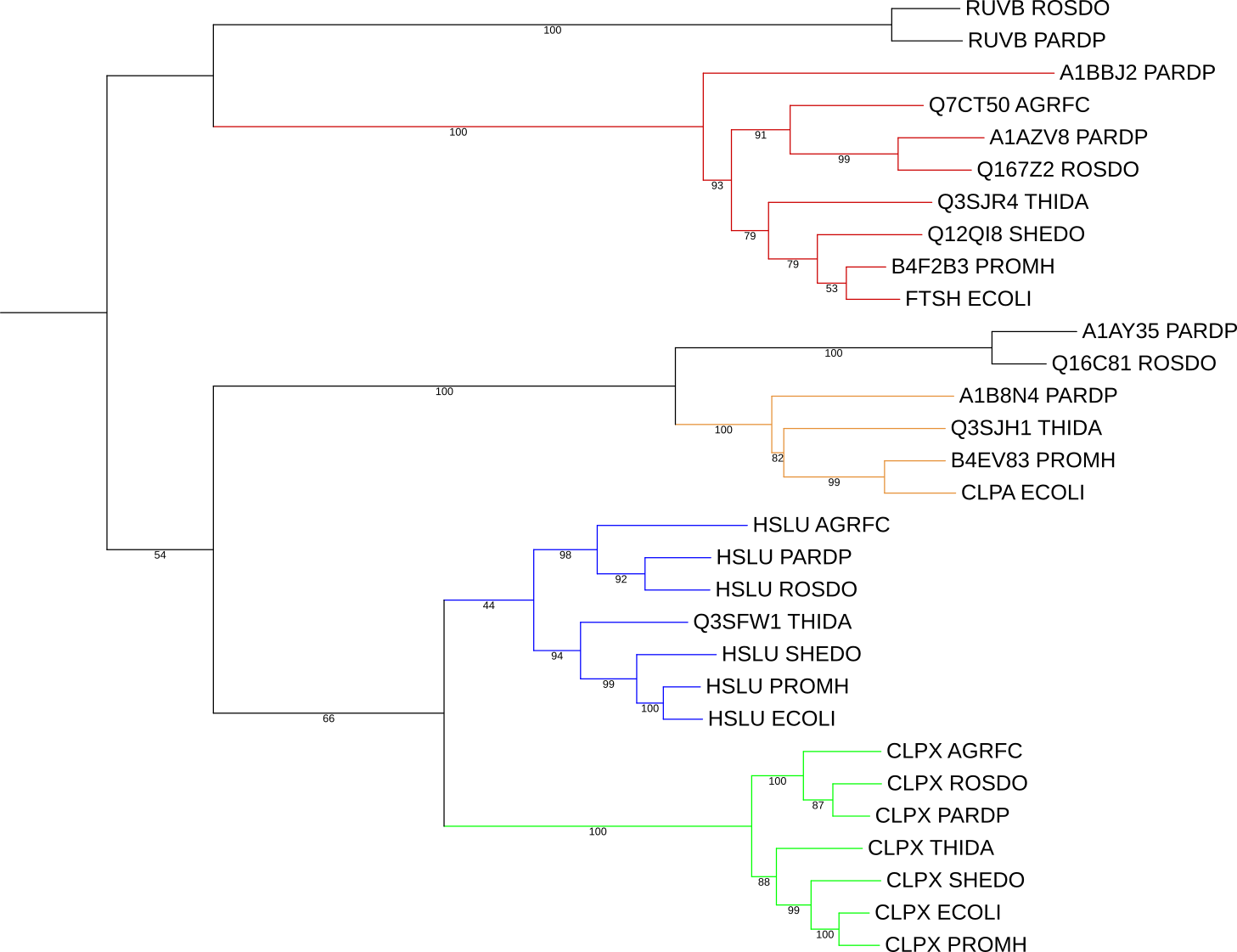
Newick формула
Пары паралогов: (2 гомологичных белка из одного организма)
1)HSLU_SHEDO и CLPX_SHEDO
2)Q3SFW1_THIDA и CLPX_THIDA
3)HSLU_AGRFC и CLPX_AGRFC
Пары ортологов: (Белки из разных организмов, разделение произошло в результате видообразования)
1)HSLU_PARDP и HSLU_ROSDO
2)CLPX_ECOLI и CLPX_PROMH
3)RUVB_PARDP и RUVB_ROSDO
Изображения дерева:


| Название | Мнемоника |
| Agrobacterium fabrum | AGRFC |
| Escherichia coli | ECOLI |
| Paracoccus denitrificans | PARDP |
| Proteus mirabilis | PROMH |
| Roseobacter denitrificans | ROSDO |
| Thiobacillus denitrificans | THIDA |
| Shewanella denitrificans | SHEDO |
Реконструированная филогения бактерий:
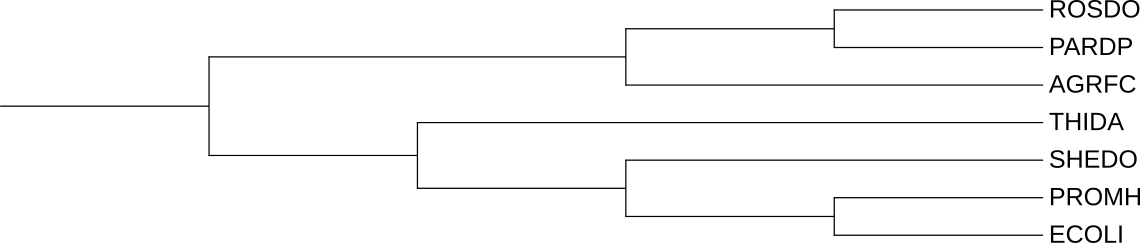
Можем наблюдать, что реконструкция филогении белков полностью совпадает с реконструкцией филогении бактерий (однако A1BBJ2 PARDP в красной группе может быть лишним, довольно спорный белок, хоть он и выполняет такую же функцию, что и A1AZV8_PARDP, и даже соседствует в клетке с ним, но кодируется не на хромосоме, а на плазмиде). А именно, это наблюдается для каждой ортологической группы, даже если она содержит не все бактерии.
В схлопнутых ортологических группах присутствуют все исследуемые бактерии (кроме жёлтой, там нет AGRFC, SHEDO, ROSDO). В ортологических группах, которые мы не схлопывали, есть только PARDP и ROSDO.