Бактерия E.Coli Хотелось найти ориджин с помощью Ori-finder, но он не работает. Из исследования OriC E.coli K-12 известна длина и структура ориджина:
Длина: 232-245 нуклеотидов.
Структура:
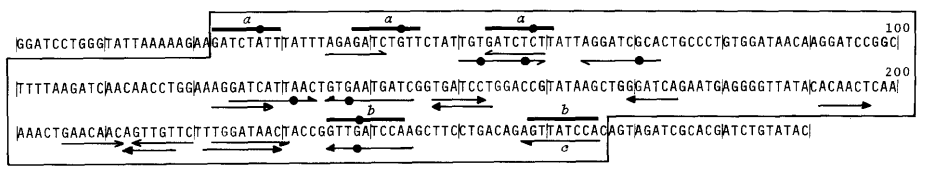
Задание 1: Краткое описание сигнала
Мы будем рассматривать ориджин репликации в качестве сигнала.
Название сигнала: OriC
В чём состоит сигнал: Это AT-богатый участок ДНК, в котором начинается процесс репликации.
Кому адресован сигнал: белок DnaA
Как должен реагировать адресат: DnaA связывается с 5 DnaA-боксами (5'-TTATNCACA-3'). После этого DnaA инициирует процесс репликации.
Сила сигнала: Сильный. Репликация ДНК - необходимый процесс для деления клетки, если бы сигнал не был сильным, то такие клетки бы "отсекались" естественным отбором.
Пример сигнала:
Бактерия E.Coli
Хотелось найти ориджин с помощью Ori-finder, но он не работает.
Из исследования OriC E.coli K-12 известна длина и структура ориджина:
Длина: 232-245 нуклеотидов.
Структура:

Использованные источники: NCBI RefSeq (Genome assembly ASM584v2), NCBI Taxonomy (Bacteria > Pseudomonadota > Gammaproteobacteria > Enterobacterales > Enterobacteriaceae > Escherichia > Escherichia coli > Escherichia coli str. K-12 substr. MG1655);
Atsuhiro Oka; Kazunori Sugimoto; Mituru Takanami; Yukinori Hirota (1980). Replication origin of theEscherichia coliK-12 chromosome: The size and structure of the minimum DNA segment carrying the information for autonomous replication. , 178(1), 9–20. doi:10.1007/bf00267207
Задание 2: Построить PWM для сигнала и оценить результаты поиска новых сайтов по этой PMW.
Будем рассматривать последовательность Козак в геноме человека.
1. С помощью скрипта скачаем 15 разных генов белков человека с отступами по 10 нуклеотидов от старт-кодона и стоп-кодона
2. Вырежем интересующий нас участок до старт-кодона(ATG) и 5 нуклеотидов после старт-кодона из наших последовательностей(их таких оказалось 12 из 15). Также сохраним отдельно исследуемые части всех 15 последовательностей, чтобы потом найти их веса по PWM.



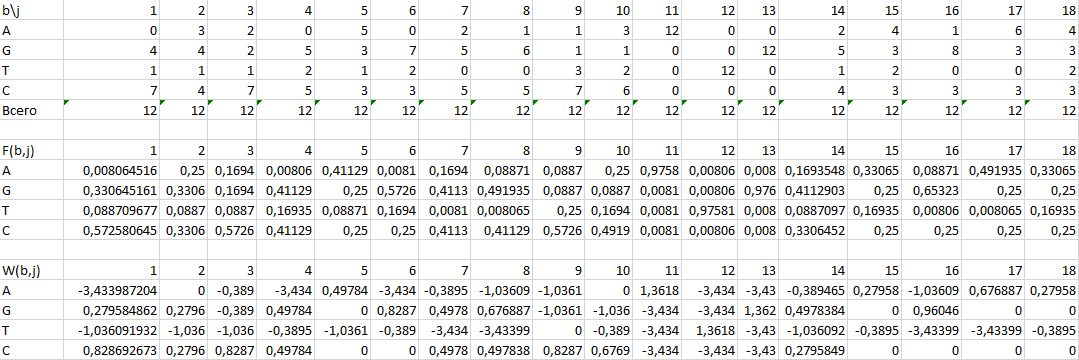
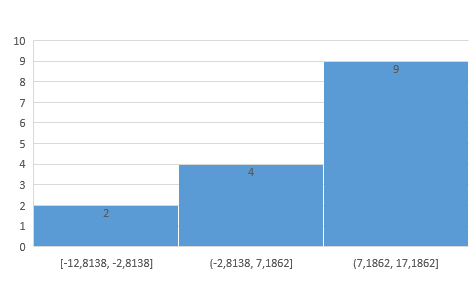
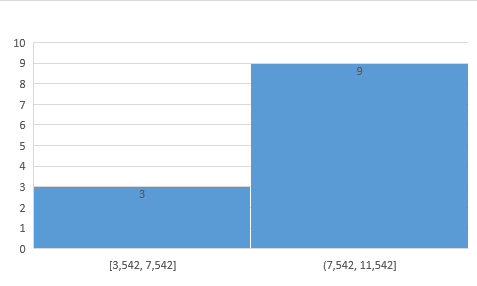
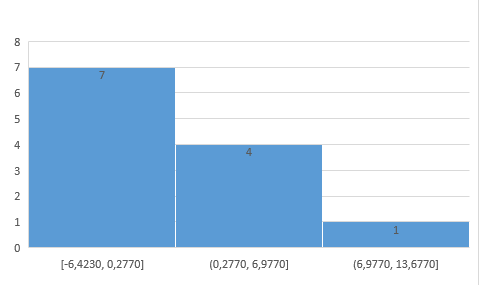
Выберем порог веса правильной находки: Пусть будет 8.
Вычислим информационное содержание IC для исследуемых последовательностей по формуле Шнайдера: Получилось число примерно 9,46. Значит IC = 10 битов.
Построим LOGO:
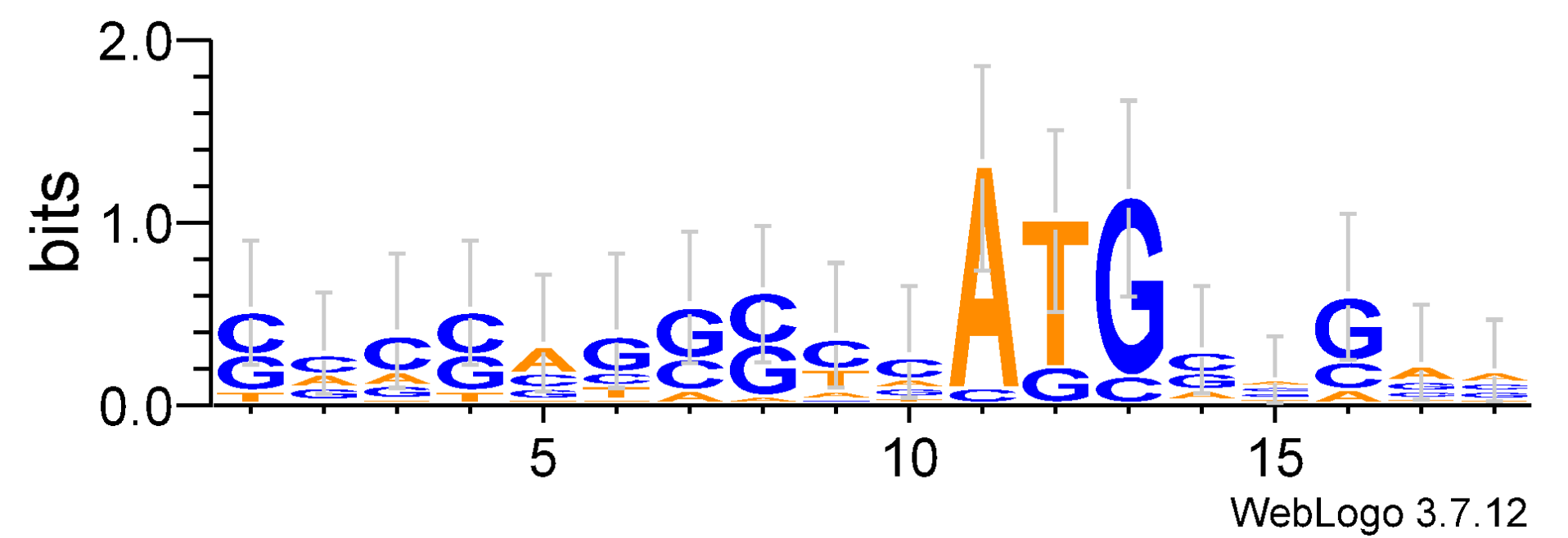
Выводы: Если взять во внимание порог веса и значения, показанные гистограммами, можно прийти к выводу, что в исследуемых последовательностях есть сигнал последовательности Козак, а в отрицательном контроле его нет. А LOGO графически показывает окрестность старт-кодона.
Назад