Доменная организация белка LEP_Ecoli (Pfam)
Схематическое изображение доменной структуры LEP_Ecoli, PDB запись: 1t7d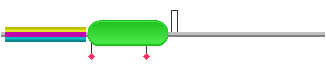
Зеленый овал - схематическое изображение домена Peptidase_S24
Трехцветный цилиндр - Pfam-B_2366
Красные ромбики - схематичное положение активных сайтов
Скобочка символизирует дисульфидный мостик
Картинка с доменами, отмеченными на пространственной структуре LEP_Ecoli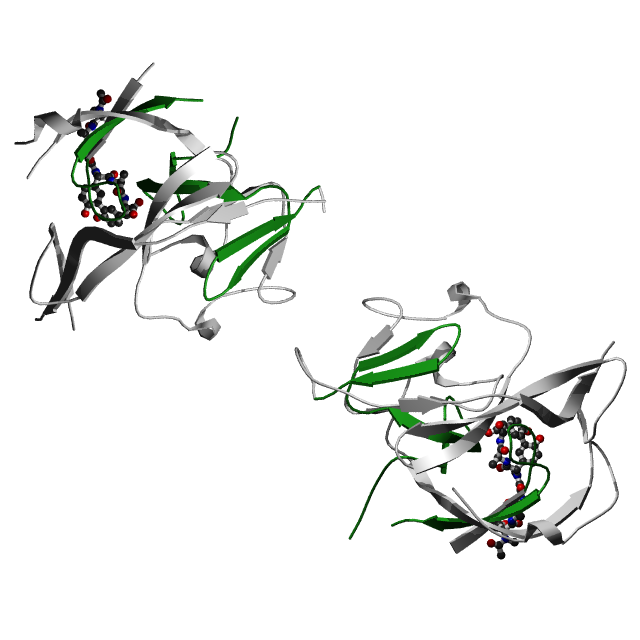
В шариковой модели изображен липопептидный ингибитор
Серым цветом отмечен домен Peptidase_S24
Зеленым цветом - Pfam-B_2366
Табличка с названием "Домены в последовательности LEP_Ecoli
|
Идентификатор Pfam
|
Название записи
|
Полное название домена
|
Положение в последовательности LEP_Ecoli
|
|
PF00717
|
Peptidase S24
|
пептидаза s24
|
86-170
|
Описание N-концевого домена белка LEP_Ecoli
Краткая характеристика домена PF00717
Протеолитические ферменты использующие каталитические свойства серина, включают широкий класс пептидаз,
таких как экзо-, эндо-, олиго- и омегапептидазы.
Их каталитические центры состоят из серина, играющего роль нуклеофила, аспарагина, как электрофила,
и гистидина, в качестве основания, причем пространственная ориентация этих аминокислот очень похожа,
несмотря на то что белки могут иметь разные укладки.
Данное семейство включает: семейство лямбда-репрессоров CI/C2y и родственных с ними белков-репрессоров бактериальных вирусов,
LexA - репрессор гена СОС-репарации,
MucA и родсвенные UmuD белки - ДНК-полимеразы,
RulA - компонент rulAB-локуса отвечающего за сопротивляемость к UV;
Табличка с названием "Организмы, в которых найдены белки с доменами PF00717"
|
Таксон
|
Количество белков с доменом PF00717
|
| Эукариоты |
Растения |
41 |
| Грибы |
55 |
| Животные |
68 |
| Бактерии |
|
2050 |
| Археи |
|
33 |
- Интересно, что:
- с доменами PF00717 известно всего 2357 белков
- чаще бывает что домен PF00717 — единственный домен в белке
- известны случаи дупликации домена PF00717.
Например Q0HJB1_SHESM

- случаев перестановки 2-х доменов местами, в то время когда один из них PF00717 - неизвестно
Изображение всех подписей в белке LEP_ECOLI
| | LEADERPTASE |
|  |
| | SPASE_I_1 |
| | SPASE_I_2 |
| | SPASE_I_3 |
| | Pept_S26A_signal_pept_1 |
| | sigpep_I_bact |
| | Pept_S24_S26_C |
| | Peptidase_S24 |
| | Peptidase_S26A |
| | Pept_S24_S26_C |
"Мотивы в белке LEP_Ecoli по данным БД InterPro"
|
Название подписи |
Положение в последовательности LEP_Ecoli |
Полное название базы данных |
* Название организации, поддерживающей данную БД. |
* Город и страна |
|
LEADERPTASE |
80 - 96; 144 - 156; 264 - 283 |
SPRINT |
|
|
|
SPASE_I_1 |
89 - 96 |
PROSITE |
Swiss Institute of Bioinformatics |
Швейцария, Женева |
|
SPASE_I_2 |
146 - 158 |
PROSITE |
Swiss Institute of Bioinformatics |
Швейцария, Женева |
|
SPASE_I_3 |
269 - 282 |
PROSITE |
Swiss Institute of Bioinformatics |
Швейцария, Женева |
|
Pept_S26A_signal_pept_1 |
70 - 308 |
PANTHER |
|
|
|
sigpep_I_bact |
63 - 305 |
CMR |
|
|
|
Pept_S24_S26_C |
75 - 324 |
CATH |
|
|
|
Peptidase_S24 |
86 - 170 |
Pfam |
|
|
|
Peptidase_S26A |
70 - 308 |
PANTHER |
|
|
|
Pept_S24_S26_C |
78 - 324 |
Superfamily |
|
|
|
На главную
Второй семестр
|



