RefSeq Идентификатор Natribaculum luteum - GCF_023008545.1.
Ссылка на страницу из базы NCBI Datasets Genome, которая соответствует этой сборке
Соответствующий идентификатор INSDC - GCA_023008545.1.
Используем полученную информацию для поиска по UniProt Proteomes:(genome_assembly:GCA_023008545.1). База данных не выдала никакого результата. Вероятно, так случилось потому, что сборка данного протоема была утверждена только в 2022 году и ее пока не успели добавить в UniProt Proteoms.
Референсный протеом для своей археи я искала в UniProt Proteomes, задав поиск по таксономическому идентифекатору семейства Natrialbaceae, поскольку протеомов как Natribaculum luteum, так и рода Natribaculum в базе данных нет: (taxonomy_id: 1644061). Таким образом я получила 98 результатов, среди которых было 30 референсных протеомов. Из 30 подходящих я искала фаворитов на основе их значений BUSCO и CPD категории. У 11 протеомов была эталонная CPD категория - Standard, среди них я выбрала протеом Natronorubrum halalkaliphilum (Proteome ID - UP000434101), так как у него был самый высокий показатель BUSCO - 99, 8%.
Задав в UniProtKB поиск по найденному идентификатору протеома, я нашла записи о всех его белках. Скачала белковые записи следующей командой:
wget 'https://rest.uniprot.org/uniprotkb/stream?compressed=true&format=txt&query=proteome:UP000434101' -O UP000434101.swiss.gz
Для того, чтобы примерно понять сколько в референсном протеоме ферментов, я воспользовалась расширенным поиском в UniProtKB. Я вбила ID протеома и добавила поиск по EC(enzyme classification), указав в этом поле *, так как нам важно количество, а не классы ферментов: (proteome:UP000434101) AND EC:*. В результате было найдено 603 записи, тогда как весь протеом состоит из 4155 белков. То есть примерно 14,51% протеома составляют энзимы. Если же в поисковом окне вбить (proteome:UP000434101) AND (cc_catalytic_activity:*), то будет получено 475 результатов, тогда энзимы составляют 11,43% протеома.
Определим количество ферментов с помощью конвейеров в BASH.
С помощью следующего конвейера было получено 605 результатов:
zgrep -E "^(ID|DE)" UP000434101.swiss.gz | grep -E "(^ID)|EC=" | grep -A1 "^ID" | grep -c "^DE"
Другой конвейер выдал значение в 475 ферментов:
zgrep -E "^(ID|CC)" UP000434101.swiss.gz | grep -E "(^ID)|CATALYTIC ACTIVITY" | grep -A1 "^ID" | grep -c "^CC"
Результаты поисков через конвейеры и UniProt практически полностью совпали. Можно сделать вывод, что поиск через каталитическую активность дает более точные результаты, так как коды EC могут встречаться не тлько в поле DE, но и в комментариях к записям, даже если сам белок не обладает каталитической активностью.
В мини-обзоре я изучала белок бактериородопсин, свойственный археям рассматриваемого семейтсва, поэтому решила поискать его в протеоме Natronorubrum halalkaliphilum с помощью следующей команды:
zgrep -i -B 5 -A 5 "bacteriorhodopsin" UP000434101.swiss.gz
В выдаче я получила пять результатов, чтобы узнать больше информации по каждой последоваткльности и исключить перекрывание результатов, я увеличила количество строк в флагах, тогда количество результатов сократилось до 4.
Данный белок кодируется четырьмя разными генами, но у меня вызвало интерес скорее то, что у первой последовательности в поле DR(Database Cross-Reference) была следующая запись:
CDD; cd15243; 7tm_Halorhodopsin; 1.
Здесь CDD(Conserved Domain Database) - база данных, которая содержит аннотации доменов белков. Ссылка на эту базу данных есть в записи, а значит белковый домен гомологичен какому-то еще, в нашем случае гомолог это 7tm_Halorhodopsin. О галородопсине я писала в своем мини-обзоре, где рассматривала его структурную схожесть с бактериородопсионом.
cd15243 - уникальный ID домена в CDD, а цифра 1 означает что данный домен повторяется в белке 1 раз.
Так как ссылка на CDD имеется только в одной из четырех записей про бактериородопсин, можно сделать вывод, что ген(GS429_03200), кодирующий данный домен, невидоспецифичный и имеется у других представителей семейтсва, у кого синтезируется галородопсин.
Также интерес представляет то, что белки, кодируемые генами GS429_06865 и GS429_06875, A0A6B0VJS4_9EURY и A0A6B0VLZ7_9EURY соответсвенно, имеют идентичную структуру. Если также учесть схожесть имен этих генов, можно сказать, что предположительно данные гены локализованы очень близко друг к другу в ДНК.
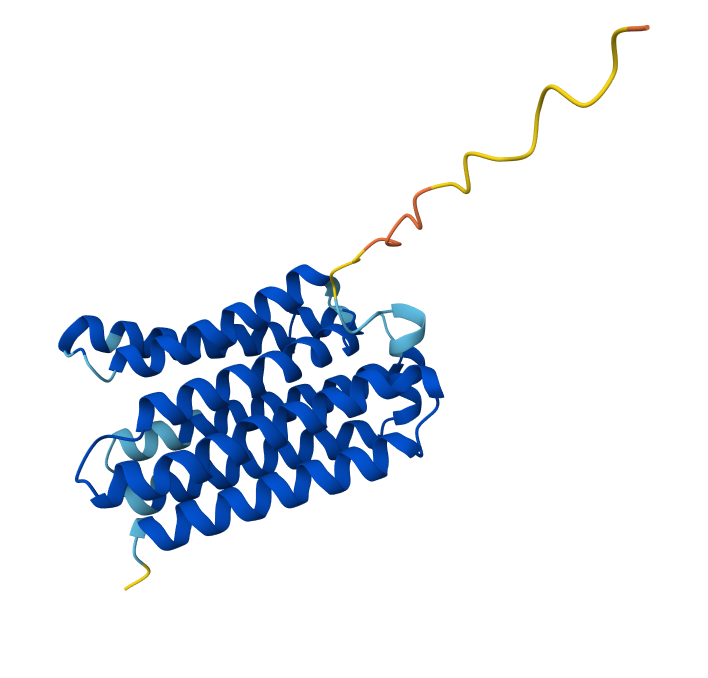
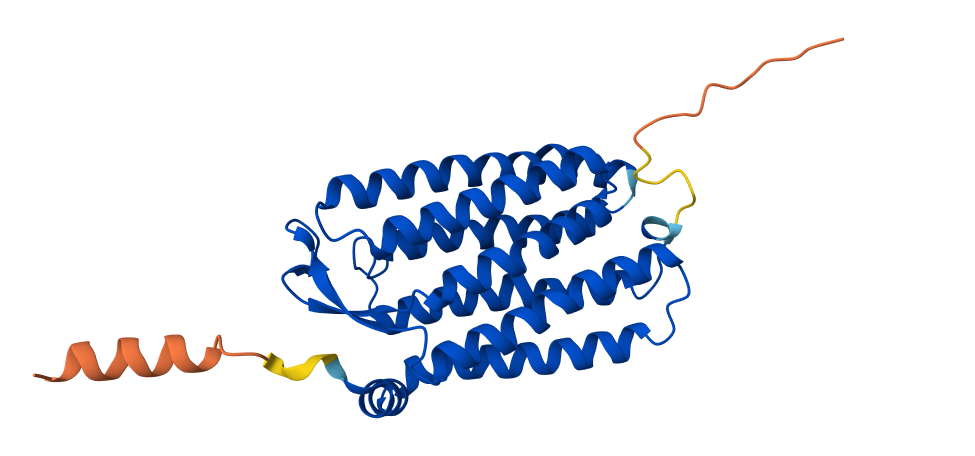
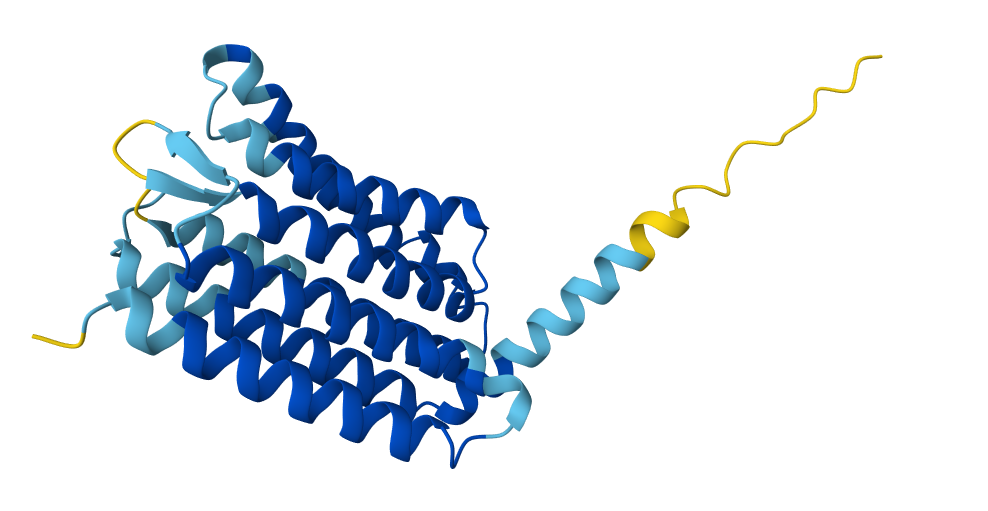
Иллюстрации слева направо:A0A6B0VM50, A0A6B0VHR4, A0A6B0VJS4, A0A6B0VLZ7.



Дополнительно: из поля CC(comments) я узнала, что бактериородопсин относится к семейтсву опсионовых белков, встречающихся как у архей, так и у бактерий и грибов(Belongs to the archaeal/bacterial/fungal opsin family).
Посмотрим, каждый ли белок протеома начинается с метионина:
zgrep -A 1 "^SQ" UP000434101.swiss.gz | grep -v "^SQ" | cut -c 1-6 | grep -c "M"
Оказывается, в исследуемом протеоме 4149 белков из 4155 начинаются с нужной аминокислоты, у оставшихся шести последовательность начинается с аланина, аспарагиновой кислоты, глутаминовой кислоты, серина, треонина и валина. Единственная связь между этими аминокислотами в том, что у них нет ароматического кольца.
zgrep -A 1 "^SQ" UP000434101.swiss.gz | grep -v "^SQ" | cut -c 1-6 | sort | uniq
Альтернативные старт-кодоны встречаются у некоторых архей. Это может быть связано с адаптацией к экстремальным условиям, так как цитозин и гуанин более устойчивы к высоким температурам, нежели аденин и тимин.