С помощью текстового поиска в файле с последовательностями белков гепарда был найден белок, аннотированный как δ-субъединица АТФ-синтазы. Последовательность этого белка:
>XP_026905295.2 ATP synthase subunit delta, mitochondrial [Acinonyx jubatus]
MLPVAVLRRPGLRCLVRQARAYAEAAAAPAPASGPGQMSFTFASPTQVFFNGANVRQVDVPTQTGA
FGILAAHVPTLQVLRPGLVVVHAEDGTTSKYFVSSGSVTVNADSSVQLLAEEAVTLDMLDVGAAKV
NLEKAQSELSGAADEASRAEIQIRIEANEALVKALEИдентификатор нуклеотидной записи, к которой относится ген, кодирующий данный белок: NC_069383.1. Последовательность ДНК, непосредственно содержащая кодирующую белок часть гена δ-субъединицы АТФ-синтазы:
>NC_069383.1:c165050974-165048698 ATP5F1D [organism=Acinonyx jubatus] [GeneID=106983723] [chromosome=A2]
TCGCCATGCTGCCCGTCGCGGTCCTGCGCCGTCCCGGCCTGCGCTGCCTCGTCCGTCAGGCCCGCG
CCTACGCGGAGGCTGCGGCTGCGCCGGCCCCGGCTTCGGGCCCGGGCCAGATGTCCTTCACCTTCG
CCTCACCCACGCAGGTTCGGATTCCGGTCGGGCTCGGGACCCCTCCTTGATCACCACCTCCAGTCG
AGATCCGCTGTGGTCCGCAATCGGAGACCTCCAACCTTTGGGTTGTCGACCCTCATTTATTCCGCT
CCCTGTGCCCCCTCCCCCGTGTGGTCCGAAATCCATCCCCAGGCCACCGCCTCCTGCTCCGGATCC
ATGTCCTCATTCCGCACCCCGGATCCGCGTCGGCGCCCCAATGTGGTCAGGACTCCCGGATCCCCA
GCCTCCGAGTCTCGGAGATCCCTAACTCCCCCTCCAGGACCCCGGACCGATGCCCCCTCCACCTGT
GTCCCTCCGAGAGCCACCCCCCAACCCCTGTGTCGTTGACCCGCGCCCTACGCCTTTCCAAGTGGT
GTGGAATCCGAGCCCCATAAAGCTTTCCGAGTTCCCGAGCCTTCCGCCTGGAATCCAACGCCCCAG
GGCTGGACCTCACCGTTCTGCCCGTCCCGCAGAACCCCGAGCCTCAGACACTTGTTGTCTTGTGTC
TTCCTGGGTCGGCGCGCTTCTCGTGCGTGCTCTTGGCAGCGTTTCTGTGGGGACGGGTTCCGTCTG
GATTTTATCTTCTGTGCACTAAGACGCTGCTAGAGAACCAAGACAAGTATGCGCGTCCTCGTGGCT
GCTTCCGTGTGTGCTCTGTCGCCGTGGGGTCTTGCCTTGGGCCGTTTCACTGCAGGGCAGACTGAG
ATCCGGGTGGGGAGGACTGGCCTGCAGGGATTCCCCGCGTGTTCTTCCAGGTATTTTTCAATGGTG
CCAACGTCCGGCAAGTGGACGTTCCCACGCAGACGGGAGCCTTTGGCATCCTGGCGGCCCACGTAC
CCACCTTGCAGGTGCTGCGGCCGGGGCTGGTTGTTGTCCACGCCGAGGACGGCACCACCTCCAAAT
ACTTTGGTGAGTGCACCGGGAAGGGGCTGGGCGGGGCCACGGGCTTCAGCAGGCCCAAGTGTCTCG
CTTCTCCGTGTGGATAGAAGCAGAGCCCACGAGAGAGACAAACCCGTGCCTCCAGTAGCTTGTGGT
GGGTCAGGGTCTCGGTGGGGGCCAGGCGTCTGAGAGGGAAGTCCAGTCCTGTCGCAGAGGAGCCGA
TGGAGAGGGCGGGGGGGGGGGGGTAACTGGATCCTAAGGAGAGCGTGCAGCAGCACAACTCTGAGG
TACAGCCAAGATGCAGACCTGGCGCCCCGCTCACCCCTGACCCTGGCTCAGCTGATGGGCTGGTGC
TGGTGGGTGAAGGAAGGAGGCCCTGTTCCCCGTGTGCAGGTGCCAGTGGGCGACCATGGGCCCTGT
GGGCATCAGCCTGTCTCCCATGCTAGACCGCTCTGGAGCCGCTTCTGTGGCTCTGGCTGACGGGGT
CCCCCGCACCCGAACACGGAACTCTCACGGCTGTCTTCTCCCGTAGTGAGTAGCGGCTCCGTCACA
GTGAACGCTGATTCCTCAGTGCAGTTGTTGGCTGAAGAGGCCGTGACCTTGGACATGCTGGACGTG
GGGGTGAGTGTTCCAGGGGAAGCCGATGGACCCAGGAGGCCACATCTGAGCCAGGGCACCAGGCGG
GGTCGCTGCTTGGCTCTCACCTGCTACCCACTTCCTGCAGGCCGCCAAGGTGAACTTGGAGAAGGC
GCAGTCGGAGCTGTCAGGGGCAGCAGACGAGGCCTCCAGGGCCGAGATCCAAATCCGCATCGAGGC
CAACGAGGCCCTGGTGAAAGCTCTCGAGTAGGCGGTCCGTGGCCCTTGCCAGCGGGGAAACCGAGG
CCCAGGACCGGACCGGGGATGTCCCGGGCAGGCTGAACCAGCTCGCGTGGGTCGTCTGATTGGGGG
TGGGGAAGTGGGGAGAGAGGGGCCGCGAGCCACCTGGGGGATTCTTGGGTGGAGGGAGCGGTCTTG
CCAAGAGGCCGCCAGGGGGCAGCACACTGTCAGGCCTCCTAGCGAAGGGGGGCTCGGCTGAGACCC
TGCTCTGCATCTCACCTTCCAGCCGGCTCCCCCCGCCCCAACCGGACGTGCCACCCATTTTCTCTC
TAACCTTGTAGACTCTGCTGTGGAGCCCTCAGCTCCTCGTCCTGCCCCCTGGGACAGCCCGCTCCCA
GCTTGAGCCCCCCATAAAAACCAGGGACCTGA
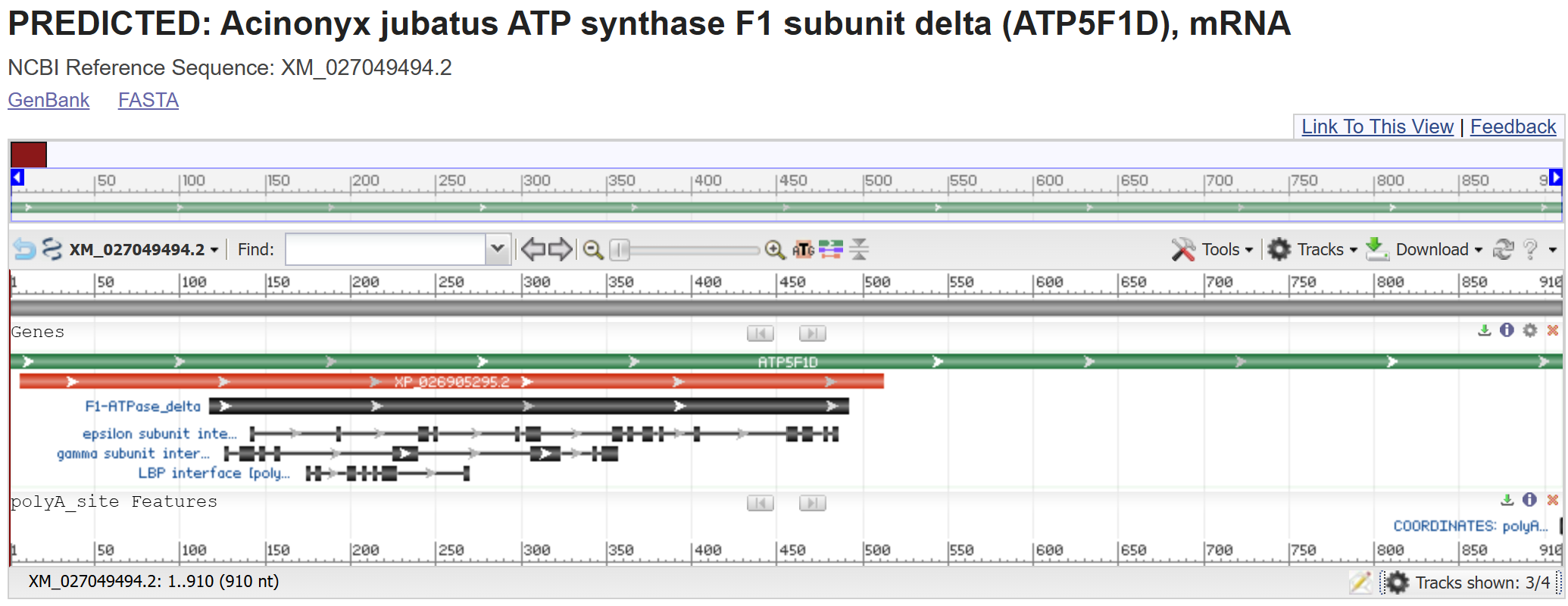
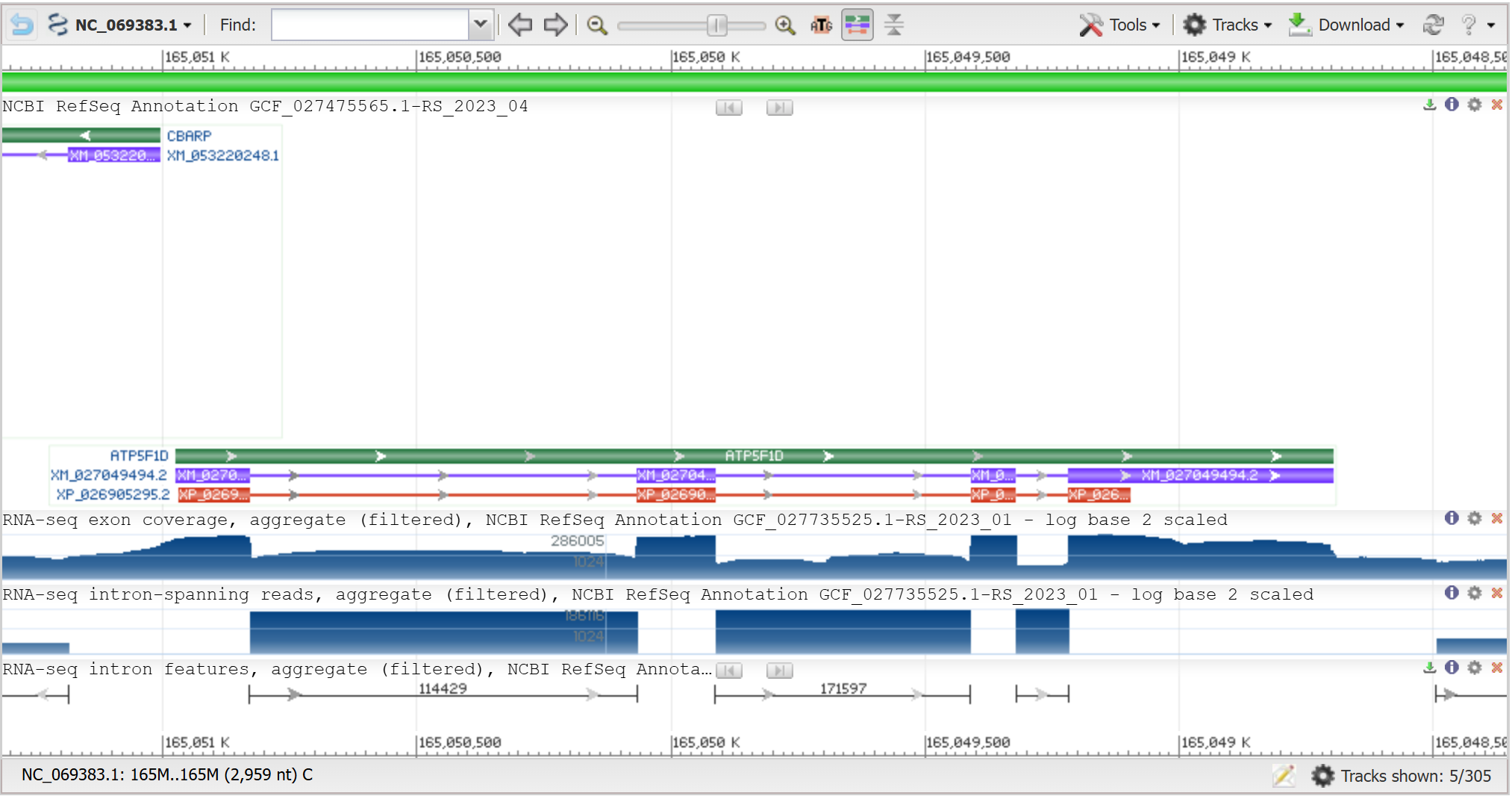
Рис. 1 и 2. Экспрессия участка гена, кодирующего δ-субъединицу АТФ-синтазы
Зеленым изображен ген, красным - мРНК, черным - белок (нижняя картинка). Координаты кодирующей белок части гена: [165,050,969..165,049,096].
Для оценки сходства изучаемого белка с гомологичными белками других организмов была проведена оценка по группе эукариотических организмов групп Araneae (Пауки) и Apoidea (Пчелы), поскольку АТФ-синтаза является эволюционно консервативным белком.
При поиске с помощью blastn никаких результатов получено не было, даже при уменьшении длины слова до 7.
С использованием tblastn при поиске среди доступных паучьих геномных сборок было получено четыре выравнивания. При поиске среди пчел (Apoidea), tblastn обнаружил 38 совпадений. По рисункам ниже можно сделать вывод, что пчелиные дельта субъединицы АТФ-синтазы по последовательности похожи на гепардову больше, чем паучьи. В обоих случаях была использована стандартная длина слова - 5.
Megablast не использовали, поскольку он нужен для поиска высоко-гомологичных последовательностей, а это не наш случай. Tblastx не был использован, поскольку он работает дольше tblastn (а во время выполнения практикума у меня blast и без того очень долго думал).
Выравнивание для пауков: Araneae.txt
Выравнивание для пчел: Apoidea.txt
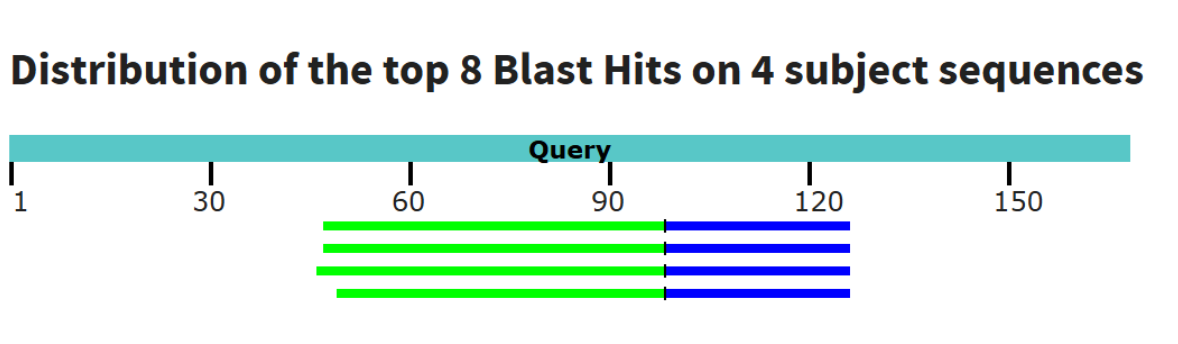
Рис 3. Выдача tblastn для пауков
Рис 4. Выдача tblastn для пчел

Проиндексируем последовательности генома нашего эукариота для работы локального BLAST, который предварительно скачали на наш компьютер:
makeblastdb -in "C:\Users\RedmiDownloads\GCF_027475565.1_VMU_Ajub_asm_v1.0_genomic.fna.gz" -dbtype nucl -out cheetah_dbДалее скачиваем последовательности 16S и 23S рРНК Escherichia coli. По полученной базе данных cheetah_db с помощью blastn проводим поиск для каждой рРНК.
Получаем выравнивание следующей командой:
blastn -task blastn -query "C:\Users\Redmi\Downloads\16S.fna.txt" -db cheetah_db -out 16_out -outfmt 7Среди 11 хитов, приемлемое значение e-value (меньше стандартного для blast 0.05) только у двух находок. Данные находки имеют в геноме следующие идентификаторы: NW_026464032.1, NC_069397.1.
Первая это скаффолд, вторая - хромосома E1. Таким образом для 16S рибосомы кишечной палочки найдено два гомолога в геноме Acinonyx jubatus.
Табличная выдача blastn для 16S рРНК кишечной палочки: 16_out
blastn -task blastn -query "C:\Users\Redmi\Downloads\23S.fna.txt" -db cheetah_db -out 23_out -outfmt 7Здесь уже из 22 хитов 4 значимые находки для NW_026464096.1 и NC_069397.1. Причем три из них выровнялись на скаффолд, по координатам последовательности и скаффолда можно понять, что эти находки составляют один гомолог, то есть всего у нас гомолога два.
Проиллюстрируем следующий участок выравнивания:
query NW_026464096.1 73.143 175 41 3 2442 2613 3492 3663 4.78e-17 96.9
query NW_026464096.1 75.581 86 21 0 1898 1983 2855 2940 3.44e-06 61.7
query NW_026464096.1 81.356 59 10 1 2233 2290 3282 3340 5.10e-04 54.5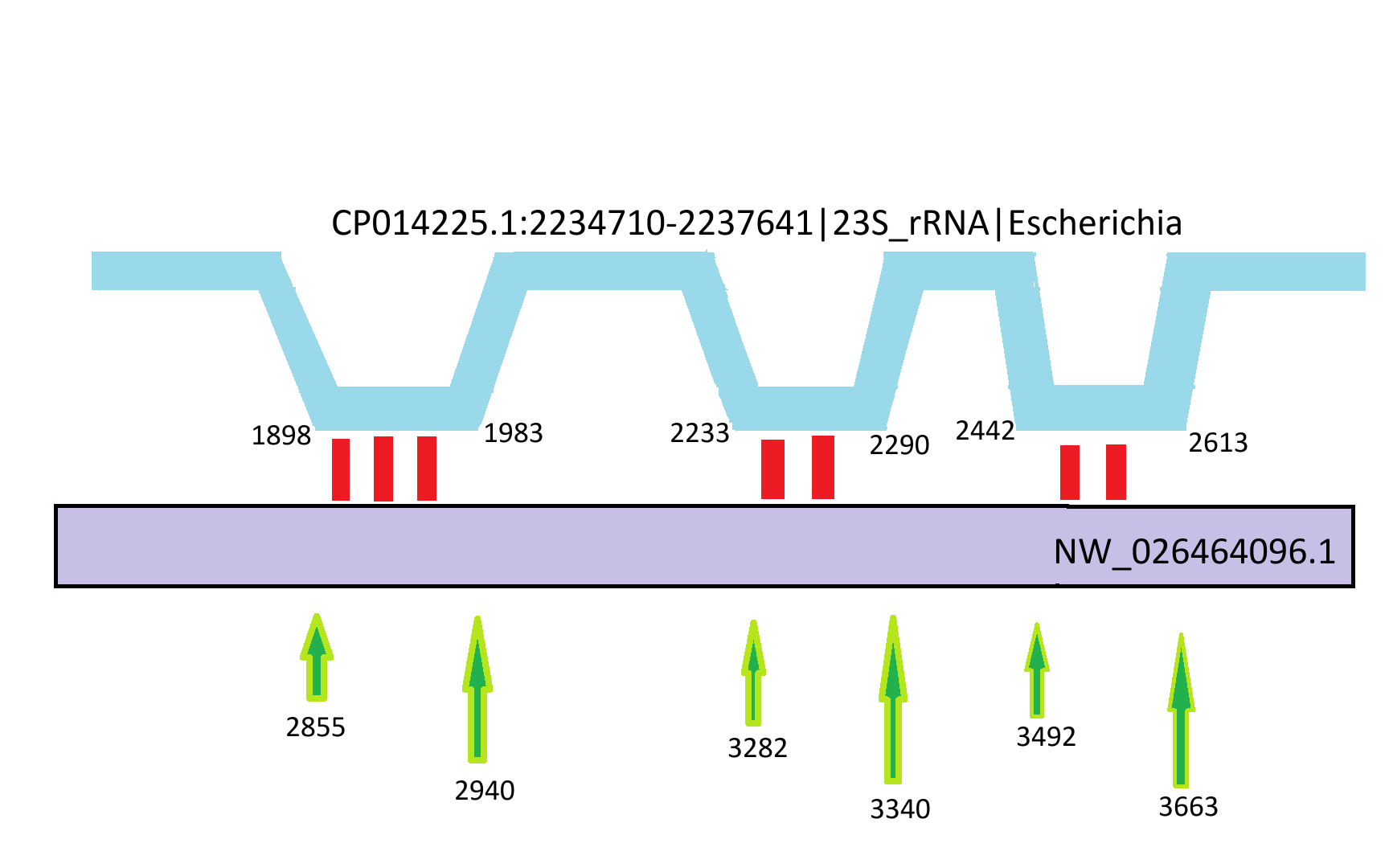
Рис 5. Локальный поиск BLAST для 23S рРНК
Табличная выдача blastn для 23S рРНК кишечной палочки: 23_out
В эукариотических организмах 16S и 23S гомологичны соответственно 18S и 28S рРНК. 18S(16S) отвечает за связывание мРНК, контроль кодон-антикодонового взаимодействия и обеспечение точности декодирования. 28S(23S) катализирует реакцию образования пептидной связи. В прокариотической инициации трансляции участвует последовательность Шайна-Дальгарно, которой нет у эукариот, а потому 16S и 23S рРНК малой субъединицы рибосомы отличаются значительнее, что проявляется в количестве хитов и e-value в выдаче blastn.
Построим карты локального сходства хромосом архей Natronorubrum bangense (NZ_CP031305.1) и Natronorubrum dagingense (NZ_CP019328.1) двумя программами: megablast, blastn. К сожалению, tblastx не может провести выравнивание, выдавая ошибку поиска.
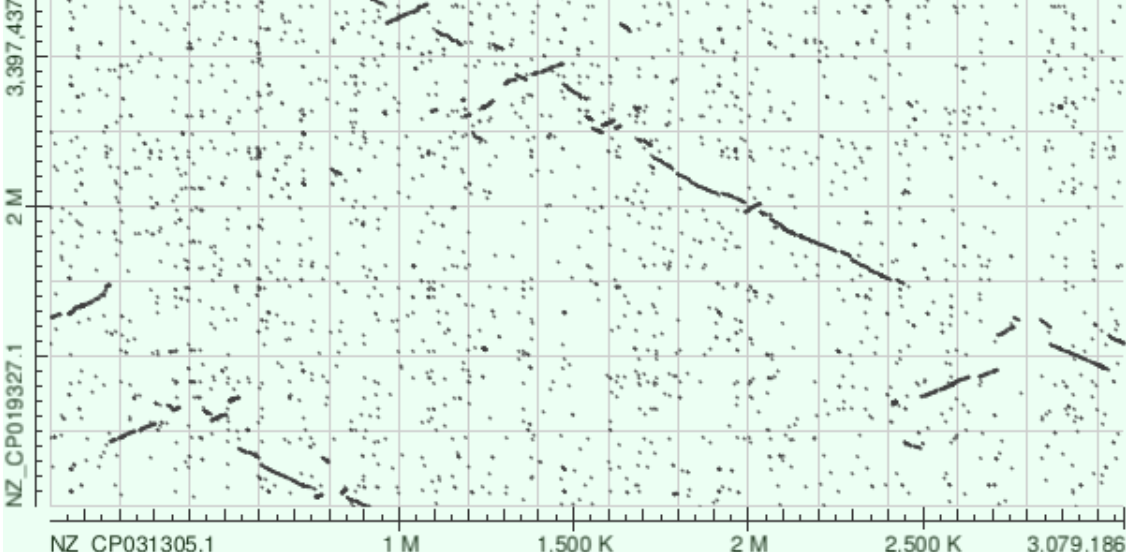
Рис 6. Карта blastn
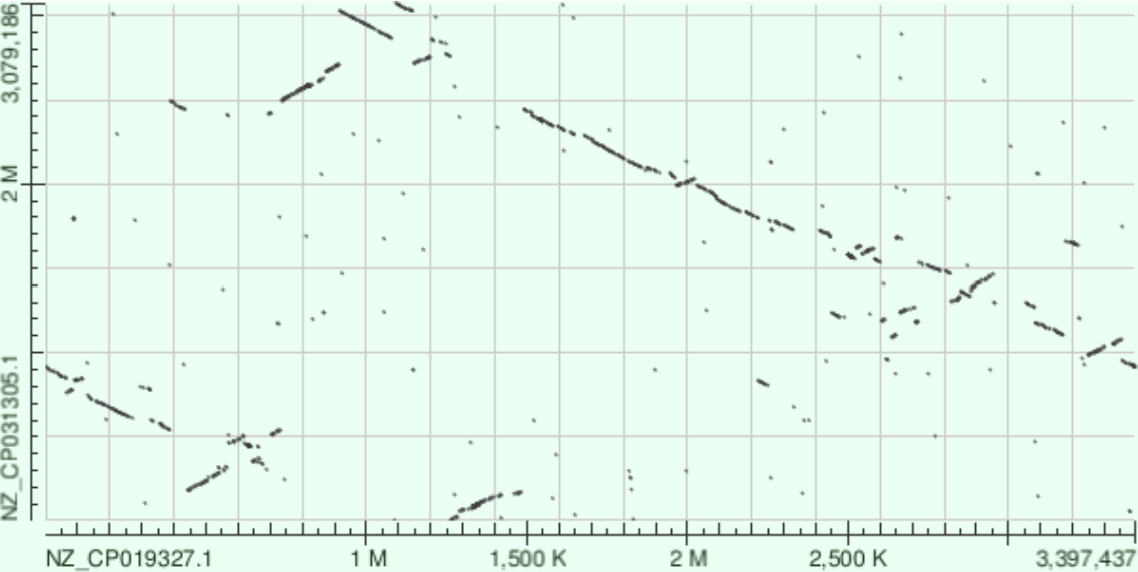
Рис 7. Карта megablast
По нижнему левому углу можно определить, что кольцевые геномы двух архей секвенировались, начиная с разных участков. Также из рисунков видно, что в геномах есть инверсии, инверсии с транслокациями, делеции и дупликации.