� Цель данного занятия ознакомится с возможностями
докинга низкомолекулярного лиганда в структуру белка
� В этом занятии будет использоваться пакет Autodock Vina и Autodock tools.
1. В банке pdb найдена SMILES нотацию для NAG. Эта нотация была сохранена в файл nag.smi.
2. C помощью obgen была построена 3D структура этого сахара в pdb формате.
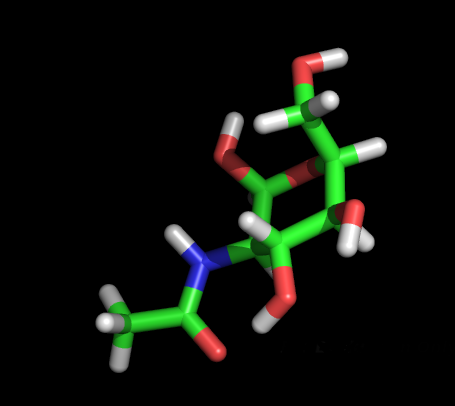
3. Скриптом prepare_ligand4.py из пакета Autodock tools создан nag.pdbqt
� файл вашего лиганда.
4. Так же, скриптом prepare_receptor4.py из пакета Autodock tools создайте
pdbqt
файл вашего белка.
5. Итак у нас есть входные файлы. Теперь надо создать файл с параметрами докинга vina.cfg.
Необходимо указать область структуры белка, в которой будет происходить поиск
� места для связывания. Удобно его задать как куб с неким центором. Координаты центра мы определим из модели
� комплекса, которую мы построили на прошлом занятии. Выберем атом сахара, который по нашему мнению
находится в центре сайта связывания и из текста pdb файла извлечем его координаты.
Постройте файл vina.cfg.
6. Проводим первый докинг:
vina --config vina.cfg --receptor model5.pdbqt --ligand nag.pdbqt --out nag_prot.pdbqt --log nag_prot.log
� Получены файлы nag_prot.pdbqt и nag_prot.log.
7. Проанализируем файл nag_prot.log. Найдём энергии 3ёх лучших расположений и геометрическую разницу между ними:
| Номер положения | энергия (ккал/моль) | геометрическая разница (u.b.rmsd) |
| 1 | -5.8 | 0.000 |
| 2 | -5.8 | 2.212 |
| 3 | -5.8 | 3.078 |
� Изобразим все состояния лиганда в активном центре
�
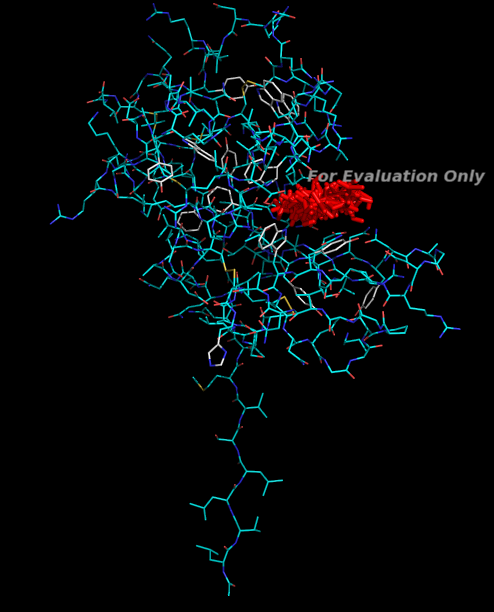
� 8. Теперь давайте проведём докинг рассматривая подвижность некоторых боковых радикалов белка. Сначала разобьем белок на две части, подвижную и неподвижную. � Для подвижной части выберем 3 аминокислоты которые vы использовали в прошлом задании для � позиционирования лиганда: ASP120, TRP82, ASN77
prepare_flexreceptor4.py -r model5.pdbqt -s ASP120_TRP82_ASN77
� Проведем докинг
vina --config vina.cfg --receptor model5_rigid.pdbqt --flex model5_flex.pdbqt --ligand nag.pdbqt --out vina_model5_flex.pdbqt --log vina_model5_flex.log
9. Рассмотрим файл vina_model5_flex.log и занесём в таблицу энергии 3ёх лучших расположений и геометрическую разницу между ними:
| Номер положения | энергия (ккал/моль) | геометрическая разница (u.b.rmsd) |
| 1 | -5.6 | 0.000 |
| 2 | -5.5 | 1.518 |
| 3 | -5.5 | 1.292 |
| 10 | -4.6 | 1.579 |
�
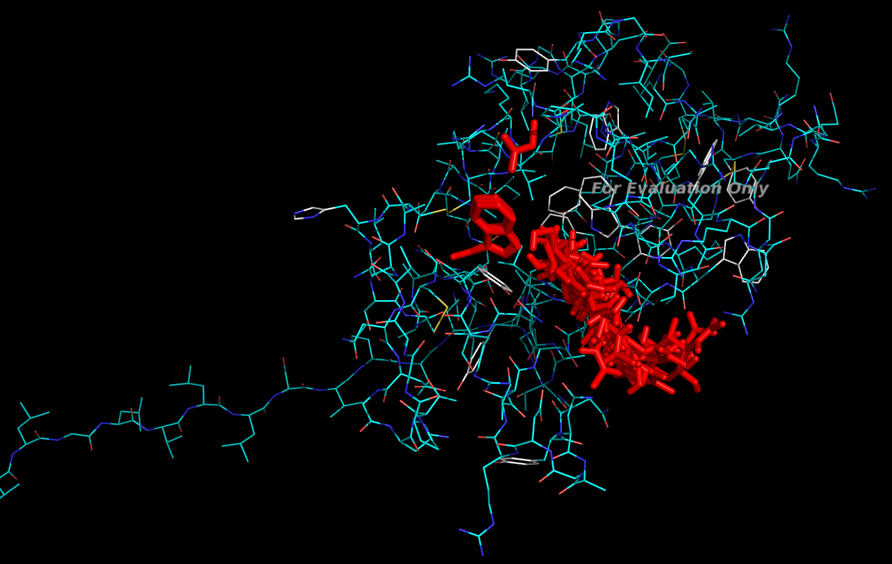
Биологический докинг учитывает изменения положений АК в белке при связывании с лигандом. � В данном случае лиганд более подвижен. Такой докинг лучше отражает действительность, так как белки в нормальных условиях подвижны.