ОБЗОР ГЕНОМА И ПРОТЕОМА БАКТЕРИИ MICROBULBIFER THERMOTOLERANS
Вадим Стрекаловских
Факультет биоинженерии и биоинформатики, Московский государственный университет им. М.В. Ломоносова, г. Москва, Российская Федерация.
Резюме:
Данная работа представляет собой обзор бактерии Microbulbifer thermotolerans и в частности её генома и протеома, которые могут иметь промышленную ценность с точки зрения производства белков, полезных для хозяйственной деятельности человека.
Ключевые слова: геном; протеом; Microbulbifer.
Microbulbifer thermotolerans – вид гамма-протеобактерий, обнаруживаемых в иловых наносах океанического дна в Восточной Азии. Род Microbulbifer и его типовой вид M. hydrolyticus были описаны в 1997 году (Gonzalez et al., 1997), после чего было открыто ещё множество новых видов этого рода. Многие из них обладают схожими физиологическими и биохимическими особенностями; например некоторые виды способны усваивать целлюлозу, хитин, агар-агар, переживать перепады температур или расти на сильно-солёных средах (Shu-Kun Tang et al., 2008). Из-за этих уникальных особенностей представители рода Microbulbifer могут представлять практический интерес для промышленных предприятий, использующих биотехнологические методы производства и переработки химических соединений (Moh et al., 2017; Lee YS, Choi YL, 2016). Для вида M. thermotolerans также показана возможность разложения простых органических соединений и нефти (Lee et al., 2014). Основным биохимическим преимуществом этой бактерии является наличие так называемых белков холодового шока (CSP, англ. cold-shock proteins; CAE, англ. cold-adapted enzymes), которые способны поддерживать жизнедеятельность клетки при низких температурах. Эти белки характеризуются высокой ферментативной активностью и устойчивостью к охлаждению и нагреванию. Таким образом, бактерии вида Microbulbifer thermotolerans обладают большим комплексом белков, которые могут оказаться полезными в биотехнологическом производстве. В данной статье производится обзор таких белков и кодирующих их последовательностей с целью поиска и выявления закономерностей распределения их по геному.
Ссылка на исходные материалы: https://ftp.ncbi.nlm.nih.gov/genomes/all/GCF/001/617/625/GCF_001617625.1_ASM161762v1.
Материалы, использованные в работе, включают в себя таблицу особенностей, нуклеотидные последовательности генома и транскриптома и аминокислотную последовательность протеома M. thermotolerans, полученные по вышеуказанному веб-адресу. Также мною использовалась таблица белок-кодирующих последовательностей, полученная путём редактирования таблицы особенностей генома в сервисе Google Sheets с применением функции создания фильтра. Фильтрация осуществлялась по элементу CDS в столбце #feature и по элементу with_protein в столбце class. Таким образом, лист «CDS» представляет собой таблицу кодирующих последовательностей (CDS, англ. coding sequence), транслируемых в белки.Программа GC-Content написана на языке Python. Она обрабатывает нуклеотидную последовательность в формате FASTA и выводит процентное содержание нуклеотидов G и C. Также программа работает со списком аминокислотных последовательностей, определяя содержание аминокислот с высоким (пролин, аланин, глицин) и низким (аспарагин, лизин, изолейцин) содержанием GC в соответствующих кодонах.Программа GC-in-RNA написана на языке Python и обрабатывает нуклеотидную последовательность структурного транскриптома (совокупности некодирующих РНК). Программа выводит процентное содержание нуклеотидов G и C.Программа GC-skew принимает на ввод нуклеотидную последовательность генома и выводит локальные (local) и общие (cumulative) значения GC-skew. По умолчанию, размер окна – 100000 нуклеотидов, размер шага – 1000. Результатом выполнения программы является соответствующая таблица со значениями и двумя графиками.Для изучения процесса рибосомного скольжения (англ. ribosomal slippage) в протеоме исследуемого организма использовались таблица рибосомных скольжений и программа Ribosomal Slippages. Таблица рибосомных скольжений представляет собой фрагмент таблицы CDS с последовательностями, отсортированными по параметру «ribosomal_slippage» в столбце attributes. Программа Ribosomal Slippages работает со списком аминокислотных последовательностей и выводит те из них, которые соответствуют изучаемым белкам, а также попарные различия в них.Таблицы статистических данных включают в себя распределение длин белков, полученное посредством применения функции COUNTIFS (лист «Histogram»), и некоторые статистические величины – средняя длина, стандартное отклонение, медиана, минимум, максимум (лист «Descriptive statistics»).
Воспользовавшись программами GC-Content и GC-in-RNA, я получил следующие результаты:
Нетрудно заметить, что гуанин и цитозин достаточно слабо преобладают над аденином и тимином. Аналогично, аминокислот с высоким содержанием G и C в соответствующих кодонах (пролин, аланин, глицин) немного больше, чем аминокислот с низким их содержанием (аспарагин, лизин, изолейцин).
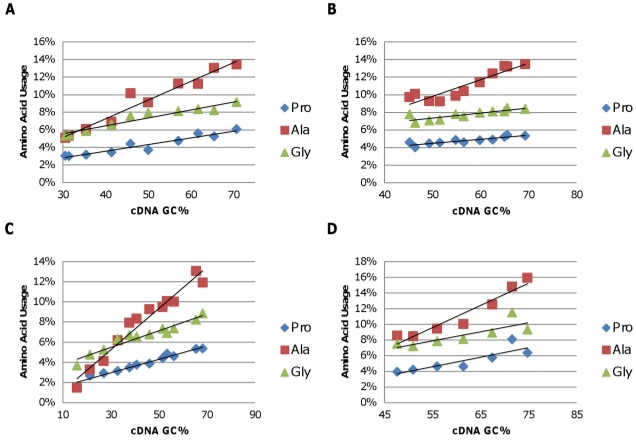
Если сравнить полученные данные с аналогичными показателями для других видов гамма-протеобактерий и других классов бактерий, то можно заметить, что Microbulbifer thermotolerans хорошо вписывается в общую тенденцию своего класса, в отличие от неродственных групп прокариот. На графиках ниже представлены данные для классов: A) Alphaproteobacteria; B) Betaproteobacteria; C) Gammaproteobacteria; D) Deltaproteobacteria (Lightfield et al., 2011).

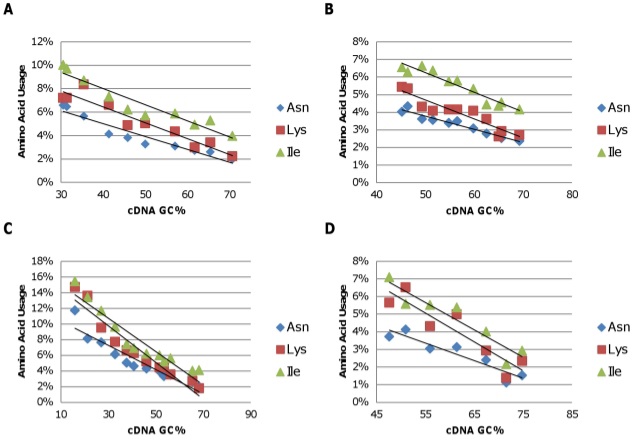
Известно, что бактерии вида M. thermotolerans характеризуются устойчивостью к повышению температуры окружающей среды. В геноме живого организма комплементарные (соединённые друг с другом) пары азотистых оснований гуанина и цитозина поддерживаются тремя водородными связями, тогда как в паре аденин-тимин водородных связей только две. По этой причине двухцепочечная молекула ДНК, обогащённая гуанином и цитозином, является более термостабильной (Yakovchuk et al., 2006). В связи с этим ранее предполагалось, что высокое содержание GC в геноме обеспечивает адаптацию организма к высоким температурам, однако более поздние исследования показали, что GC-состав и устойчивость к нагреванию не коррелируют между собой (Hurst LD, Merchant AR., 2001). В примере с M. thermotolerans гуанин и цитозин преобладают недостаточно значительно, чтобы оказывать влияние на способность бактерии переживать перепады температур.
Согласно тому же недавнему исследованию (Hurst LD, Merchant AR., 2001), термоустойчивость прокариотных организмов зависит от GC-состава структурных РНК – рибосомных, транспортных и др. В таблице особенностей генома содержатся сведения о 61 последовательности структурных РНК, в том числе 48 транспортных и 9 рибосомных. Применение программы GC-in-RNA к файлу с нуклеотидными последовательностями РНК показало, что содержание GC-оснований в транскриптоме составляет 56,00% – меньше, чем в геноме. Таким образом, рассматриваемый вид является скорее исключением из правила; по всей видимости, обеспечение выживаемости бактерии в условиях перепада температур полностью берут на себя белки теплового и холодового шока, для которых показано сохранение каталитической активности в широком диапазоне температур (Wistow, 1990).


Графики выше построены с применением программы GC-skew.py. Данные, использованные при построении графиков, находятся в таблице «GC-skew Microbulbifer» среди сопроводительных материалов.
GC-skew – это показатель, определяющий разницу между содержанием гуаниновых и цитозиновых нуклеотидов на отдельных участках цепи. Он вычисляется по формуле (G – C)/(G + C), где G и C – количества соответствующих нуклеотидов в участке цепи. Известно, что две цепочки ДНК, образующиеся во время репликации генома, имеют разные соотношения гуанина и цитозина. Гуанин преобладает на лидирующей цепи (удлиняющейся от 3'-конца), цитозин – на запаздывающей цепи (которая наращивается от 5'-конца). Соответственно, для лидирующей цепи характерны положительные значения GC-skew, а для запаздывающей – отрицательные. Было также замечено, что минимальному значению общего GC-skew соответствует точка начала репликации (oriC), а максимальному – точка завершения (ter). Анализ таблицы значений GC-skew позволяет установить, что oriC находится на нулевой позиции в последовательности, а ter располагается на 1869000 позиции, с погрешностью в 1000 нуклеотидов.
|
ID |
# feature |
start |
end |
strand |
name |
product_length |
attributes |
|
110 |
CDS |
123 693 |
124 822 |
+ |
IS3 family transposase |
376 |
ribosomal_slippage |
|
880 |
CDS |
1 067 420 |
1 068 549 |
- |
IS3 family transposase |
376 |
ribosomal_slippage |
|
1050 |
CDS |
1 275 064 |
1 276 193 |
- |
IS3 family transposase |
376 |
ribosomal_slippage |
|
1244 |
CDS |
1 495 195 |
1 496 290 |
+ |
peptide chain release factor 2 |
364 |
ribosomal_slippage |
|
1935 |
CDS |
2 306 590 |
2 307 719 |
+ |
IS3 family transposase |
376 |
ribosomal_slippage |
|
2308 |
CDS |
2 817 010 |
2 818 139 |
- |
IS3 family transposase |
376 |
ribosomal_slippage |
|
2655 |
CDS |
3 244 680 |
3 245 809 |
+ |
IS3 family transposase |
376 |
ribosomal_slippage |
|
2689 |
CDS |
3 275 000 |
3 276 129 |
+ |
IS3 family transposase |
376 |
ribosomal_slippage |
|
2875 |
CDS |
3 515 080 |
3 516 209 |
- |
IS3 family transposase |
376 |
ribosomal_slippage |
|
2941 |
CDS |
3 600 530 |
3 601 659 |
+ |
IS3 family transposase |
376 |
ribosomal_slippage |
Табл. 1. Последовательности с рибосомным скольжением.
Анализ таблицы особенностей генома позволяет выделить 10 специфических последовательностей, которые подвержены так называемому «рибосомному скольжению» – изменению открытой рамки считывания в процессе трансляции. 9 из 10 соответствующих белков имеют одинаковую длину и принадлежат к одному семейству протеинов IS3, однако располагаются на разных цепях на удалении друг от друга. Этот факт наводит на мысль о том, что их CDS представляют собой мобильные генетические элементы, способные дублироваться и перемещаться по геному. Т.к. все 9 белков являются траспозазами – ферментами транспозиции, процесса перемещения генов, – то можно сделать вывод, что они катализируют собственное размножение в пределах генома. Последовательности, кодирующие такие белки, называются инсерционными (Alberts et al., 2002). Вероятно, эти гены бактерия получила от вирусов-бактериофагов посредством горизонтального переноса и, развив устойчивость к заражению, интегрировала их в свой геном.
При помощи пограммы Ribosomal Slippages.py мне удалось установить попарные различия в аминокислотных последовательностях девяти транспозаз:
110: T 880: A
1050: E 1935: D
1935: D 2308: E
110: S 880: G
Таким образом, белок с ID 110 отличается от других тем, что в его последовательности на 56 месте находится не аланин, а треонин, а на 346 месте – серин вместо глицина. У белка с ID 1935 на 287 позиции располагается аспарагиновая кислота, а у всех других белков – глутаминовая. Такие малочисленные расхождения могут свидетельствовать либо об эволюционно недавнем появлении транспозонов у вида Microbulbifer thermotolerans, либо о высокой консервативности генов, что неудивительно по причине их малой длины и слабой экспрессии.
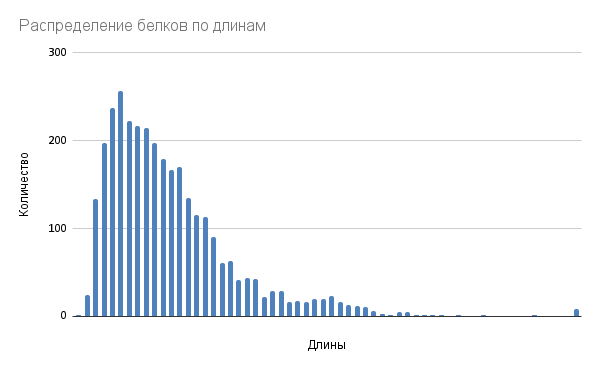
Для данной гистограммы размер кармана составляет 30 единиц. Как можно заметить, среди всех белков преобладает длина 151-181 а.о. (аминокислотных остатков). Также присутствуют несколько локальных малозаметных пиков, среди которых выделяется группа протеинов длиной около 901-931 а.о. Возможно, этот пик включает в себя крупные функционально специфические белки (например, структурные или транспортные).
В данной работе был произведён обзор некоторых аспектов генома, транскриптома и протеома бактерии Microbulbifer thermotolerans, представляющих интерес с точки зрения биоинженерии. Показано, что данный вид бактерий может быть успешно использован в исследовательских и производственных целях, т.к. он способен обитать в условиях, близких к экстремальным, имеет компактный геном, включающий ряд транспозонов, и сравнительно небольшой протеом (3220 единиц), в составе которого присутствуют физиологически значимые белки теплового и холодового шока. Также выявлены закономерности в распределении белков по длинам.
1. Таблица особенностей генома:
GCF_001617625.1_ASM161762v1_feature_table.txt.gz
https://docs.google.com/spreadsheets/d/1RZOx4y5HT7rF1LsQxqRroIGL1NK2NVc_QAbH1H9EJ6I/edit?usp=sharing
2. Нуклеотидная последовательность генома:
GCF_001617625.1_ASM161762v1_genomic.fna.gz
https://drive.google.com/file/d/1ahz_JBtGg7Gc1C5St02zvktR7cEaV-C9/view?usp=sharing
3. Нуклеотидная последовательность транскриптома:https://drive.google.com/file/d/1h_Qbpehk2eplt9rCNcWaMyL7Ugrc7Rso/view?usp=sharing
4. Аминокислотные последовательности:
https://drive.google.com/file/d/14kh6C7WgzO48RvUzcKyfU8K1PfozwCfl/view?usp=sharing
5. Таблица белок-кодирующих CDS:
https://docs.google.com/spreadsheets/d/1ACo5r3u9IN2ORavQJ4SfE19Wj-UyJrSjmnX-LXR49EI/edit?usp=sharing
6. Программа для определения GC-состава генома:
https://drive.google.com/file/d/1fDthzq3XjykwyKKb4WyJBnx2qvYvh4ho/view?usp=sharing
7. Программа для определения GC-состава транскриптома:
https://drive.google.com/file/d/1ltFo6Q0pHXTWK1nl5HdEVMsdCyus2vFy/view?usp=sharing
8. Программа для определения GC-skew генома:
https://drive.google.com/file/d/1xW7MbjiYr8W2iV-4UqilFtHAgtsv0Th2/view?usp=sharing
9. Таблица со значениями GC-skew и два графика:
https://docs.google.com/spreadsheets/d/1eWUUEpytmNqw9owz7N1FJcTC_0Al7t8iJAFErh55QH8/edit?usp=sharing
10. Таблицы статистических данных протеома и гистограмма длин белков:
https://docs.google.com/spreadsheets/d/1Vd4xNOo8-EixDyDk783E7yTw1c4DIsVKv2wXPkHeS38/edit?usp=sharing
11. Таблица рибосомных скольжений:
https://docs.google.com/spreadsheets/d/1hfc7c6F361oDU0fjrYIsnBHKfL5FNbJBqH8ce1bxKTc/edit?usp=sharing
12. Программа для определения различий в белках IS3:
https://drive.google.com/file/d/1uxysgnrBFVLwFzY-4YZRVeJQpFzWHKFp/view?usp=sharing