Мини-обзор генома и протеома археи Natronomonas pharaonis
Гришин Вадим Дмитриевич
Факультет биоинженерии и биоинформатики, Московский государственный университет имени М. В. Ломоносова, Ленинские горы д. 1 стр. 73, 119234, Москва, Россия
e-mail: vg_scientist@mail.ru
Аннотация — Данный обзор посвящён анализу генома и протеома галоалкалифильной археи Natronomonas pharaonis. В статье используются различные методы исследования белков, а также генов и их нуклеотидного состава с помощью формул электронных таблиц (MIN, MAX, COUNTIF и др.) и сценариев командной строки Bash. В ходе исследования были получены результаты по распределению длин белков протеома, частотам встречаемости старт-кодонов в геноме, распределению числа белок-кодирующих генов и генов разных типов РНК, GC%-составу генома, распределению длин межгенных промежутков и межгенных перекрываний в геноме, а также распределение по числу различных аминокислот и соответствующих им архейных тРНК. В конце был сделан вывод о закономерностях, обнаруженных в геноме и протеоме археи, с точки зрения эволюционной адаптации к условиям обитания данного вида организмов.
Ключевые слова: геном, протеом, Natronomonas pharaonis, электронные таблицы, командная строка Bash
ВВЕДЕНИЕ
| Домен | Archaea |
| Царство | Euryarcheota |
| Класс | Halobacteria |
| Отряд | Halobacteriales |
| Семейство | Halobacteriaceae |
| Род | Natronomonas |
| Вид | Natronomonas pharaonis |
Natronomonas pharaonis — это аэробные галоалкалифильные археи (см. рис. 1), интересные с точки зрения азотистого обмена. Они являются экстремофилами — живут при очень высоких значениях pH (около 11) и при очень высоких концентрациях солей в карбонатных озёрах. Название виду дал штамм Габара (DSM2160), найденный впервые в озере Габара в Египте. Все представители данного вида содержат одну кольцевую хромосому и две кольцевые плазмиды (PL23 и PL131, имеющих размеры 23 и 131 килобаза соответственно). У Natronomonas pharaonis было отмечено повышенное содержание кислых аминокислот (аспартата и глутамата) в составе протеома (около 19,3%). Это расценивается как адаптация к условиям гипертонической и щелочной среды, в которой обитают эти археи. Стоит также упомянуть, что данный вид не обладает генами, синтезирующими ферменты гликолиза, а потому эти археи не способны к расщеплению сахаров [1]. Археи весьма интересны с точки зрения анализа генома, поскольку имеют много общих черт как с представителями бактерий, так и с представителями эукариотических организмов. Анализ частот встречаемости старт-кодонов в нормальных генах и псевдогенах, длин пересечений генов и межгенных промежутков, а также исследование иных характеристик генома, вероятно, позволят получить новые важные сведение о данном организме и найдут непосредственное приложение в современной науке.
МАТЕРИАЛЫ И МЕТОДЫ
1. Получение данных. Данные о нуклеотидных последовательностях белок-кодирующих генов, нуклеотидном составе всего генома и данные о локальных особенностях генома изучаемой археи были получены из банка геномов [4] с сайта NCBI. Названия файлов, использовавшиеся при исследовании генома и протеома, следующие: GCF_000026045.1_ASM2604v1_cds_from_genomic.fna; GCF_000026045.1_ASM2604v1_feature_table.txt; GCF_000026045.1_ASM2604v1_genomic.fna.
2. Для подсчёта длин белков, закодированных в геноме археи, в командной строке Bash была использована команда infoseq *_cds_from_genomic.fna -nocolumns -out cds.csv, генерирующая таблицу свойств последовательностей, хранящихся в файле с CDS, в формате .csv. Далее эта таблица была импортирована в соответствующий лист книги Google Sheets, и на основе её данных была построена гистограмма, отображающая среднее значение длин белков, закодированных в геноме. Использовались такие методы электронных таблиц, как COUNTIFS, MAX, MIN.
3. Для анализа частот встречаемости старт-кодонов в геноме изучаемой археи я использовал такие методы электронных таблиц (Google sheets), как “VLOOKUP” и “IFERROR”, а также методы командной строки Bash (см. S8).
4. Для подсчёта числа генов белков и генов разных типов РНК в каждом репликоне использовался метод электронных таблиц “COUNTIFS”. Путём анализа таблицы с данными было обнаружено, что необходимо провести отбор значений по ключевым словам в соответствующих столбцах, чтобы подсчитать число генов белков и РНК. Для этого я воспользовался функцией “фильтр”, которая позволила мне отобрать только удовлетворяющие условиям задачи значения, после чего построил таблицу, в ячейках которой были подсчитаны количества генов белков и генов различных видов РНК (5S рРНК, 16S рРНК, 23S рРНК, тРНК) отдельно для хромосомы (содержащей один единственный репликон) и каждой из двух плазмид.
5. Анализ GC%-состава генома проводился с помощью методов электронных таблиц, а в частности, MIN, MAX, COUNTIFS.
6. Исследование длин межгенных промежутков осуществлялось при помощи построения гистограммы, а также с помощью различных методов электронных таблиц, таких как MIN, MAX, COUNTIFS.
7. Для исследование длин пересечений генов в той же книге, в которой проводилось построение гистограммы длин межгенных промежутков, в новом листе была построена гистограмма длин пересечений генов на основе данных из листа и таблицы локальных особенностей генома при помощи методов электронных таблиц MIN, MAX, COUNTIFS.
8. Изучение соответствия различного числа архейных тРНК определённым аминокислотам проводилось с помощью метода электронных таблиц COUNTIF.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Длины белков, закодированных в геноме археи Natronomonas pharaonis. Гистограмма была построена с помощью некоторых методов электронных таблиц, описанных в разделе “Материалы и методы” (см. рис. 2). По оси абсцисс отложены диапазоны длин (в аминокислотах), в которые могут попадать белки исследуемой археи; по оси ординат отложено количество значений длин белков, попадающих в определенные диапазоны.
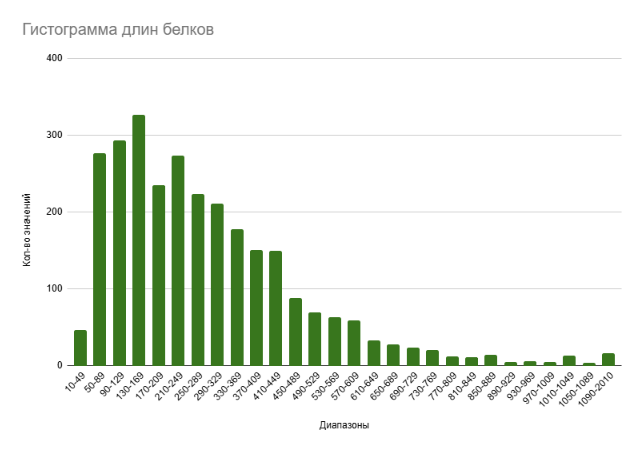
По гистограмме видно, что большая часть белков этой археи имеют размеры от 50 до 450 аминокислотных остатков. Превалирующие значение относительно небольших белков в протеоме археи позволяет сделать вывод о том, что у данного вида нет необходимости в синтезе больших белков, состоящих из нескольких доменов и выполняющих сложные функции, и им достаточно использование лишь небольших белков с относительно простыми функциями (например, регуляторной или сигнальной) и простой пространственной структурой. Также подавляющее количество маленьких белковых молекул говорит об оперонной организации генов археи с целью компактизации генома [5].
Частоты встречаемости старт-кодонов в геноме. Таблица со старт-кодонами содержит информацию о частоте встречаемости того или иного старт-кодона в нормальных генах и псевдогенах (см. таблицу 1).
| Кодоны | Все CDS | Псевдо CDS | Нормальные CDS |
|---|---|---|---|
| ATG | 2529 | 9 | 2520 |
| GTG | 257 | 2 | 255 |
| TTG | 14 | 0 | 14 |
| ATA | 6 | 0 | 6 |
| ATC | 6 | 2 | 4 |
| CTG | 4 | 1 | 3 |
| CGC | 3 | 3 | 0 |
| ATT | 2 | 1 | 1 |
| Другие | 13 | 13 | 0 |
Из данных таблицы нетрудно заметить, что для всех генов, как для нормальных, так и для псевдогенов, наиболее часто встречаемым старт-кодоном оказался кодон ATG (2529 во всех генах). Также из данных таблицы видно, что наиболее часто встречаемыми старт-кодонами, помимо упомянутого выше, являются старт-кодоны GTG (257), TTG (14), CTG (4). Вероятно, повышенная частота встречаемости данных старт-кодонов связана с тем, что в ходе накопления мутаций геномом археи в некоторых генах заменился первый нуклеотид наиболее распространённого старт-кодона ATG, однако изменённый старт-кодон, находящийся в начале белок-кодирующей последовательности, продолжил выполнять свою функцию, благодаря частичной комплементарности антикодона тРНК CAU [6].
Число генов белков и генов разных типов РНК в геноме. Таблица была построена с помощью описанных в разделе “Материалы и методы” методов электронных таблиц (см. таблицу 2).
| Репликон | Белок-кодирующие гены | 5s рРНК | 16s рРНК | 23s рРНК | тРНК |
|---|---|---|---|---|---|
| Chromosome | 2651 | 1 | 1 | 1 | 46 |
| plasmid PL23 | 37 | 0 | 0 | 0 | 0 |
| plasmid PL131 | 115 | 0 | 0 | 0 | 0 |
Из данных таблицы видно, что большая кольцевая хромосома содержит подавляющее большинство генов, кодирующих белки (2651, ~95% об общего числа генов.), и гены всех типов РНК (по одному для каждого вида рРНК, 46 генов для тРНК). Также нетрудно заметить, что плазмиды содержат только белок-кодирующие гены, что логично, ибо плазмиды содержат, как правило, лишь небольшой набор генов, повышающих приспособленность организмов к условиям окружающей среды [7], в то время как основная генетическая информация, в частности, о различных видах РНК содержится в нуклеоиде (большой кольцевой хромосоме).
GC%-состав генома. На основе данных таблицы со свойствами последовательностей генов, кодирующих белки, была построена гистограмма, отображающая распределение процентного содержания нуклеотидов G и C в геноме (см. рис. 3). По оси абсцисс отложены диапазоны значений GC-состава в процентах, по оси ординат — количество значений, которым соответствует данное процентное содержание гуанина и цитозина.
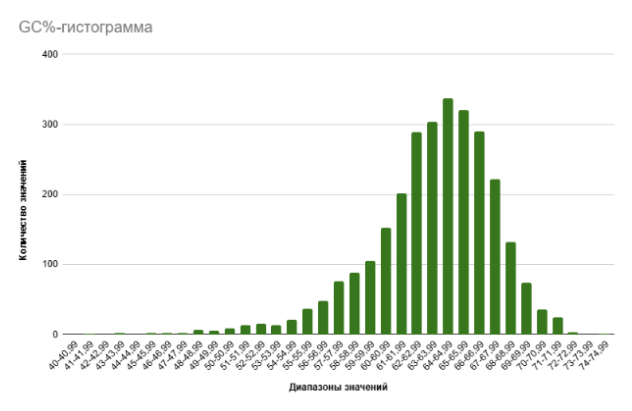
Гистограмма строилась стандартными методами электронных таблиц, описанными в разделе “Материалы и методы”. По данной гистограмме нетрудно увидеть, что наиболее часто встречающиеся значения нуклеотидного состава G и С лежат в диапазоне от 62 до 67 процентов, что в среднем составляет около 63,4% для хромосомной ДНК. Также заметен небольшой пик в районе 50-54%, который, по всей видимости, возникает из-за того, что такое процентное содержание нуклеотидов G и C чаще всего встречается в CDS плазмид. Вероятно, такое высокое содержание гуанина и цитозина в геноме связано с экстремальными условиями среды обитания Natronomonas pharaonis (повышенное содержание данных нуклеотидов увеличивает “прочность” и “устойчивость” генома за счёт образования между комплементарными основаниями тройной водородной связи).
Длины межгенных промежутков в геноме. На основе данных таблицы о локальных особенностях генома исследуемой археи была построена гистограмма длин межгенных промежутков на +-цепи ДНК (см. рис. 4).
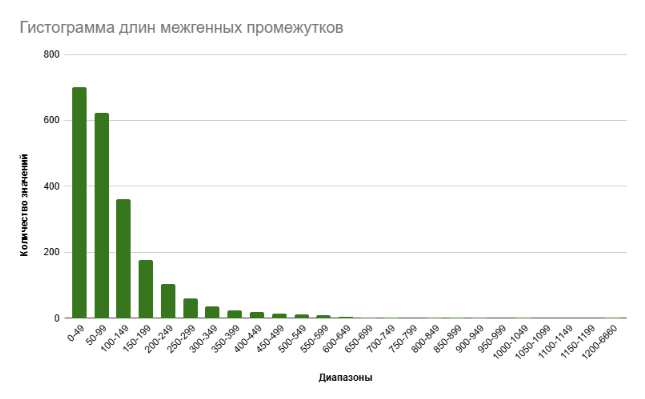
Проанализировав данную гистограмму, можно сделать вывод о том, что большая часть межгенных интервалов не превышает значений в 50-100 нуклеотидов. Это свидетельствует о весьма близком взаимном расположении генов по сравнению с эукариотическим организмами, у которых большая часть генома представлена некодирующими генами и интронами, что позволяет предположить наличие у данной археи полицистронных оперонов — групп генов, находящихся под одним общим промотором и синтезирующих несколько белковых продуктов, регулирующих общий метаболический процесс.
Длины межгенных пересечений в геноме. На основе релевантных данных из таблицы локальных особенностей генома археи была построена гистограмма длин перекрываний между генами на +-цепи ДНК (см. рис. 5).
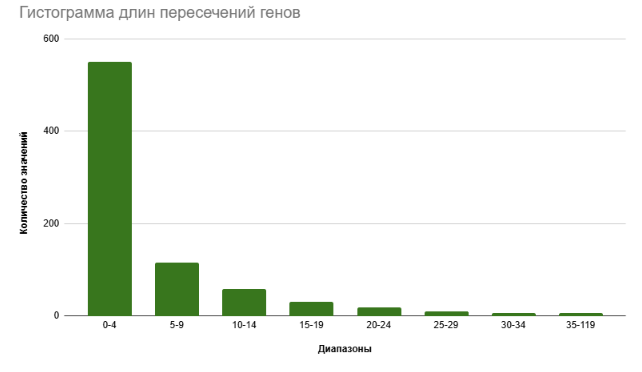
Как видно из самой гистограммы, подавляющее большинство генов имеют перекрывания не более 4 нуклеотидов, что также свидетельствует о наличии оперонов в геноме археи.
Число различных протеиногенных аминокислот и соответствующих им тРНК. Было проанализировано, сколько видов тРНК соответствуют каждой аминокислоте в протеоме исследуемой археи (см. таблицу 3).
| Аминокислота | Число тРНК | Аминокислота | Число тРНК |
|---|---|---|---|
| Ala | 3 | Lys | 2 |
| Arg | 4 | Met | 2 |
| Asp | 1 | Phe | 1 |
| Asn | 1 | Pro | 4 |
| Cys | 1 | Ser | 4 |
| Gln | 2 | Val | 3 |
| Glu | 2 | Thr | 3 |
| Gly | 3 | Trp | 1 |
| His | 1 | Tyr | 1 |
| Ile | 2 | Sec | 0 |
| Leu | 5 | Pyr | 0 |
Всего в геноме археи закодировано 46 различных типов тРНК, что немного меньше, чем у эукариот (около 50). Это связано с тем, что по многим характеристикам генома археи весьма схожи с эукариотами, и потому их генетической код не должен сильно отличаться. Также можно сделать вывод о том, что для архей характерен так называемый “вобблинг” (англ. wobbling) — явление, объясняющие наличие меньшего числа различных видов тРНК по сравнению с количество триплетов, кодирующих протеиногенные аминокислоты (всего триплетов за исключением стоп-кодонов 61) [8]. Из данных таблицы видно, что аминокислоте лейцин у Natronomonas pharaonis соответствует целых 5 различных тРНК (у эукариот всего 4), а метионину — 2 (у эукариот 1) [9]. Нестандартных протеиногенных аминокислот, таких как селеноцистеин или пирролизин, этот вид не содержит.
ЗАКЛЮЧЕНИЕ
Полученные данные позволяют сделать вывод о том, что свойства генома архей схожи с таковыми как у бактерий (наличие плазмид, кодирующих небольшое число генов, повышающих приспособленность организма к условиям окружающей среды; наличие разных видов старт-кодов в белок-кодирующих генах и псевдогенах; оперонная организация генома; маленькие расстояния между CDS; синтез небольших белков с простой пространственной структурой), так и у эукариот (синтез белка начинается с именно с метионина, а не с формилметионина, как у бактерий). Также конкретно для данного вида было выявлено повышенное содержание нуклеотидов гуанина и цитозина в геноме, что, вероятно, связано с экстремальными условиями окружающей среды (pH = 11 и высокое содержание солей), в которой обитают эти археи.
БЛАГОДАРНОСТИ
Автор выражает благодарность всему преподавательскому составу курса биоинформатики на ФББ МГУ за доступное изложение необходимого для написания данного мини-обзора материала, а также за обучение методам работы с реальными биоинформатическими данными.
СОПРОВОДИТЕЛЬНЫЕ МАТЕРИАЛЫ
Таблица S1. Гистограмма длин белков.
Protein length hist
Таблица S2. Частоты старт-кодонов отдельно для нормальных генов и псевдогенов.
Start_codons
Таблица S3. Подсчёт числа генов белков и генов разных типов РНК в геноме археи.
Per_replicons
Таблица S4. Гистограмма GC%-состава белок-кодирующих генов в геноме археи.
В гугл-таблице подробно указаны все необходимые значения (шаг, карманы, минимальное значение и др.), для построения гистограммы.
GC_hist
Таблица S5. Гистограмма длин межгенных промежутков.
Intervals Hist
Таблица S6. Гистограмма длин межгенных перекрываний.
Intersections Hist
Таблица S7. Подсчёт количества разных видов тРНК для каждой аминокислоты.
Counts_tRNA
S8. Сценарий командной строки Bash для подсчёта старт-кодонов в геноме археи.
Start_codones_Bash.pdf
ЛИТЕРАТУРА
1. Michaela Falb, Peter Palm, Friedhelm Pfeiffer, Karin Rodewald, Volker Hickmann, Jörg Tittor, and Dieter Oesterhelt (2005). "Living with two extremes: Conclusions from the genome sequence of Natronomonas pharaonis". http://www.genome.org/cgi/doi/10.1101/gr.3952905
2. Oren A; Ventosa A (2000). "International Committee on Systematic Bacteriology Subcommittee on the taxonomy of Halobacteriaceae. Minutes of the meetings, 16 August 1999, Sydney, Australia". Int. J. Syst. Evol. Microbiol. 50 (3): 1405–1407. https://doi.org/10.1099%2F00207713-50-3-1405 PMID 10843089.
3. en:Image:Halobacteria.jpg. NASA: Cluster of cells of Halobacterium sp. strain NRC-1. Taken from http://science.nasa.gov/headlines/y2004/10sep_radmicrobe.html
4. Геном археи из банка геномов на сайте NCBI: Index of /genomes/all/GCF/000/026/045/GCF_000026045.1_ASM2604v1.
5. Kyrpides, NC; Ouzounis, CA (20 July 1999). "Transcription in archaea". Proceedings of the National Academy of Sciences of the United States of America. 96 (15): 8545–50. Bibcode:1999PNAS...96.8545K. doi:10.1073/pnas.96.15.8545. PMC 17553. PMID 10411912.
6. Lobanov, A. V.; Turanov, A. A.; Hatfield, D. L.; Gladyshev, V. N. Dual functions of codons in the genetic code (англ.) // Critical Reviews in Biochemistry and Molecular Biology : journal. — 2010. — Vol. 45, no. 4. — P. 257—265. — https://dx.doi.org/10.3109%2F10409231003786094. — PMID 20446809. — PMC 3311535.
7. Hayes F. The function and organization of plasmids. (англ.) // Methods In Molecular Biology (Clifton, N.J.). — 2003. — Vol. 235. — P. 1—17. — https://dx.doi.org/10.1385%2F1-59259-409-3%3A1. — PMID 12904641.
8. Crick, F.H.C. (August 1966). "Codon—anticodon pairing: The wobble hypothesis" (PDF). Journal of Molecular Biology. 19 (2): 548–555. CiteSeerX 10.1.1.693.2333. doi:10.1016/S0022-2836(66)80022-0. PMID 5969078.
9. "Amino Acid Translation Table" .Oregon State University.