Семейства белковых доменов. База данных белковых доменов Pfam.
В предыдущем практикуме, посвященном множественному эволюционному выравниванию, для выполнения задания было выбрано семейство белковых доменов под названием tRNA synthetases class II (D, K and N)
с AC PF00152. Аминоацил-тРНК синтетазы (они же АРСазы) катализируют реакции присоединения остатка протеиногенной аминокислоты к молекуле тРНК в процессе трансляции. Эти ферменты делятся на два класса
(I и II), принципиально различающиеся по некоторым физико-химическим характеристикам, таким как, например, более предпочтительный сайт для присоединения аминоацильного остатка к сахару (для ферментов,
относящихся к первому классу, более предпочтительно присоединение по 2'-гидрокисилу сахара, для ферментов второго класса — по 3'-гидроксильной группе).
Среди особенностей доменных архитектур, в которых представлен рассматриваемый домен, можно выделить присутствие в подавляющем большинстве наиболее широко представленных архитектур
тРНК-антикодонового домена (OB-fold nucleic acid binding domain), который, очевидно, будет присутствовать на пару с тРНК-синтетазным доменом в ферментах, осуществляющих присоединение аминокислоты к тРНК.
Касательно 3D-структуры, можно подчеркнуть, что для белков, содержащих данный домен, характерен α/β-тип третичной укладки (центральные бета-листы окружены альфа-спиралями).
Таксономическая представленность белков, содержащих описываемый домен, в значительной степени преобладает в супергруппе Бактерий и составляет более 75% от всех последовательностей. Представленность
среди Эукариот и Архей составляет примерно 20% и 4%, соответственно. Краткие характеристики с пояснениями к ним приведены в Таблице 1.
| Характеристика | Информация | Комментарии |
|---|---|---|
| AC PFAM | PF00152 | Код доступа (Accession code) выбранного семейства белковых доменов |
| ID PFAM | tRNA synthetases class II (D, K and N) | Полное имя (=ID, Name) выбранного семейства белковых доменов |
| #SEED | 83 | Число последовательностей в множественном выравнивании seed |
| #All | 159k | Общее число белковых, содержащих домен выбранного семейства |
| #SW | 2k | Число белков, содержащих домен выбранного семейства, и содержащийся в базе данных Swiss-Prot |
| #architectures | 746 | Количество разных доменных архитектур, содержащих домен из рассматриваемого семейства |
| #3D | 126 | Количество известных пространственных структур домена из рассматриваемого семейства |
| Taxonomy | 158182 | Суммарная таксономическая представленность домена в различных супергруппах организмов (указано число последовательностей) |
| #eukaryota | 30909 | Таксономическая представленность домена в супергруппе Эукариот |
| #archaea | 6594 | Таксономическая представленность домена в супергруппе Архей |
| #bacteria | 118680 | Таксономическая представленность домена в супергруппе Бактерий |
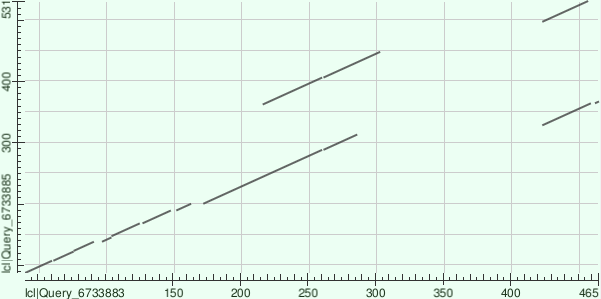
Далее, с помощью программы BLASTp была построена карта локального сходства (DotPlot) последовательностаей двух репрезентативных белков их разных доменных архитектур. Для построения карты целеноправленно была выбрана архитектура, представленная в подавляющем большинстве белков (AC репрезентативного белка P15178, количество белков 78142), и архитектура, представленная в очень небольшом числе белков (AC репрезентативного белка Q4TEL0, количество белков 64) для проверки наличия корреляции между представленностью доменной архитектуры и числом и типом мутаций, отображаемых на карте локального сходства. Ниже приведены Таблица 2, в которой описаны основные характеристики доменных архитектур и сама карта локального сходства последовательностей репрезентативных белков (см. рис. 2).
| Название | Информация | Комментарии |
|---|---|---|
| Доменная архитектура 1 | PF01336 - PF00152 (tRNA_anti-codon - tRNA-synt_2) | Последовательность AC доменов, входящих в состав доменной архитектуры. В скобках — названия соответствующих доменов |
| Белок с архитектурой 1 | P15178 (Aspartate-tRNA ligase) | AC и название репрезентативного белка для данной доменной архитектуры |
| Доменная архитектура 2 | PF20917 - PF01336 - PF00152 - PF00152 (AsnRS_N - tRNA_anti-codon - tRNA-synt_2 - tRNA-synt_2) | Аналогично комментарию к доменной архитектуре 1 |
| Белок с архитектурой 2 | Q4TEL0 (Asparagine-tRNA ligase) | Аналогично комментарию к белку с архитектурой 1 |
Для проверки достоверного различия доменов, входящих в состав разных доменных архитектур, были выбраны две доменные архитектуры с AC репрезентативного белка A0A0B3ARG6 и A0A814DDK0, представленные 15 и 24 белками, соответственно.
Координаты достоверных блоков одной архитектуры, не расширающиеся на вторую, в множественном выравнивании двух доменных архитектур блоков следующие: 1-74, 76-114, 116-195, 209-234, 240-243, 267-274, 339-344, 364-373, 378-455, 459-471 колонки.
Исходя из полученных в ходе множественного выравнивания двух доменных архитектур данных, а именно из-за присутствия в выравнивании доменной архитектуры с репрезентативным белком лизил-тРНК-лигаза (AC A0A814DDK0) большого количества достоверных блоков выравнивания, можно предположить, что домены, входящие в состав двух данных доменных архитектур, достоверно различаются, следовательно, эти доменные архитектуры образовались из уже разошедшихся в эволюции доменов. Ссылка на проект в Jalview.